Apoptosis
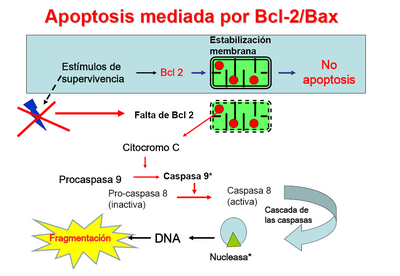
Bcl-2 es una familia de proteínas formada por alrededor de 25 miembros que regulan procesos de permeabilización mitocondrial y constituyen un punto clave en la vía intrínseca de apoptosis celular.
Su nombre deriva de la proteína fundadora, el protooncogén Bcl-2 (B-cell lymphoma 2), segundo miembro de un grupo de proteínas inicialmente descrito en estudios de la translocación recíproca entre los cromosomas 14 y 18 observada en linfomas foliculares. A diferencia de otros oncogenes estudiados hasta la fecha, Bcl-2 no estaba implicado en el control de la proliferación celular sino en el bloqueo de la muerte celular, más específicamente de los procesos de contracción citoplasmática, condensación nuclear, desorganización de lamembrana plasmática y ruptura endonucleolítica del ADN. A su vez, por su localización mitocondrial, fue un primer indicador de la importancia de este orgánulo en los procesos de apoptosis. Siguientes investigaciones demostraron la existencia de dos subfamilias homólogas pro-apoptóticas, Bax y BH3. Bax fue el primer homólogo identificado por su interacción con Bcl-2: ratones deficientes en Bax presentaban una expansión selectiva de poblaciones celulares y la relación Bcl-2/Bax se descubrió clave en la susceptibilidad a apoptosis por la vía intrínseca.
- Subfamilia Bcl-2 (anti-apoptótica): Bcl-2, Bcl-XL, Bcl-W, MCL-1, BCL2A1,BCL-B
- Subfamilia Bax (pro-apoptótica): Bax, Bak, Bok
- Subfamilia BH3 (pro-apoptótica): Bad, Bid, Bik, Blk, BimL, PUMA, NOXA,BMF, HRK
Estas subfamilias vienen definidas en parte por la homología con cuatro regiones denominadas dominios de homología Bcl-2 (BH). La subfamilia anti-apoptótica presenta una conservación de los 4 dominios y estudios con Bcl-Wpermitieron determinar que los dominios BH1, BH2 y BH3 eran los responsables de formar una región hidrofóbica de interacción con los dominios BH3 de las pro-apoptóticas, produciendo su inhibición. Los miembros de la subfamilia Bax presentan dominios BH1-3 por lo que también se les conoce como subfamilia “multidominio”, en contraposición con la subfamilia BH3 que sólo presenta homología en el mínimo dominio de muerte celular, la hélice anfipática BH3.
Células deficientes en BAX y BAK se han observado como resistentes a todos los estímulos conocidos de la vía de muerte celular intrínseca por lo que se les considera un requisito indispensable para que la vía resulte operativa. En células viables, BAX y BAK aparecen como monómeros, la primera anclada laxamente en la cara citosólica de diversos orgánulos y la segunda en el interior de la mitocondria. Ante una señal de muerte celular BAX se inserta en la membrana externa mitocondrial en forma de homooligómero y BAK sufre a su vez un cambio conformacional que incluye su oligomerización y la permeabilización de la membrana externa mitocondrial con la liberación de factores del espacio intermembrana como el citocromo c. El mecanismo exacto de esta liberación se desconoce aunque se plantean dos teorías principales. La primera postularía que las formas oligoméricas de BAX y BAK crearían poros capaces de liberar el citocromo c basándose en su similitud con toxinas bacterianas formadoras de poros y a la evidencia de experimentos in vitro. Una segunda teoría afirmaría que BAX y BAK interaccionarían con moléculas intrínsecas de membrana y producirían una transición de permeabilidad. Estos mismos procesos tendrían lugar en el retículo endoplásmico mediando la liberación de Ca2+.
Los miembros de la subfamilia BH3 funcionarían como iniciadores de la vía que integrarían de forma selectiva las diversas respuestas específicas de muerte y supervivencia. Por ejemplo, la caspasa-8 activada por señales de receptores de muerte celular en superficie origina una serie de modificaciones de la proteína BID que servirá como ligando para favorecer la oligomerización de Bax y Bak y, por lo tanto, su activación. La actividad de esta subfamilia está regulada a nivel transcripcional y postraduccional como, por ejemplo, por factores como p53 en respuesta a daño genético o por fosforilación en respuestas a factores de crecimiento. Actualmente se estudia si todas las moléculas de la familia BH3 tendrían funciones idénticas o podrían llegar a ser incluso excluyentes entre sí.
La familia Bcl-2, aparte de en el linfoma folicular de células B, está implicada en un gran número de cánceres, incluyendo el melanoma y los carcinomas de pecho, próstata y pulmón. También se cree relacionado con la resistencia a los tratamientos antitumorales convencionales.
Las proteínas bcl-2 (por sus siglas en inglés para gen de la linfoma de célula B) son asociadas con lasmembranas y su actividad. La proteína bcl-2 es una parte de un sistema complejo de señalamiento que controla la apoptosis. La apoptosis (muerte celular) puede ser inducida por una variedad de señales incluyendo daño irreparable del ADN. Esta forma de suicidio celular previene la expansión de las células dañadas. Bcl-2 sirve para prevenir la apoptosis. Entonces, su sobreexpresión puede prevenir la apoptosis en células que están dañadas. Esto puede llevar a una división contínua de las líneas de células mutadas y eventualmente al cáncer. También la sobreexpresión de bcl-2 puede contribuir a la metástasis en ciertos cánceres.
Entonces, su sobreexpresión puede prevenir la apoptosis en células que están dañadas. Esto puede llevar a una división contínua de las líneas de células mutadas y eventualmente al cáncer. También la sobreexpresión de bcl-2 puede contribuir a la metástasis en ciertos cánceres.
Si los controles de apoptosis son disrumpidos, entonces los medicamentos que trabajan para inducir la apoptosis ya no sirven tan efectivamente. Desde luego, medicamentos están siendo desarrollados para disminuir el bcl-2 y permitir que otros agentes anticancerosos funcionen más efectivamente (y en dosis más bajas). Uno de tales agentes es el nucleótido antisentido "Genasense", lo cual ha mostrado en las pruebas de fase I que reduce la producción de bcl-2 y está actualmente en las pruebas de fases II y III como un tratamiento suplemental para varios tipos de cáncer. Más sobre fármacos antisentido.
Más sobre fármacos antisentido.
También existen otros medicamentos que indirectamente reducen la cantidad de la proteína Bcl-2 tales como el ácido todo trans retinoico, paclitaxel, vincristina, y docetaxel. Estos medicamentos son a menudo combinados con otros agentes quimoterapeúticos durante el tratamiento. Métodos nuevos que todavía no han sido probados en humanos incluyen las péptidos que se unen a Bcl-2 para inactivar la proteína y la antamicina A que se une a las proteínas relacionadas al Bcl-2.
Ya que la sobreexpresión de Bcl-2 puede afectar el éxito del tratamiento del cáncer, sabiendo si éste está funcionando correctamente puede ser una herramienta de diagnóstico muy importante. Esteprotooncogen se vuelve activado en un oncogen por la translocación que causa la sobreexpresión de este gen, y una cantidad aumentada de la proteína Bcl-2 se ha descubierto en varios tipos de cáncer distintos.
Las proteínas c-FLIP (inhibidor celular proteico de FLICE,1 cellular FLICE inhibitory protein) son inhibidores celulares capaces de detener las señales de los receptores de muerte, que provocan la apoptosiscelular. Llevan tiempo siendo estudiadas por su posible importancia en futuros tratamientos deenfermedades degenerativas y están presentes sobre todo en el tejido muscular, el linfoide, en células delsistema inmunitario y en el corazón.
Estructura
La proteína FLIP fue por primera vez descrita como el producto de un gen viral (v-FLIP). Dichas proteínas virales contienen dos DED2 (dominio efector de muerte celular, death effector domain), por lo que pertenecen a la misma familia de proteínas que el FADD3 o las caspasas 8 y 10, que también poseen DED.
Al tratar de encontrar un homólogo celular de estas proteínas, se detectaron secuencias en ADNchumano con una elevada similitud. Se distinguen así las FLIP celulares (c-FLIP) de las que diferenciamos dos tipos: unas de cadena larga, las c-FLIPL, y una de cadena corta, las c-FLIPS.
Las proteínas c-FLIP están codificadas por cuatro ARN: uno corresponde a las FLIPS, ya que no se detecta en las pruebas de caspasa, y los otros tres, menos expresados en el cuerpo, son de las FLIPL.
La forma corta de la proteína (26 kDa), compuesta por 221 aminoácidos, tiene dos dominios de muerte celular y está estructuralmente relacionada con los inhibidores de apoptosis FLIP virales (v-FLIP).
La forma larga (55 kDa), de 480 aminoácidos, tiene un dominio similar a la caspasa pero cuyocentro activo tiene en lugar de cisteína una tirosina, anulando su función normal. Estructuralmente es muy parecida a las FLICE o caspasa 8. Su gen se encuentra en la región q33-q34 del cromosoma 2, igual que los de las caspasas 8 y 10. Esto lleva a la conclusión de que cFLIP puede provenir de la duplicación del gen de la caspsa 8, de ahí todas sus similitudes estructurales.
Nivel atómico
Se ha conseguido determinar la estructura del v-FLIP (MC159) a escala atómica, mediante cristalografía de rayos X. Al tener una estructura prácticamente idéntica en sus dominios de muerte (DED) a los DEDs de las c-FLIP, los resultados de esta investigación son extrapolables a las proteínas celulares.
La estructura del DED de la proteína v-FLIP (y, por tanto, de las c-FLIP) no sigue el mismo canon que los demás dominios de muerte celular. El DED1 del MC159 carece de la hélice 3 (propia de efectores de muerte celular como el FADD o el Fas), y en su lugar hay una estructura de bucle corto. Los DED se unen fuertemente entre ellos mediante interacciones hidrofóbicas: la cara 2/5 de la hélice del DED1 se une a la cara 1/4 de la hélice del DED2. Esto es completamente diferente a lo ocurrido intermolecularmente cuando, por ejemplo, la Apaf-1 (Apoptosis protease-activating factor-1) del apoptosoma, activa la procaspasa 9 (que dará lugar a la caspasa 9 y, después, a la caspasa 3). Las interacciones entre estas dos moléculas son principalmente electrostáticas y su punto de unión se encuentra en secciones diferentes de la hélice.
Todos los DEDs conocidos conservan una parte hidrofóbica fundamental para la interacción entre moléculas. En el MC159, dicha parte está enterrada en la interfaz en el DED1 y expuesta en el DED2. Los residuos hidrófobos DED-DED se conservan en otro complejo DED que contienen proteínas como las procaspasas 8 y 10 y la c-FLIP. En este aspecto el MC159 tiene un "empaquetado" de DEDs homólogo al de estas proteínas.
Al inicio de la hélice 6 de las proteínas que contienen DED se encuentra una secuencia nombrada RxDL, que interactúa con dos residuos de ácidos; un aspartato y unglutamato de la hélice 2. Dichos residuos tienen el papel estructural de sostener la hélice 2 y 6 y de estabilizar el pliegue DED.
La estructura en el nivel atómico de estos dominios de muerte celular es lo que confiere a proteínas como c-FLIP o FLICE las funciones que desempeñan en las células una vez sintetizadas.4
Cadenas de aminoácidos
La proteína c-FLIPL está formada por tres cadenas de aminoácidos:
| Cadena A |
0-KEQRLKEQLGAQQEPVKKSIQESEAFLPQSIPEERYKMKSKPLGICLIIDCIGNETELLRDTFTSLGYEV
70-QKFLHLSMHGISQILGQFACMPEHRDYDSFVCVLVSRGGSQSVYGVDQTHSGLPLHHIRRMFMGDSCPYL
140-AGKPKMFFIQNYVVSEGQLENSSLLEVDGPAMKNVEFKAQKRGLCTVHREADFFWSLCTADMSLLEQSHS
210-SPSLYLQCLSQKLRQERKRPLLDLHIELNGYMYDWNSRVSAKEKYYVWLQHTLRKKLILSYT
|
| Cadena B |
0-SESQTLDKVYQMKSKPRGYCLIINNHNFAKAREKVPKLHSIRDRNGTHLDAGALTTTFEELHFEIKPHDD
70-CTVEQIYEILKIYQLMDHSNMDCFICCILSHGDKGIIYGTDGQEAPIYELTSQFTGLKCPSLAGKPKVFF
140-IQACQGDNYQKGIPVETASEEQPYLEMALSSPQTRYIPDEADFLLGMATVNNCVSYRNPAEGTWYIQSLC
210-QSLRERCPRGDDILTILTEVNYEVSNKDDKKNMGKQMPQPTFTLRKKLVFPSDVEHHHHHH
|
| Cadena C |
0-AIETX
|
Centro activo
Las proteínas c-FLIPL tienen dos centros activos, uno en la cadena A y otro en la cadena B. En la cadena A, el centro activo llega desde el aminoácido 107 al 152, pese a que los aminoácidos que actúan en la adhesión de la caspasa 8 a esta proteína se extienden entre los aminoácidos 107 y 214. En la cadena B la zona activa ocupa los aminoácidos que se encuentran entre el 104 y el 144, y la zona de la proteína encargada de fijar el sustrato que necesita su centro activo para funcionar está localizada entre los aminoácidos 44 y 204.
Además del centro activo, la cadena B tiene un dominio entre los aminoácidos 151 y 178 con capacidad de escisión proteolítica, es decir, de cortar una proteína y obtener cadenas de péptidos más cortas. En concreto la proteína c-FLIPL es capaz de dividir la caspasa 8 en dos dímeros inactivos, que no actúan en la señal de muerte celular.
Cabe destacar que ambos centros activos deben trabajar simultáneamente para que la proteína pueda cumplir su función de forma adecuada.
Función
La investigación sobre las proteínas c-FLIP comenzó al analizar su implicación en la homeostasis tisular como reguladores de la apoptosis.
La apoptosis celular se inicia al activarse el complejo proteico DISC5 (complejo de señalización de la muerte inducida, death inducing signaling complex). En él se encuentran ligadas la proteína receptora de muerte de la célula y las proteínas de muerte celular (Capasa 8, Fas, FADD,...). Para inhibir la formación de este complejo, las proteínas c-FLIP son capaces de interferir en las uniones proteicas entre efectores de muerte celular.
La falta de cualquiera de las proteínas del complejo DISC impide su efectividad. Una de las proteína clave del disc es la Caspasa 8 o FLICE, que se sintetiza en una forma inactiva y sólo se activa al homodimerizarse.
Las proteínas c-FLIPL se asemejan estructuralmente a estas proteasas FLICE, aunque en el caso de las FLIP el dominio caspasa no es funcional (la cisteína del centro activo está sustituida por una tirosina). Como los precursores de la caspasa se activan y autoprocesan como dímeros, la sobreexpresión de FLICE en las células se traduce en una alta presencia de dímeros y oligómeros de esta proteasa. Inicialmente se pensó que las FLIPL se comportarían de manera semejante por su similitud estructural, pero finalmente se comprobó que los dímeros que se formaban en presencia de proteasa e inhibidor de apoptosis eran entre caspasas funcionales y no funcionales, es decir, se establecían estrechas asociaciones entre FLIP y FLICE.
Como la muerte celular se produce cuando esta caspasa 8 se une al complejo de señalización DISC, si la proteína c-FLIP entra en el proceso impide la correcta homodimerización de FLICE y, por tanto, inhibe la formación del complejo DISC.
Ésta es la manera de explicar cómo las proteínas FLIPL son capaces de inhibir la apoptosis celular.
Experimentalmente se ha comprobado también que la forma corta de las proteínas FLIP, las FLIPS, son capaces igualmente de realizar una función inhibidora asociándose fuertemente con otras proteínas de muerte celular, en este caso las FADD. Estos experimentos se realizaron sobre todo con células T, debido a la elevada concentración de proteínas FLIP en ellas. Se utilizan sin embargo células T en estados de desarrollo tempranos, ya que la presencia de FLIP decae con el tiempo y estas células se hacen susceptibles a la apoptosis celular. De esta manera se llegó a la conclusión de que las proteínas FLIP son capaces de bloquear la muerte celular inducida por receptores de muerte de manera primaria.6
Sistema nervioso
La proteína c-FLIPL es imprescindible en el sistema nervioso por sus múltiples funciones. Impulsa la diferenciación de las células nerviosas así como el desarrollo de las conexiones interneuronales. Además, es responsable de la existencia y el crecimiento de algunas células nerviosas como las motoneuronas.
Durante la fase embrionaria c-FLIPL se expresa en diversas regiones del sistema nervioso y, a medida que éste se va desarrollando, se concentran en las neuronas. En una etapa más avanzada, la proteína forma parte de las células gliales.
Un déficit de c-FLIPL supone un desarrollo del sistema nervioso nulo, ya que las neurotrofinas por sí solas resultarían insuficientes para realizar este trabajo.7
Sistema inmunitario
Una función importante de las proteínas c-FLIP es la estimulación de la autofagocitosis de la célula, que se complementa con la inhibición de la apoptosis: la célula no muere pero renueva sus compartimentos, permitiendo que siga realizando sus funciones.
Las proteínas c-FLIP tienen una doble importancia en el funcionamiento de los linfocitos T. Las células T no sólo deben realizar la autofagocitosis para renovarse y poder reemplazar los compartimentos dañados, sino que tienen también que fagocitar los antígenos que quieren eliminar. Por eso un linfocito T con niveles bajos de c-FLIP, al detectar una antígeno, tiene una respuesta mucho menos efectiva que un linfocito T normal, y además se ha comprobado que mueren por apoptosis alrededor de 36 horas después de la detección de este antígeno.8 Esto se traduce en una respuesta inmunitaria muy deficiente, con graves consecuencias para el funcionamiento del organismo y para la salud.
Aplicaciones médicas
El nivel de expresión de c-FLIP es determinante en la resistencia frente a la apoptosis inducida por ligandos de muerte como TRAIL9 (además de los ya mencionados Fas, FADD, etc.).
Así como una cierta expresión de proteínas c-FLIP en la célula inhibe la apoptosis, se ha demostrado también que la presencia moderada de esta proteína en algunascélulas tumorales las sensibiliza a la muerte celular inducida.10 De igual forma, se comprobó experimentalmente que la transfección de altas concentraciones del vector de expresión de FLIPL solo (sin el de FLIPS) lleva a la muerte espontánea en varios tipos de linfocitos T. Esto conduce a considerar que los niveles de expresión de FLIPL pueden determinar la vida o la muerte de una célula. Por este motivo esta proteína resulta atractiva como diana en terapias antitumorales así como de enfermedades degenerativas, debido a su papel bifuncional en la regulación de la apoptosis.
En relación con las células tumorales, existen ya tratamientos (no en humanos) que son capaces de disminuir el nivel de expresión de la proteína FLIPL y, por tanto, de hacer a las células cancerosas susceptibles a la muerte celular. Estos tratamientos alteran la ruta de síntesis de las c-FLIP. Entre ellos se incluyen agentes que deterioran el ADN, inhibidores de la síntesis de proteínas e inhibidores de histonas deacetilasas.11 Otros inhibidores de proteínas, como algunas quinasas, contribuyen también a la disminución de los niveles de expresión de c-FLIP. La combinación de alguno de estos agentes con TRAIL resulta una terapia efectiva para la eliminación de células tumorales. El desarrollo de dichos inhibidores junto con el uso de ARN antisentido12 dirigidos específicamente contra c-FLIP es ahora uno de los nuevos campos de investigación en terapia antitumoral.
FLICE-like celular proteína inhibidora (c-FLIP) ha sido identificado como una proteasa muerto, regulador de la procaspasa-8-como de la muerte inducida por ligando apoptosis, basado en observaciones que c-FLIP impide factor de necrosis tumoral alfa (TNF-alfa ), Fas-L, y TNF-relacionados-ligando inductor de apoptosis (TRAIL) inducida por apoptosis mediante la unión a FADD y / o la caspasa-8 o -10 en una manera dependiente de ligando, que a su vez impide que induce a la muerte complejo de señalización ( DISC) la formación y la posterior activación de la cascada de caspasas. c-FLIP es una familia de variantes de corte y empalme alternativo, y existe principalmente como largo (c-FLIP (L)) y corto (c-FLIP (S)) variantes de empalme en las células humanas. Aunque c-FLIP tiene actividad apoptogenic en algunos contextos celulares, que actualmente está atribuirse a la heterodimerización con la caspasa-8 en el DISC, la acumulación de pruebas indica un papel anti-apoptótica de c-FLIP en diversos tipos de cánceres humanos. Por ejemplo, los pequeños RNAs de interferencia (siRNAs) que específicamente derribado expresión de c-FLIP (L) en diversas líneas celulares de cáncer humano, por ejemplo, de pulmón y cáncer de cuello uterino células, aumentada reclutamiento DISCO inducida por TRAIL, y por lo tanto una mayor estimulación caspasa efectora y apoptosis. Por lo tanto, las perspectivas para el índice terapéutico de drogas c-FLIP-objetivo parece excelente, no sólo de la eficacia observada en modelos experimentales de terapia contra el cáncer, sino también por la comprensión actual de la acción de doble c-FLIP en los tejidos normales apoya la noción de que c-FLIP-dirigida la terapia del cáncer se tolera bien.Curiosamente, Taxol, TRAIL, así como varias clases de moléculas pequeñas inducen downregulation c-FLIP en las células neoplásicas. Se están realizando esfuerzos para desarrollar fármacos de molécula pequeña que inducen la regulación a la baja de c-FLIP y otras terapias dirigidas contra el cáncer-c-FLIP. En esta revisión, evaluamos las perspectivas para mejorar el tratamiento del cáncer a través de la terapéutica c-FLIP-dirigida.


No hay comentarios:
Publicar un comentario