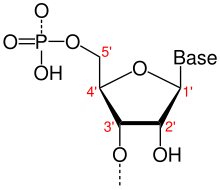
La direccionalidad , en biología molecular y bioquímica , es la orientación química de extremo a extremo de una sola cadena de ácido nucleico . En una sola hebra de ADN o ARN , la convención química de nombrar átomos de carbono en el anillo de azúcar del nucleótidosignifica que habrá un extremo 5 ' , que frecuentemente contiene un grupo fosfato unido al carbono 5' del anillo de ribosa , y un extremo 3 '(usualmente pronunciado "cinco extremos principales" y "tres extremos principales"), que típicamente no está modificado a partir del sustituyente ribosa-OH. En una doble hélice de ADN.Los filamentos se ejecutan en direcciones opuestas para permitir el emparejamiento de bases entre ellos, lo cual es esencial para la replicación o transcripción de la información codificada.
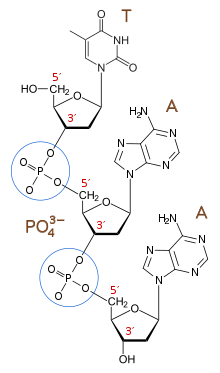
Los ácidos nucleicos solo se pueden sintetizar in vivo en la dirección 5'-a-3 ', ya que las polimerasas que ensamblan varios tipos de cadenas nuevas generalmente dependen de la energía producida al romper los enlaces del nucleósido trifosfato para unir nuevos monofosfatos de nucleósido a la 3'- Grupo hidroxilo (-OH), a través de un enlace fosfodiéster . Las posiciones relativas de las estructuras a lo largo de una hebra de ácido nucleico, incluidos los genes y diversos sitios de unión a proteínas , generalmente se señalan ya sea hacia arriba(hacia el extremo 5 ') o hacia abajo (hacia el extremo 3′). (Véase también en sentido ascendente y descendente .)
La direccionalidad se relaciona con, pero es independiente de, el sentido . La transcripción de ARN monocatenario de una plantilla de ADN bicatenario requiere la selección de una cadena de la plantilla de ADN como la cadena plantilla que interactúa directamente con el ARN naciente debido a una secuencia complementaria . La otra cadena no se copia directamente, pero necesariamente su secuencia será similar a la del ARN. Los sitios de inicio de la transcripción generalmente ocurren en ambas cadenas del ADN de un organismo, y especifican la ubicación, la dirección y las circunstancias bajo las cuales ocurrirá la transcripción. Si la transcripción codifica una o (rara vez) más proteínas , la traducción de cada proteína por el ribosomaprocederá en una dirección de 5 ′ a 3 ′, y extenderá la proteína desde su extremo N hacia su extremo C Por ejemplo, en un gen típico, un codón de inicio (5'-ATG-3 ') es una secuencia de ADN dentro de la cadena de sentido. La transcripción comienza en un sitio en sentido ascendente (en relación con la cadena de sentido) y, a medida que avanza a través de la región, copia el 3′-TAC-5 'de la cadena de plantilla para producir 5′-AUG-3' dentro de un ARN mensajero . El mRNA es escaneado por el ribosoma desde el extremo 5 ', donde el codón de inicio dirige la incorporación de una metionina (los procariotas usan N-formilmetionina en su lugar) en el extremo N de la proteína. Por convención, cadenas sencillas de ADN y ARN. las secuencias se escriben en una dirección de 5′ a 3 ′, excepto cuando sea necesario para ilustrar el patrón de emparejamiento de bases.
5′-end [ editar ]
El extremo 5 '(que se pronuncia como "fin de cinco") designa el extremo de la cadena de ADN o ARN que tiene el quinto carbono en el anillo de azúcar de la desoxirribosa o ribosa en su extremo. Un grupo fosfatounido al extremo 5 'permite la unión de dos nucleótidos , es decir, la unión covalente de un 5'-fosfato al grupo 3'-hidroxilo de otro nucleótido, para formar un enlace fosfodiéster . La eliminación del fosfato 5 'previene la ligadura. Para prevenir la ligadura de ácido nucleico no deseada (por ejemplo, autoligado de un vector plasmídico en la clonación de ADN ), los biólogos moleculares comúnmente eliminan el 5'-fosfato con unafosfatasa .
El extremo 5 'del ARN mensajero naciente es el sitio en el que se produce el límite posterior a la transcripción , un proceso que es vital para producir ARN mensajero maduro. La limitación aumenta la estabilidad del ARN mensajero mientras sufre una traducción , lo que proporciona resistencia a los efectos degradantes de las exonucleasas . [ cita requerida ] Consiste en un nucleótido metilado ( metilguanosina ) unido al ARN mensajero en un enlace raro de 5'-a-5'-trifosfato.
La región flanqueante en 5 ' de un gen a menudo denota una región de ADN que no se transcribe en ARN. La región flanqueante 5 'contiene el promotor del gen , y también puede contener potenciadores u otros sitios de unión a proteínas.
La región 5 ' no traducida (5'-UTR) es una región de un gen que se transcribe en el ARNm, y se encuentra en el extremo 5' del ARNm. Esta región de un ARNm puede o no traducirse , pero generalmente está involucrada en la regulación de la traducción. La región 5 'no traducida es la porción del ADN que comienza en el sitio de la tapa y se extiende a la base justo antes del codón de inicio de la traducción AUG de la secuencia de codificación principal. Esta región puede tener secuencias, como el sitio de unión al ribosoma y la secuencia de Kozak , que determinan la eficiencia de traducción del ARNm, o que pueden afectar la estabilidad del ARNm.
3′-end [ editar ]
El extremo 3 '(tres primarios) de una cadena se denomina así debido a que termina en el grupo hidroxilo del tercer carbono en el anillo de azúcar , y se conoce como el extremo de la cola . El 3'-hidroxilo es necesario en la síntesis de nuevas moléculas de ácido nucleico, ya que está ligado (unido) al 5'-fosfato de un nucleótido separado, lo que permite la formación de cadenas de nucleótidos unidos.
Los biólogos moleculares pueden usar nucleótidos que carecen de un 3'-hidroxilo (dideoxirribonucleótidos) para interrumpir la replicación del ADN. Esta técnica se conoce como el método de terminación de cadena dideoxi o el método de Sanger, y se utiliza para determinar el orden de los nucleótidos en el ADN .
El extremo 3 'del ARN mensajero naciente es el sitio de la poliadenilación postranscripcional , que une una cadena de 50 a 250 residuos de adenosina para producir ARN mensajero maduro. Esta cadena ayuda a determinar cuánto tiempo dura el ARN mensajero en la célula, lo que influye en la cantidad de proteína que se produce a partir de ella.
La región flanqueante 3 ' es una región de ADN que no se copia en el ARNm maduro, pero que está presente adyacente al extremo 3' del gen. Originalmente se pensó que el ADN que flanqueaba 3 'no se transcribía en absoluto, pero se descubrió que se transcribía en ARN y se eliminaba rápidamente durante el procesamiento de la transcripción primaria para formar el ARNm maduro. La región del flanco 3′ a menudo contiene secuencias que afectan la formación del extremo 3′ del mensaje. También puede contener potenciadores u otros sitios a los que se pueden unir las proteínas.
La región 3 ' no traducida (3'-UTR) es una región del ADN que se transcribe en ARNm y se convierte en el extremo 3' del mensaje, pero que no contiene la secuencia codificante de la proteína. Todo lo que se encuentra entre el codón de parada y la cola poliA se considera no traducido en 3′. La región 3 'no traducida puede afectar la eficiencia de traducción del ARNm o la estabilidad del ARNm. También tiene secuencias que son necesarias para la adición de la cola poli (A) al mensaje, incluido el hexanucleótido AAUAAA.
En biología molecular , el cap de cinco primos ( cap 5 ' ) es un nucleótido especialmente alterado en el extremo 5' de algunas transcripciones primarias , como el ARN mensajero precursor . Este proceso, conocido como cobertura de ARNm , está altamente regulado y es vital en la creación de un ARN mensajero estable y madurocapaz de someterse a la traducción durante la síntesis de proteínas . El ARNm mitocondrial [1] y el ARNm cloroplástico [2] no están limitados.
Estructura [ editar ]
En los eucariotas , el cap 5 '(cap-0), que se encuentra en el extremo 5' de una molécula de mRNA, consiste en un nucleótido de guaninaconectado al mRNA a través de un inusual enlace trifosfato de 5 'a 5' . Esta guanosina se metila en la posición 7 directamente después de la limitación in vivo por una metiltransferasa . [3] [4] [5] [6] Se conoce como una tapa de 7-metilguanyilato , abreviada m 7 G.
En eucariotas multicelulares y algunos virus, [7] existen otras modificaciones, incluida la metilación de los grupos hidroxi 2 ' de los primeros 2 azúcares ribosa del extremo 5' del ARNm. cap-1 tiene un grupo 2'-hidroxi metilado en el primer azúcar ribosa, mientras que cap-2 tiene grupos 2'-hidroxi metilados en los dos primeros azúcares ribosa, que se muestran a la derecha. La tapa 5 'es químicamente similar al extremo 3' de una molécula de ARN (el carbono 5 'de la ribosa de la tapa está unido, y la 3' no se ha unido). Esto proporciona una resistencia significativa a las exonucleasas 5 ' . [ cita requerida ]
Los pequeños ARN nucleares contienen 5'-caps únicos. Los snRNA de clase Sm se encuentran con las cápsulas de 5'-trimetilguanosina, mientras que los snRNA de clase Lsm se encuentran con las tapas de 5'-monometilfosfato. [8]
En las bacterias , y potencialmente también en organismos superiores, algunos ARN se tapan con NAD + , NADH , o 3 'dephospho-coenzima A . [9] [10]
En todos los organismos, las moléculas de ARNm pueden ser destapadas en un proceso conocido como decapado de ARN mensajero .
Proceso de limitación [ editar ]
El punto de partida para cubrir con 7-metilguanilato es el extremo 5 'inalterado de una molécula de ARN, que termina en un grupo trifosfato. Esto presenta un nucleótido final seguido de tres grupos fosfato unidos al carbono 5 '. [3] El proceso de limitación se inicia antes de la finalización de la transcripción, ya que se está sintetizando el pre-ARNm naciente.
- Uno de los grupos fosfato terminales se elimina mediante la ARN trifosfatasa , dejando un grupo bisfosfato (es decir, 5 '(ppN) [pN] n );
- El GTP se agrega al bisfosfato terminal mediante la ARNm guanililtransferasa , perdiendo un pirofosfatodel sustrato de GTP en el proceso. Esto da como resultado el enlace trifosfato 5′ – 5 ', que produce 5' (Gp) (ppN) [pN] n ;
- El 7-nitrógeno de la guanina es metilado por mRNA (guanina N 7 -) - metiltransferasa , con S -adenosyl- L-metionina siendo desmetiló para producir S -adenosyl- L : la homocisteína , resultando en 5' (m7Gp) (PPN) [pN] n (cap-0);
- Pueden ocurrir modificaciones adyacentes a la tapa, normalmente en el primer y segundo nucleótidos, produciendo hasta 5 '(m7Gp) (ppN *) (pN *) [pN] n (cap-1 y cap-2); [7]
- Si el nucleótido cap-adyacente más cercano es 2'- O ribosa metil-adenosina (es decir, 5' (m7Gp) (PDMA) [pN] n ), puede ser más metilado en la posición de metilo N6 para formar N 6 -methyladenosine , dando como resultado 5 '(m7Gp) (ppm6Am) [pN] n . [3]
El mecanismo de cobertura con NAD + , NADH o 3'-dephospho-coenzima A es diferente. La limitación con NAD +, NADH o 3'-defosfo-conenzima A se logra a través de un "mecanismo de limitación ab initio", en el que NAD + , NADH o 3'-desfosfo-coenzima A sirve como un "nucleótido de inicio no canónico "(NCIN) para el inicio de la transcripción por la ARN polimerasa y, por lo tanto, se incorpora directamente al producto de ARN. [9] Tanto la ARN polimerasa bacteriana como la ARN polimerasa II eucariótica son capaces de llevar a cabo este "mecanismo de cobertura ab initio". [9]
Orientación [ editar ]
Para cubrir con 7-metilguanilato, el complejo enzimático de cobertura (CEC) se une a la ARN polimerasa II antes de que comience la transcripción. Tan pronto como el extremo 5 'del nuevo transcrito emerge de la ARN polimerasa II, la CEC lleva a cabo el proceso de limitación (este tipo de mecanismo garantiza la limitación, como ocurre con la poliadenilación ). [11] [12] [13] [14] Las enzimas para la cobertura solo pueden unirse a la ARN polimerasa II , lo que garantiza la especificidad solo de estas transcripciones, que son casi completamente ARNm. [12] [14]
El límite con NAD + , NADH o 3'-dephospho-coenzima A se dirige a la secuencia del promotor . [9] El límite con NAD +, NADH o 3'-defosfo-coenzima A se produce solo en los promotores que tienen ciertas secuencias en el sitio de inicio de la transcripción e inmediatamente después del mismo, y por lo tanto se produce solo para los ARN sintetizados a partir de ciertos promotores. [9]
Función [ editar ]
La tapa 5 'tiene cuatro funciones principales:
- Regulación de la exportación nuclear; [15] [16]
- Prevención de la degradación por exonucleasas ; [9] [17] [18] [19]
- Promoción de la traducción (ver ribosoma y traducción ); [3] [4] [5]
- Promoción de la escisión del intrón proximal en 5 '. [20]
La exportación nuclear de ARN está regulada por el complejo de unión de la tapa (CBC), que se une exclusivamente al ARN cubierto con 7-metilguanyilato. El CBC es entonces reconocido por el complejo de poros nucleares y se exporta. Una vez en el citoplasma después de la ronda pionera de traducción, el CBC se reemplaza por los factores de traducción eIF4E y eIF4G del complejo eIF4F . [6] Este complejo es reconocido por otra maquinaria de iniciación de la traducción, incluido el ribosoma. [21]
El tapado con 7-metilguanylate previene la degradación de 5 'de dos maneras. Primero, se evita la degradación del ARNm por exonucleasas 5 '(como se mencionó anteriormente) al parecer funcionalmente un extremo 3'. En segundo lugar, el CBC y eIF4E / eIF4G bloquean el acceso de las enzimas de descapsulación a la tapa. Esto aumenta la vida media del ARNm, esencial en los eucariotas, ya que los procesos de exportación y traducción llevan mucho tiempo.
El decapado de un ARNm cubierto con 7-metilguanilato es catalizado por el complejo de destape formado por al menos Dcp1 y Dcp2, que debe competir con eIF4E para unirse al cap. Por lo tanto, la tapa de 7-metilguanilato es un marcador de un ARNm que se traduce activamente y es utilizado por las células para regular las semividas del ARNm en respuesta a nuevos estímulos. Los ARNm no deseados se envían a los cuerpos P para almacenamiento temporal o decapado, cuyos detalles aún se están resolviendo. [22]
El mecanismo de la promoción de la escisión del intrón proximal en 5 'no se conoce bien, pero la tapa del 7-metilguanilato parece girar e interactuar con el spliceosome en el proceso de empalme, lo que promueve la escisión del intrón.
Un activador transcripcional es una proteína ( factor de transcripción ) que aumenta la transcripción de un gen o un conjunto de genes. La mayoría de los activadores son proteínas de unión al ADN que se unen a los potenciadores o elementos promotores proximales .
La mayoría de los activadores funcionan mediante la secuencia de unión específicamente a un sitio de ADN ubicado en o cerca de un promotor y realizando interacciones proteína-proteína con la maquinaria de transcripción general ( ARN polimerasa y factores de transcripción generales ), facilitando así la unión de la maquinaria de transcripción general al promotor . El sitio de ADN enlazado por el activador se conoce como un "sitio activador". La parte del activador que produce las interacciones proteína-proteína con la maquinaria de transcripción general se conoce como una "región activadora". La parte de la maquinaria de transcripción general que realiza las interacciones proteína-proteína con el activador se conoce como un "objetivo de activación".
Ejemplo [ editar ]
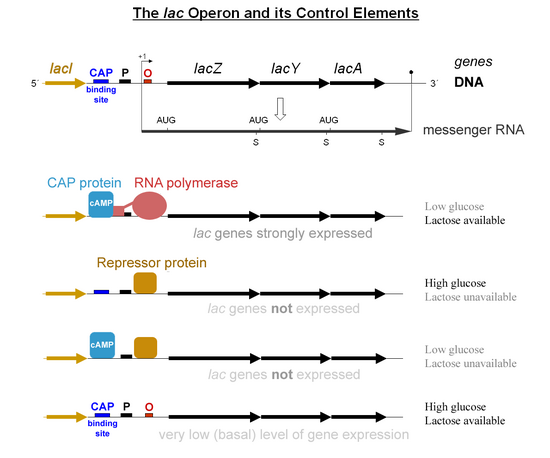
La proteína activadora de catabolitos (CAP; también conocida como proteína receptora de cAMP , CRP) activa la transcripción en el operón lac de la bacteria Escherichia coli . [1] El monofosfato de adenosina cíclico (AMPc) se produce durante la inanición de la glucosa, se une a la PAC, causa un cambio conformacional que permite que la PAC se una a un sitio de ADN ubicado adyacente al promotor lac. Luego, CAP realiza una interacción proteína-proteína directa con la ARN polimerasa que recluta la ARN polimerasa al promotor lac.


No hay comentarios:
Publicar un comentario