El código genético es el conjunto de reglas utilizadas por las célulasvivas para traducir información codificada dentro del material genético ( secuencias de ADN o ARNm ) en proteínas . La traducción se realiza por el ribosoma , que une los aminoácidos en un orden especificado por el ARN mensajero (ARNm), utilizando moléculas de ARN de transferencia(ARNt) para transportar aminoácidos y leer el ARNm tres nucleótidos a la vez. El código genético es altamente similar entre todos los organismos y se puede expresar en una tabla simple con 64 entradas. [1]
El código define cómo las secuencias de tripletes de nucleótidos, llamados codones , especifican qué aminoácido se agregará a continuación durante la síntesis de proteínas . Con algunas excepciones, [2] un codón de tres nucleótidos en una secuencia de ácido nucleico especifica un solo aminoácido. La gran mayoría de los genes están codificados con un solo esquema (consulte la tabla de codones de ARN). Ese esquema se refiere a menudo como el código genético canónica o estándar, o simplemente el código genético, aunque códigos de variante(como en humanos mitocondrias existen).
Mientras que el "código genético" determina la secuencia de aminoácidos de una proteína, otras regiones genómicas determinan cuándo y dónde se producen estas proteínas de acuerdo con varios "códigos reguladores de genes".
Historia [ editar ]
Los esfuerzos para comprender cómo se codifican las proteínas comenzaron después de que se descubrió la estructura del ADN en 1953. George Gamow postuló que deben emplearse conjuntos de tres bases para codificar los 20 aminoácidos estándar utilizados por las células vivas para construir proteínas, lo que permitiría un máximo de 4 3 = 64 aminoácidos. [3]
Codones [ editar ]
El experimento de Crick, Brenner, Barnett y Watts-Tobin primero demostró que los codones consisten en tres bases de ADN. Marshall Nirenberg y Heinrich J. Matthaei fueron los primeros en revelar la naturaleza de un codón en 1961.
Se utilizó un sistema libre de células para traducir un poli- uracilosecuencia de ARN (es decir, UUUUU ...) y descubrieron que el polipéptido que habían sintetizado consistía en sólo el aminoácido fenilalanina . [4] Por lo tanto, dedujeron que el codón UUU especificaba el aminoácido fenilalanina.
Esto fue seguido por experimentos en Severo Ochoa laboratorio 's que demostraban que el poli- adeninasecuencia de ARN (AAAAA ...) codifica para el polipéptido poli- lisina [5] y que el poli- citosina secuencia de ARN (CCCCC ...) Codificado para el polipéptido poli-prolina. [6] Por lo tanto, el codón AAA especificó el aminoácido lisina y el codón CCC especificó el aminoácido prolina . Usando varios copolímeros, se determinó la mayoría de los codones restantes.
El trabajo posterior de Har Gobind Khorana identificó el resto del código genético. Poco después, Robert W. Holley determinó la estructura del ARN de transferencia (ARNt), la molécula adaptadora que facilita el proceso de traducción del ARN en proteínas. Este trabajo se basó en los estudios anteriores de Ochoa, otorgando a este último el Premio Nobel de Fisiología o Medicina en 1959 por el trabajo sobre la enzimología de la síntesis de ARN. [7]
Extendiendo este trabajo, Nirenberg y Philip Leder revelaron la naturaleza del triplete del código y descifraron sus codones. En estos experimentos, se pasaron varias combinaciones de ARNm a través de un filtro que contenía ribosomas , los componentes de las células que traducen el ARN en proteína. Tripletes únicos promovieron la unión de ARNt específicos al ribosoma. Leder y Nirenberg pudieron determinar las secuencias de 54 de 64 codones en sus experimentos. [8] Khorana, Holley y Nirenberg recibieron al Nobel de 1968 por su trabajo. [9]
Los tres codones de parada fueron nombrados por los descubridores Richard Epstein y Charles Steinberg. "Ámbar" lleva el nombre de su amigo Harris Bernstein, cuyo apellido significa "ámbar" en alemán. [10] Los otros dos codones de parada fueron llamados "ocre" y "opal" para mantener el tema de "nombres de colores".
Códigos genéticos expandidos (biología sintética) [ editar ]
En una amplia audiencia académica, el concepto de la evolución del código genético del código genético original y ambiguo a un código bien definido ("congelado") con el repertorio de 20 (+2) aminoácidos canónicos es ampliamente aceptado. [11] Sin embargo, hay diferentes opiniones, conceptos, enfoques e ideas, que es la mejor manera de cambiarlo experimentalmente. Incluso se proponen modelos que predicen "puntos de entrada" para la invasión de aminoácidos sintéticos del código genético. [12]
Desde 2001, se han agregado 40 aminoácidos no naturales a la proteína mediante la creación de un codón único (recodificación) y un par correspondiente de transferencia ARN: aminoacil - ARNt-sintetasa para codificarlo con diversas propiedades fisicoquímicas y biológicas con el fin de utilizarse como Una herramienta para explorar la estructura y función de las proteínas o para crear proteínas nuevas o mejoradas. [13] [14]
H. Murakami y M. Sisido extendieron algunos codones para tener cuatro y cinco bases. Steven A. Bennerconstruyó un codón funcional 65 ( in vivo ). [15]
En 2015, N. Budisa , D. Söll y colaboradores informaron la sustitución total de todos los 20,899 residuos de triptófano (codones UGG) con tienopirrol-alanina no natural en el código genético de la bacteria Escherichia coli . [dieciséis]
En 2016 se creó el primer organismo semisintético estable. Era una bacteria (de una sola célula) con dos bases sintéticas (llamadas X e Y). Las bases sobrevivieron a la división celular. [17] [18]
En 2017, investigadores en Corea del Sur informaron que habían diseñado un ratón con un código genético extendido que puede producir proteínas con aminoácidos no naturales. [19]
En mayo de 2019, los investigadores, en un hito, informaron sobre la creación de una nueva forma sintética(posiblemente artificial ) de vida viable , una variante de la bacteria Escherichia coli , al reducir el número natural de 64 codones en el genoma bacteriano a 59 codones en cambio, para codificar 20 aminoácidos . [20] [21]
Características [ editar ]
Marco de lectura [ editar ]
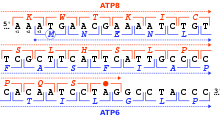
Un marco de lectura se define por el triplete inicial de nucleótidos a partir del cual comienza la traducción. Establece el marco para una serie de codones sucesivos que no se superponen, lo que se conoce como " marco de lectura abierto " ( ORF ). Por ejemplo, la cadena 5'-AAATGAACG-3 '(ver figura), si se lee desde la primera posición, contiene los codones AAA, TGA y ACG; si se lee desde la segunda posición, contiene los codones AAT y GAA; y si se lee desde la tercera posición, contiene los codones ATG y AAC. Por lo tanto, cada secuencia puede leerse en su dirección 5 '→ 3' en tres marcos de lecturaCada uno de ellos produce una secuencia de aminoácidos posiblemente distinta: en el ejemplo dado, Lys (K) -Trp (W) -Thr (T), Asn (N) -Glu (E) o Met (M) -Asn (N) , respectivamente (al traducir con el código mitocondrial de vertebrados). Cuando el ADN es de doble cadena, se definen seis marcos de lectura posibles , tres en la orientación hacia adelante en una cadena y tres en la cadena opuesta. [23] : 330 marcos de codificación de proteínas se definen por un codón de inicio , generalmente el primer codón AUG (ATG) en la secuencia de ARN (ADN).
Iniciar / detener codones [ editar ]
La traducción comienza con un codón de inicio de cadena o un codón de inicio . El codón de inicio por sí solo no es suficiente para comenzar el proceso. También se requieren secuencias cercanas como la secuencia Shine-Dalgarno en E. coli y factores de iniciación para comenzar la traducción. El codón de inicio más común es AUG, que se lee como metionina o, en bacterias, como formilmetionina . Los codones de inicio alternativos que dependen del organismo incluyen "GUG" o "UUG"; estos codones normalmente representan valina y leucina , respectivamente, pero como codones de inicio se traducen como metionina o formilmetionina. [24]
Los tres codones de parada tienen nombres: UAG es ámbar , UGA es ópalo (a veces también se llama umber ) y UAA es ocre . Los codones de parada también se denominan codones de "terminación" o "sin sentido". Señalan la liberación del polipéptido naciente desde el ribosoma porque ningún ARNt análogo tiene anticodones complementarios a estas señales de parada, lo que permite que un factor de liberación se una al ribosoma. [25]
Efecto de las mutaciones [ editar ]
Durante el proceso de replicación del ADN , ocasionalmente se producen errores en la polimerización de la segunda cadena. Estos errores, mutaciones , pueden afectar el fenotipo de un organismo , especialmente si ocurren dentro de la secuencia codificadora de proteínas de un gen. Las tasas de error suelen ser de 1 error en cada 10 a 100 millones de bases, debido a la capacidad de "corrección de pruebas" de las ADN polimerasas . [27] [28]
Las mutaciones sin sentido y las mutaciones sin sentido son ejemplos de mutaciones puntuales que pueden causar enfermedades genéticas como la enfermedad de células falciformes y la talasemia, respectivamente. [29] [30] [31] Las mutaciones missense clínicamente importantes generalmente cambian las propiedades del residuo de aminoácido codificado entre estados básicos, ácidos, polares o no polares, mientras que las mutaciones sin sentido dan como resultado un codón de parada . [23] : 266
Las mutaciones que interrumpen la secuencia del marco de lectura por indels ( inserciones o eliminaciones ) de un no múltiplo de 3 bases de nucleótidos se conocen como mutaciones de cambio de marco . Estas mutaciones generalmente resultan en una traducción completamente diferente del original, y probablemente causen la lectura de un codón de parada , que trunca la proteína. [32] Estas mutaciones pueden afectar la función de la proteína y, por lo tanto, son raras en vivoSecuencias codificantes de proteínas. Una de las razones por las que la herencia de las mutaciones de cambio de marco es rara es que, si la proteína que se traduce es esencial para el crecimiento bajo las presiones selectivas a las que se enfrenta el organismo, la ausencia de una proteína funcional puede causar la muerte antes de que el organismo se vuelva viable. [33] Las mutaciones de cambio de marco pueden provocar enfermedades genéticas graves, como la enfermedad de Tay-Sachs . [34]
Aunque la mayoría de las mutaciones que cambian las secuencias de proteínas son dañinas o neutrales, algunas mutaciones tienen beneficios. [35] Estas mutaciones pueden permitir al organismo mutante resistir tensiones ambientales particulares mejor que los organismos de tipo salvaje , o reproducirse más rápidamente. En estos casos, una mutación tenderá a ser más común en una población a través de la selección natural . [36] Los virusque utilizan el ARN como material genético tienen tasas de mutación rápidas, [37] lo que puede ser una ventaja, ya que estos virus evolucionan rápidamente y, por lo tanto, evaden las respuestas defensivas del sistema inmunológico . [38]En grandes poblaciones de organismos que se reproducen asexualmente, por ejemplo, E. coli , pueden coexistir múltiples mutaciones beneficiosas. Este fenómeno se denomina interferencia clonal y causa competencia entre las mutaciones. [39]
Degeneración [ editar ]
La degeneración es la redundancia del código genético. Este término fue dado por Bernfield y Nirenberg. El código genético tiene redundancia pero no ambigüedad (consulte las tablas de codones a continuación para ver la correlación completa). Por ejemplo, aunque los codones GAA y GAG especifican ácido glutámico(redundancia), ninguno especifica otro aminoácido (sin ambigüedad). Los codones que codifican un aminoácido pueden diferir en cualquiera de sus tres posiciones. Por ejemplo, el aminoácido leucina se especifica mediante YU R o CU N (UUA, UUG, CUU, CUC, CUA o CUG) codones (diferencia en la primera o tercera posición indicada mediante notación IUPAC ), mientras que el aminoácido serina está especificado por UC No los codones AG Y(UCA, UCG, UCC, UCU, AGU o AGC) (diferencia en la primera, segunda o tercera posición). [40] : 102–117: 521–522 Una consecuencia práctica de la redundancia es que los errores en la tercera posición del tripón del codón causan solo una mutación silenciosa o un error que no afectaría a la proteína debido a la hidrofilia o hidrofobiase mantiene por sustitución equivalente de aminoácidos; por ejemplo, un codón de NUN (donde N = cualquier nucleótido) tiende a codificar aminoácidos hidrófobos. NCN produce residuos de aminoácidos que son pequeños en tamaño y moderados en hidropatía; NAN codifica residuos hidrofílicos de tamaño medio. El código genético está tan bien estructurado para la hidropatía que un análisis matemático ( Descomposición de Valor Singular ) de 12 variables (4 nucleótidos x 3 posiciones) produce una notable correlación (C = 0.95) para predecir la hidropatía del aminoácido codificado directamente desde el Secuencia de nucleótidos triplete, sin traducción. [41] [42] Observe en la tabla a continuación, ocho aminoácidos no están afectados en absoluto por las mutaciones en la tercera posición del codón, mientras que en la figura anterior, una mutación en la segunda posición es probable que cause un cambio radical en las propiedades fisicoquímicas de aminoácido codificado. Sin embargo, los cambios en la primera posición de los codones son más importantes que los cambios en la segunda posición en una escala global. [43] La razón puede ser que la inversión de carga (de una carga positiva a una negativa o viceversa) solo puede ocurrir en mutaciones en la primera posición, pero nunca en cambios en la segunda posición de un codón. Dicha inversión de carga puede tener consecuencias dramáticas para la estructura o función de una proteína. Este aspecto puede haber sido subestimado en gran medida por estudios anteriores.
Sesgo de uso del codón [ editar ]
La frecuencia de los codones, también conocida como sesgo de uso del codón , puede variar de una especie a otra con implicaciones funcionales para el control de la traducción . La siguiente tabla de uso de codones es para el genoma humano. [44]
| espectáculo
Tabla de frecuencia del codón del genoma humano
|
|---|
Tablas de codones estándar [ editar ]
Tabla ARN codón [ editar ]
| Propiedades bioquímicas de losaminoácidos . | no polar | polar | BASIC | ácido | Terminación: parar el codón |
| 1ra base | 2da base | 3ra base | |||||||
|---|---|---|---|---|---|---|---|---|---|
| U | do | UNA | sol | ||||||
| U | UUU | (Phe / F)Phenylalanine | UCU | (Ser / S)Serina | UAU | (Tyr / Y)Tirosina | UGU | (Cys / C)Cisteína | U |
| UUC | UCC | UAC | UGC | do | |||||
| UUA | (Leu / L)Leucina | UCA | UAA | Parada ( ocre) [B] | UGA | Stop ( Opal) [B] | UNA | ||
| UUG[A] | UCG | UAG | Detener (ámbar ) [B] | UGG | (Trp / W)triptófano | sol | |||
| do | CUU | CCU | (Pro / P)Prolina | CAU | (His / H)Histidina | CGU | (Arg / R)Arginina | U | |
| CUC | CCC | CAC | CGC | do | |||||
| CUA | CCA | CAA | (Gln / Q)Glutamina | CGA | UNA | ||||
| CUG[A] | CCG | CAG | CGG | sol | |||||
| UNA | AUU | (Ile / I)Isoleucina | ACU | (Thr / T)treonina | AAU | (Asn / N)Asparagine | AGU | (Ser / S)Serina | U |
| AUC | ACC | AAC | AGC | do | |||||
| AUA | ACA | AAA | (Lys / K)Lisina | AGA | (Arg / R)Arginina | UNA | |||
| AUG[A] | (Met / M)Metionina | ACG | AAG | AGG | sol | ||||
| sol | GUU | (Val / V) Valina | GCU | (Ala / A)Alanina | GAU | (Asp / D)Ácido aspártico | GGU | (Gly / G)Glicina | U |
| GUC | GCC | GAC | GGC | do | |||||
| GUA | GCA | GAA | (Glu / E) ácido glutámico | GGA | UNA | ||||
| GUG | GCG | MORDAZA | GGG | sol | |||||
- A El codón AUG codifica la metionina y sirve como un sitio de iniciación: el primer AUG enla región de codificación deunARNmes donde comienza la traducción a la proteína. [45]Los otros codones de inicio enumerados por GenBank son raros en los eucariotas y generalmente codifican para Met / fMet. [46]
- B ^ ^ ^ La base histórica para designar los codones de parada como ámbar, ocre y ópalo se describe en una autobiografía de Sydney Brenner [47] y en un artículo histórico de Bob Edgar. [48]
| Aminoácidos | Codones de ADN | Comprimido | Aminoácidos | Codones de ADN | Comprimido | |
|---|---|---|---|---|---|---|
| Ala / A | GCU, GCC, GCA, GCG | GCN | Leu / L | UUA, UUG, CUU, CUC, CUA, CUG | Tu, cun | |
| Arg / R | CGU, CGC, CGA, CGG, AGA, AGG | CGN, AGR | Lys / K | AAA, AAG | AAR | |
| Asn / N | AAU, AAC | Aay | Met / M | AGO | ||
| Asp / D | GAU, GAC | GAY | Phe / f | UUU, UUC | UUY | |
| Cys / C | UGU, UGC | UGY | Pro / P | CCU, CCC, CCA, CCG | CCN | |
| Gln / Q | CAA, CAG | COCHE | Ser / S | UCU, UCC, UCA, UCG, AGU, AGC | UCN, AGY | |
| Pegamento | GAA, GAG | GAR | Thr / T | ACU, ACC, ACA, ACG | ACN | |
| Gly / G | GGU, GGC, GGA, GGG | GGN | Trp / W | UGG | ||
| Su / h | CAU, CAC | ISLA PEQUEÑA | Tyr / Y | UAU, UAC | UAY | |
| Ile / I | AUU, AUC, AUA | AUH | Val / V | GUU, GUC, GUA, GUG | PISTOLA | |
| COMIENZO | AGO | DETENER | UAA, UGA, UAG | URA, UAG | ||
Tabla de codones de ADN [ editar ]
Códigos genéticos alternativos [ editar ]
Aminoácidos no estándar [ editar ]
En algunas proteínas, los aminoácidos no estándar se sustituyen por codones de parada estándar, dependiendo de las secuencias de señal asociadas en el ARN mensajero. Por ejemplo, UGA puede codificar para selenocisteína y UAG puede codificar para pirrolisina . La selenocisteína llegó a ser vista como el aminoácido 21 y la pirrolisina como el 22. [49] A diferencia de la selenocisteína, la UAG codificada por pirrolisina se traduce con la participación de una aminoacil-ARNt sintetasa dedicada . [50] Tanto la selenocisteína como la pirrolisina pueden estar presentes en el mismo organismo. [51] Aunque el código genético normalmente se fija en un organismo, el procariota achaeal Acetohalobium arabaticumPuede expandir su código genético de 20 a 21 aminoácidos (incluyendo la pirrolisina) en diferentes condiciones de crecimiento. [52]
Variaciones [ editar ]
Las variaciones en el código estándar se predijeron en la década de 1970. [53] El primero fue descubierto en 1979, por investigadores que estudian genes mitocondriales humanos . [54] Más adelante se descubrieron muchas variantes leves, [49] incluyendo varios códigos mitocondriales alternativos. [55] Estas variantes menores incluyen, por ejemplo, la traducción del codón UGA como triptófano en especies de Mycoplasma y la traducción de CUG como serina en lugar de leucina en levaduras del "clado CTG" (como Candida albicans ). [56] [57] [58]Debido a que los virus deben usar el mismo código genético que sus anfitriones, las modificaciones al código genético estándar podrían interferir con la síntesis o el funcionamiento de las proteínas virales. Sin embargo, virus como los totivirus se han adaptado a la modificación del código genético del huésped. [59] En bacterias y arqueas , GUG y UUG son codones de inicio comunes. En casos raros, ciertas proteínas pueden usar codones de inicio alternativos. [49]Sorprendentemente, también existen variaciones en la interpretación del código genético en los genes codificados en el núcleo humano: en 2016, los investigadores que estudian la traducción de malato deshidrogenasa encontraron que en aproximadamente el 4% de los ARNm que codifican esta enzima se usa naturalmente el codón de parada Para codificar los aminoácidos triptófano y arginina.[60] Este tipo de recodificación es inducida por un contexto de codón de parada de lectura alta [61] y se conoce como lectura traduccional funcional . [62]
Los códigos genéticos variantes utilizados por un organismo pueden inferirse identificando genes altamente conservados codificados en ese genoma y comparando su uso de codones con los aminoácidos en proteínas homólogas de otros organismos. Por ejemplo, el programa FACIL [63] infiere un código genético al buscar qué aminoácidos en los dominios de proteínas homólogas están más a menudo alineados con cada codón. Las probabilidades de aminoácidos resultantes para cada codón se muestran en un logotipo de código genético, que también muestra el soporte para un codón de parada.
A pesar de estas diferencias, todos los códigos conocidos de origen natural son muy similares. El mecanismo de codificación es el mismo para todos los organismos: codones de tres bases, ARNt , ribosomas, lectura de una sola dirección y traducción de codones individuales en aminoácidos individuales. [64]
Lista de codones alternativos [ editar ]
| espectáculo
Lista de codones alternativos
|
|---|
Origen [ editar ]
El código genético es una parte clave de la historia de la vida , según la cual las moléculas de ARN autorreplicables precedieron a la vida tal como la conocemos. La principal hipótesis para el origen de la vida es la hipótesis del mundo del ARN . Cualquier modelo para la aparición del código genético está íntimamente relacionado con un modelo de transferencia de ribozimas (enzimas de ARN) a proteínas como las principales enzimas en las células. De acuerdo con la hipótesis del mundo del ARN, las moléculas de ARN de transferencia parecen haber evolucionado antes de las sintetasas modernas de aminoacil-ARNt , por lo que estas últimas no pueden ser parte de la explicación de sus patrones. [sesenta y cinco]
Un hipotético código genético desarrollado al azar motiva aún más a un modelo bioquímico o evolutivo para su origen. Si los aminoácidos se asignaran al azar a los codones de tripletes, habría 1.5 × 10 84 códigos genéticos posibles. [66] : 163 Este número se encuentra al calcular el número de formas en que se pueden colocar 21 elementos (20 aminoácidos más una parada) en 64 contenedores, en donde cada elemento se usa al menos una vez. [67] Sin embargo, la distribución de las asignaciones de codones en el código genético no es aleatoria. [68]En particular, el código genético agrupa ciertas asignaciones de aminoácidos.
Los aminoácidos que comparten la misma vía biosintética tienden a tener la misma primera base en sus codones. Esto podría ser una reliquia evolutiva de un código genético temprano y más simple con menos aminoácidos que luego evolucionó para codificar un conjunto más grande de aminoácidos. [69] También podría reflejar propiedades estéricas y químicas que tuvieron otro efecto en el codón durante su evolución. Los aminoácidos con propiedades físicas similares también tienden a tener codones similares, [70] [71] reduciendo los problemas causados por mutaciones puntuales y errores de traducción. [68]
Dado el esquema de codificación de tripletes genéticos no aleatorios, una hipótesis sostenible para el origen del código genético podría abordar múltiples aspectos de la tabla de codones, como la ausencia de codones para D-aminoácidos, patrones de codones secundarios para algunos aminoácidos, confinamiento de sinónimos. posiciones a la tercera posición, el pequeño conjunto de solo 20 aminoácidos (en lugar de un número que se acerca a 64) y la relación de los patrones de codón de parada con los patrones de codificación de aminoácidos. [72]
Tres hipótesis principales abordan el origen del código genético. Muchos modelos pertenecen a uno de ellos o a un híbrido: [73]
- Congelación aleatoria: el código genético fue creado al azar. Por ejemplo, las ribozimas similares a ARNtpueden tener afinidades diferentes para los aminoácidos, con los codones que emergen de otra parte de la ribozima que exhibió variabilidad aleatoria. Una vez que se codificaron suficientes péptidos , cualquier cambio aleatorio importante en el código genético habría sido letal; Por lo tanto, se convirtió en "congelado". [74]
- Afinidad estereoquímica: el código genético es el resultado de una alta afinidad entre cada aminoácido y su codón o anti-codón; la última opción implica que las moléculas pre-tRNA coincidieron con sus aminoácidos correspondientes por esta afinidad. Más tarde, durante la evolución, este emparejamiento se reemplazó gradualmente con el emparejamiento por aminoacil-ARNt sintetasas. [72] [75] [76]
- Optimalidad: el código genético continuó evolucionando después de su creación inicial, de modo que el código actual maximiza alguna función de aptitud , por lo general algún tipo de minimización de errores. [72] [73]
Las hipótesis han abordado una variedad de escenarios: [77]
- Los principios químicos gobiernan la interacción específica del ARN con los aminoácidos. Los experimentos con aptámeros mostraron que algunos aminoácidos tienen una afinidad química selectiva por sus codones. [78] Los experimentos mostraron que de los 8 aminoácidos analizados, 6 muestran alguna asociación ARN triplete-aminoácido. [66] [76]
- Expansión biosintética. El código genético creció de un código anterior más simple a través de un proceso de "expansión biosintética". La vida primordial "descubrió" nuevos aminoácidos (por ejemplo, como subproductos del metabolismo ) y luego incorporó algunos de estos en la maquinaria de codificación genética. [79] Aunque se ha encontrado mucha evidencia circunstancial que sugiere que se usaron menos tipos de aminoácidos en el pasado, [80] hipótesis precisas y detalladas sobre qué aminoácidos ingresaron al código en qué orden son controvertidos. [81] [82]
- La selección natural ha llevado a asignaciones de codones del código genético que minimizan los efectos de las mutaciones . [83] Una hipótesis reciente [84] sugiere que el código de triplete se derivó de códigos que se utilizaron más tiempo que los codones de triplete (como los codones de cuadruplete). Una decodificación más larga que la tripleta aumentaría la redundancia de codones y sería más resistente a los errores. Esta característica podría permitir una descodificación precisa de maquinaria compleja de traslación, como el ribosoma , como antes de que las células comenzaran a producir ribosomas.
- Canales de información: los enfoques de la teoría de la información modelan el proceso de traducir el código genético en los aminoácidos correspondientes como un canal de información propenso a errores. [85] El ruido inherente (es decir, el error) en el canal plantea al organismo una pregunta fundamental: ¿cómo se puede construir un código genético para resistir el ruido [86] mientras se traduce información de manera precisa y eficiente? Estos modelos de "distorsión de frecuencia" [87] sugieren que el código genético se originó como resultado de la interacción de las tres fuerzas evolutivas en conflicto: las necesidades de diversos aminoácidos, [88] para la tolerancia a errores [83]y por un costo mínimo de recursos. El código surge en una transición cuando el mapeo de los codones a los aminoácidos se convierte en no aleatorio. La aparición del código se rige por la topología definida por los errores probables y está relacionada con el problema de coloreado del mapa . [89]
- Teoría de juegos: los modelos basados en juegos de señalización combinan elementos de la teoría de juegos, la selección natural y los canales de información. Tales modelos se han utilizado para sugerir que los primeros polipéptidos probablemente eran cortos y tenían una función no enzimática. Los modelos de teoría de juegos sugirieron que la organización de cadenas de ARN en las células puede haber sido necesaria para evitar el uso "engañoso" del código genético, es decir, evitar que el antiguo equivalente de virus abrume al mundo del ARN. [90]
- Codones de parada: los codones para paradas de traducción también son un aspecto interesante del problema del origen del código genético. Como ejemplo para abordar la evolución del codón de parada, se ha sugerido que los codones de parada son tales que es más probable que terminen la traducción en una etapatemprana en el caso de un error de cambio de marco . [91] En contraste, algunos modelos moleculares estereoquímicos explican el origen de los codones de parada como "no asignables".





No hay comentarios:
Publicar un comentario