En biología molecular , la complementariedad describe una relación entre dos estructuras, cada una siguiendo el principio de bloqueo y clave. En la naturaleza, la complementariedad es el principio básico de la replicación y transcripción del ADN, ya que es una propiedad compartida entre dos secuencias de ADN o ARN , de manera que cuando se alinean antiparalelas entre sí, las bases de nucleótidos en cada posición en las secuencias serán complementarias , mucho Como mirar en el espejo y ver el revés de las cosas. Este emparejamiento de base complementario permite a las células copiar información de una generación a otra e incluso encontrar y reparar daños A la información almacenada en las secuencias.
El grado de complementariedad entre dos cadenas de ácido nucleico puede variar, desde la complementariedad completa (cada nucleótido se encuentra frente a su opuesto) hasta la no complementariedad (cada nucleótido no se encuentra frente a su opuesto) y determina la estabilidad de las secuencias para estar juntas. Además, varias funciones de reparación del ADN, así como funciones reguladoras, se basan en la complementariedad del par de bases. En biotecnología, el principio de complementariedad de pares de bases permite la generación de híbridos de ADN entre el ARN y el ADN, y abre la puerta a herramientas modernas como las bibliotecas de ADNc . Si bien la mayor complementariedad se ve entre dos cadenas separadas de ADN o ARN, también es posible que una secuencia tenga una complementariedad interna que resulte en la secuenciaAtando a sí mismo en una configuración plegada.
DNA y la base de ARN par complementariedad [ editar ]
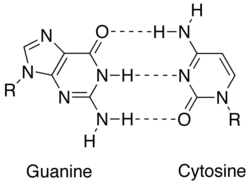
La complementariedad se logra mediante distintas interacciones entre nucleobases : adenina , timina ( uracilo en el ARN ), guanina y citosina . La adenina y la guanina son purinas , mientras que la timina, la citosina y el uracilo son pirimidinas . Las purinas son más grandes que las pirimidinas. Ambos tipos de moléculas se complementan entre sí y solo pueden emparejarse con el tipo opuesto de nucleobase. En el ácido nucleico, las nucleobases se mantienen unidas por enlaces de hidrógeno, que solo funciona de manera eficiente entre la adenina y la timina y entre la guanina y la citosina. El complemento básico A = T comparte dos enlaces de hidrógeno, mientras que el par de bases G≡C tiene tres enlaces de hidrógeno. Todas las demás configuraciones entre nucleobases dificultarían la formación de doble hélice. Las hebras de ADN están orientadas en direcciones opuestas, se dice que son antiparalelas . [1]
| Ácido nucleico | Nucleobases | Complemento de base |
| ADN | adenina (A), timina (T), guanina (G), citosina (C) | A = T, G≡C |
| ARN | adenina (A), uracilo (U), guanina (G), citosina (C) | A = U, G≡C |
Una cadena complementaria de ADN o ARN puede construirse basándose en la complementariedad de las nucleobases. [2] Cada par de bases, A = T vs. G≡C, ocupa aproximadamente el mismo espacio, lo que permite una formación de doble hélice de ADN torcido sin distorsiones espaciales. El enlace de hidrógeno entre las nucleobases también estabiliza la doble hélice del ADN. [3]
La complementariedad de las cadenas de ADN en una doble hélice permite utilizar una cadena como plantilla para construir la otra. Este principio juega un papel importante en la replicación del ADN , ya que establece la base de la herencia al explicar cómo la información genética puede transmitirse a la próxima generación. La complementariedad también se utiliza en la transcripción del ADN , que genera una cadena de ARN a partir de una plantilla de ADN. [4]
Los mecanismos de reparación del ADN , como la lectura de la prueba, se basan en la complementariedad y permiten la corrección de errores durante la replicación del ADN mediante la eliminación de nucleobases no coincidentes. [1]
Las cadenas de ácidos nucleicos también pueden formar híbridos en los que el ADN monocatenario puede recocerse fácilmente con ADN o ARN complementario. Este principio es la base de las técnicas de laboratorio comúnmente realizadas, como la reacción en cadena de la polimerasa , la PCR. [1]
Dos cadenas de secuencias complementarias se denominan sentido y antisentido . La cadena de sentido es, en general, la secuencia transcrita de ADN o el ARN que se generó en la transcripción. Mientras que la hebra anti-sentido es la hebra que es complementaria a la secuencia sensorial.
Auto-complementariedad y bucles de horquilla [ editar ]
La auto-complementariedad se refiere al hecho de que una secuencia de ADN o ARN puede plegarse sobre sí misma, creando una estructura similar a doble hebra. Dependiendo de qué tan cerca estén las partes de la secuencia que son auto-complementarias, la hebra puede formar bucles, uniones, protuberancias o bucles internos. [1] Es más probable que el ARN forme este tipo de estructuras debido a la unión de pares de bases no vista en el ADN, como la unión de guanina con uracilo. [1]
Funciones regulatorias [ editar ]
Se puede encontrar complementariedad entre los estiramientos cortos de ácido nucleico y una región de codificación o un gen transcrito, y los resultados en el emparejamiento de bases. Estas secuencias cortas de ácido nucleico se encuentran comúnmente en la naturaleza y tienen funciones reguladoras, como el silenciamiento de genes. [1]
Transcripciones antisentido [ editar ]
Las transcripciones antisentido son tramos de ARNm no codificante que son complementarios a la secuencia codificante. [5] Los estudios de genoma amplio han demostrado que las transcripciones antisentido de ARN ocurren comúnmente dentro de la naturaleza. En general, se cree que aumentan el potencial de codificación del código genético y agregan una capa general de complejidad a la regulación de genes. Hasta el momento, se sabe que el 40% del genoma humano se transcribe en ambas direcciones, lo que subraya el significado potencial de la transcripción inversa. [6] Se ha sugerido que las regiones complementarias entre transcripciones sentido y antisentido permitirían la generación de híbridos de ARN de doble cadena, que pueden desempeñar un papel importante en la regulación de genes. Por ejemplo, el ARNm del factor 1α inducido por hipoxia yEl ARNm de β-secretasa se transcribe bidireccionalmente, y se ha demostrado que la transcripción antisentido actúa como un estabilizador para el guión de sentido. [7]
miRNAs y siRNAs [ editar ]
Los miRNAs , microRNA, son secuencias cortas de RNA que son complementarias a las regiones de un gen transcrito y tienen funciones reguladoras. Las investigaciones actuales indican que los miRNA circulantes pueden utilizarse como nuevos biomarcadores, por lo tanto, muestran evidencia prometedora para ser utilizados en el diagnóstico de enfermedades. [8] Los MiRNA se forman a partir de secuencias más largas de ARN que se liberan por una enzima Dicer a partir de una secuencia de ARN que proviene de un gen regulador. Estas cadenas cortas se unen a un complejo RISC. Se combinan con secuencias en la región corriente arriba de un gen transcrito debido a su complementariedad para actuar como un silenciador para el gen de tres maneras. Una es evitar que el ribosoma se una e iniciar la traducción. Dos es degradando el ARNm al que se ha unido el complejo. Y tres es proporcionar una nueva secuencia de ARN de doble cadena (ARNds) sobre la que Dicer puede actuar para crear más miARN para encontrar y degradar más copias del gen. Los ARN pequeños de interferencia (ARNsi) tienen una función similar a la de los ARNip; provienen de otras fuentes de ARN, pero tienen un propósito similar al de los miARN. [1] Debido a su corta duración, las reglas de complementariedad significa que aún pueden ser muy discriminatorios en sus objetivos de elección. Dado que hay cuatro opciones para cada base en la cadena y una longitud de 20 pb a 22 pb para un mi / siRNA, eso lleva a más de1 × 10 12 combinacionesposibles . Dado que el genoma humano tiene una longitud de ~ 3,1 mil millones de bases, [9] esto significa que cada miRNA solo debe encontrar una coincidencia una vez en todo el genoma humano por accidente.
Besos horquillas [ editar ]
Las horquillas para besar se forman cuando una sola hebra de ácido nucleico se complementa con sí misma creando bucles de ARN en forma de horquilla. [10] Cuando dos horquillas entran en contacto entre sí in vivo , las bases complementarias de las dos cadenas se forman y comienzan a desenrollar las horquillas hasta que se forma un complejo de ARN de doble cadena (ARNbc) o el complejo se desenrolla de nuevo a dos filamentos debido a desajustes en las horquillas. La estructura secundaria de la horquilla antes de besarla permite una estructura estable con un cambio de energía relativamente fijo. [11]El propósito de estas estructuras es un equilibrio de la estabilidad del bucle de la horquilla frente a la fuerza de unión con una hebra complementaria. Demasiado fuerte de un enlace inicial a una mala ubicación y las hebras no se desenrollarán lo suficientemente rápido. Demasiado débil de una unión inicial y las hebras nunca formarán el complejo deseado. Estas estructuras de horquilla permiten la exposición de bases suficientes para proporcionar un control suficientemente fuerte en el enlace inicial y un enlace interno suficientemente débil para permitir el despliegue una vez que se haya encontrado una coincidencia favorable. [11]
Bioinformática [ editar ]
La complementariedad permite que la información encontrada en el ADN o ARN se almacene en una sola hebra. La cadena complementaria se puede determinar a partir de la plantilla y viceversa como en las bibliotecas de ADNc. Esto también permite el análisis, como comparar las secuencias de dos especies diferentes. Se han desarrollado abreviaturas para escribir secuencias cuando hay desajustes (códigos de ambigüedad) o para acelerar la forma de leer la secuencia opuesta en el complemento (ambigramas).
Biblioteca de ADNc [ editar ]
Una biblioteca de ADNc es una colección de genes de ADN expresados que se consideran una herramienta de referencia útil en los procesos de identificación y clonación de genes. Las bibliotecas de ADNc se construyen a partir de ARNm utilizando transcriptasa inversa (RT) de ADN polimerasa dependiente de ARN, que transcribe una plantilla de ARNm en ADN. Por lo tanto, una biblioteca de cDNA solo puede contener inserciones que están destinadas a transcribirse en mRNA. Este proceso se basa en el principio de complementariedad ADN / ARN. El producto final de las bibliotecas es ADN de doble cadena, que puede insertarse en plásmidos. Por lo tanto, las bibliotecas de cDNA son una herramienta poderosa en la investigación moderna. [1] [12]
Códigos de ambigüedad [ editar ]
Al escribir secuencias para la biología sistemática , puede ser necesario tener códigos IUPAC que signifiquen "cualquiera de los dos" o "cualquiera de los tres". El código IUPAC R (cualquier purina ) es complementario a Y (cualquier pirimidina ) y M (amino) a K (ceto). W (débil) y S (fuerte) generalmente no se intercambian [13], pero algunas herramientas lo han cambiado en el pasado. [14] W y S denotan "débil" y "fuerte", respectivamente, e indican un número de los enlaces de hidrógeno que un nucleótido usa para emparejarse con su compañero complementario. Un socio usa el mismo número de bonos para hacer un par complementario. [15]
Un código IUPAC que excluye específicamente uno de los tres nucleótidos puede ser complementario a un código IUPAC que excluye el nucleótido complementario. Por ejemplo, V (A, C o G - "no T") puede ser complementario de B (C, G o T - "no A").
| Símbolo [16] | Descripción | Bases representadas | ||||
|---|---|---|---|---|---|---|
| UNA | un denine | UNA | 1 | |||
| do | c ytosina | do | ||||
| sol | g uanine | sol | ||||
| T | t hymine | T | ||||
| U | u racil | U | ||||
| W | w EAK | UNA | T | 2 | ||
| S | s trong | do | sol | |||
| METRO | a m ino | UNA | do | |||
| K | k eto | sol | T | |||
| R | pu r ine | UNA | sol | |||
| Y | p y rimidina | do | T | |||
| segundo | no A ( B viene después de A) | do | sol | T | 3 | |
| re | no C ( D viene después de C) | UNA | sol | T | ||
| H | no G ( H viene después de G) | UNA | do | T | ||
| V | no T ( V viene después de T y U) | UNA | do | sol | ||
| N o - | a n y base (no es un hueco) | UNA | do | sol | T | 4 |
Ambigramas [ editar ]
Se pueden usar caracteres específicos para crear una notación de ácido nucleico ( ambigráfica ) adecuada para las bases complementarias (es decir, guanina = b , citosina = q , adenina = n y timina = u ), lo que hace que sea posible complementar secuencias de ADN completas simplemente Girando el texto "al revés". [17] Por ejemplo, con el alfabeto anterior, buqn (GTCA) se leería como ubnq (TGAC, complemento inverso) si se voltea al revés.
- qqubqnnquunbbqnbb
- bbnqbuubnnuqqbuqq
Las notaciones ambigráficas visualizan fácilmente estiramientos de ácidos nucleicos complementarios, como las secuencias palindrómicas. [18] Esta función se mejora cuando se utilizan fuentes o símbolos personalizados en lugar de caracteres ASCII ordinarios o incluso Unicode.
En genética , el ADN complementario ( ADNc ) es ADN sintetizado a partir de una plantilla de ARN monocatenario (por ejemplo, ARN mensajero ( ARNm ) o microARN ) en una reacción catalizada por la enzima transcriptasa inversa . El ADNc se usa a menudo para clonar genes eucariotas en procariotas . Cuando los científicos desean expresar una proteína específica en una célula que normalmente no expresa esa proteína (es decir, una expresión heteróloga ), transferirán el ADNc que codifica la proteína a la célula receptora. ADNc también se produce naturalmente porRetrovirus(como VIH-1 , VIH-2 , virus de inmunodeficiencia de simio , etc.) y luego se integra en el genoma del huésped, donde crea un provirus . [1]
El término ADNc también se usa, típicamente en un contexto de bioinformática , para referirse a una secuencia de transcripción de ARNm, expresada como bases de ADN (GCAT) en lugar de bases de ARN (GCAU).
Síntesis [ editar ]
Aunque existen varios métodos para hacerlo, el ADNc se sintetiza a menudo a partir de ARNm maduro (completamente empalmado) utilizando la enzima transcriptasa inversa . Esta enzima, que ocurre naturalmente en los retrovirus, opera en una sola cadena de ARNm, generando su ADN complementario basado en el emparejamiento de pares de bases de ARN (A, U, G y C) a sus complementos de ADN (T, A, C y G). , respectivamente).
Para obtener cDNA eucariótico cuyos intrones se han eliminado:
- Una célula eucariota transcribe el ADN (de los genes) a ARN ( pre-ARNm ).
- La misma célula procesa las hebras de pre-ARNm eliminando los intrones y agregando una cola de poli-Ay un tapón de metil-guanina 5 ' (esto se conoce como modificación post-transcripcional )
- Esta mezcla de cadenas de ARNm maduras se extrae de la célula. La cola poli-A del ARNm postranscripcional se puede aprovechar con cuentas de oligo (dT) en un ensayo de cromatografía de afinidad .
- Un cebador oligonucleotídico poli- T se hibrida en la cola poli-A de la plantilla de ARNm maduro, o se pueden agregar cebadores hexaméricos aleatorios que contienen cada posible hebra de 6 bases de ADN y por lo tanto pueden hibridar en cualquier lugar en el ARN (la transcriptasa inversa lo requiere Segmento de doble cadena como cebador para comenzar su operación.)
- Se agrega la transcriptasa inversa , junto con los trifosfatos de desoxinucleótidos ( A , T , G , C ). Esto sintetiza una cadena complementaria de ADN hibridado con la cadena de ARNm original.
- Para sintetizar una cadena de ADN adicional, tradicionalmente se digiere el ARN de la cadena híbrida, utilizando una enzima como la ARNasa H , o mediante el método de digestión alcalina.
- Después de la digestión del ARN, queda un ADN monocatenario (ADNss) y debido a que los ácidos nucleicos monocatenarios son hidrófobos, tiende a girar alrededor de sí mismo. Es probable que el ssDNA forme un bucle en el extremo 3 '.
- Desde el bucle de la horquilla, una ADN polimerasa puede usarla como cebador para transcribir una secuencia complementaria para el ADNc de ss.
- Ahora, debería quedarse con un cDNA de doble cadena con una secuencia idéntica al mRNA de interés.
Aplicaciones [ editar ]
El ADN complementario se usa a menudo en la clonación de genes o como sondas genéticas o en la creación de una biblioteca de ADNc . Cuando los científicos transfieren un gen de una célula a otra para expresar el nuevo material genético como una proteína en la célula receptora, el ADNc se agregará al receptor (en lugar de a todo el gen), porque el ADN de un gen completo puede incluir ADN que no codifica para la proteína o que interrumpe la secuencia de codificación de la proteína (por ejemplo, intrones ). Las secuencias parciales de ADNc a menudo se obtienen como etiquetas de secuencia expresadas (EST) .
Con la amplificación de las secuencias de ADN a través de la reacción en cadena de la polimerasa (PCR) ahora común, uno típicamente llevará a cabo la transcripción inversa como un paso inicial, seguido de la PCR para obtener una secuencia exacta de ADNc para la expresión intracelular. Esto se logra diseñando cebadores de ADN específicos de secuencia que hibridan con los extremos 5 'y 3' de una región de ADNc que codifica una proteína. Una vez amplificada, la secuencia se puede cortar en cada extremo con nucleasas e insertarse en una de las muchas secuencias de ADN circular pequeñas conocidas como vectores de expresión. Dichos vectores permiten la auto-replicación, dentro de las células, y potencialmente la integración en el ADN del huésped. Por lo general, también contienen un promotor fuerte para impulsar la transcripción del ADNc objetivo en el ARNm, que luego se traduce en proteína.
El 13 de junio de 2013, la Corte Suprema de los Estados Unidos dictaminó en el caso de Association for Molecular Pathology v. Myriad Genetics que, si bien los genes humanos que ocurren naturalmente no pueden ser patentados , el ADNc es una patente elegible porque no ocurre de manera natural. [2]
Virus [ editar ]
Algunos virus también utilizan el ADNc para convertir su ARN viral en ARNm (ARN viral → ADNc → ARNm). El ARNm se usa para hacer proteínas virales para controlar la célula huésped.
Un ejemplo de este primer paso del ADN viral al ADNc puede verse en el ciclo de infección del VIH. Aquí, la membrana de la célula huésped se adhiere a la envoltura lipídica del virus, lo que permite que la cápside viral con dos copias del ARN del genoma viral ingrese al huésped. La copia de cDNA se realiza a través de la transcripción inversa del RNA viral, un proceso facilitado por la chaperona CypA y una cápside viral asociada a la transcriptasa inversa.







No hay comentarios:
Publicar un comentario