(Redirigido desde el cuadro CCAAT )
En biología molecular , un cuadro CCAAT (también a veces abreviado un cuadro CAAT o cuadro CAT ) es un patrón distinto de nucleótidos con secuencia consenso GGCCAATCT que se produce en sentido ascendente en 60 a 100 bases al sitio de transcripción inicial . El cuadro CAAT señala el sitio de unión para el factor de transcripción de ARN , y suele ir acompañado de una secuencia de consenso conservada . Es un ADN invariante.secuencia en aproximadamente menos 70 pares de bases desde el origen de la transcripción en muchos promotores eucarióticos. Los genes que tienen este elemento parecen requerir que el gen se transcriba en cantidades suficientes. Con frecuencia está ausente de los genes que codifican proteínas utilizadas en prácticamente todas las células. Este cuadro junto con el cuadro de GC es conocido por los factores de transcripción generales vinculantes. Ambas secuencias de consenso pertenecen al promotor regulador . La expresión génica completa se produce cuando las proteínas activadoras de la transcripción se unen a cada módulo dentro del promotor regulador. La unión específica de proteínas es necesaria para la activación de la caja CCAAT. Estas proteínas se conocen como proteínas de unión a caja CCAAT / factores de unión a caja CCAAT.
Un cuadro CCAAT es una característica que se encuentra con frecuencia antes de las regiones de codificación de eucariotas, pero no se encuentra en procariotas.
Secuencia de consenso [ editar ]
En la dirección de la transcripción de la línea plantilla, la secuencia de consenso , o el orden calculado de los residuos más frecuentes, para el cuadro CAAT fue 3'-TG ATTGG (T / C) (T / C) (A / G) - 5 '. El uso de paréntesis denota que cualquiera de las dos bases está presente, pero no se especifica en cuanto a sus frecuencias relativas. Por ejemplo, "(T / C)" significaría que la timina o la citosina se seleccionan preferentemente para. [3]Dentro de metazoa(reino animal), el factor de unión al núcleo (CBF) -DNA complejo conserva un alto grado de conservación dentro del motivo de unión CCAAT, así como las secuencias que flanquean este motivo pentamérica. El motivo CCAAT en plantas (la espinaca se usó en un experimento) difiere ligeramente del metazoa en que en realidad es un motivo de unión a CAAT; el promotor carece de uno de los dos residuos C del motivo pentamericano, y la adición artificial de la segunda C no tiene efectos significativos sobre la actividad de unión. Algunas secuencias carecen completamente de la caja CAAT. En segundo lugar, los nucleótidos circundantes en las plantas no coinciden con la secuencia de consenso anterior determinada por Bi et al. [4]
Promotor del núcleo [ editar ]
La caja CAAT es lo que se conoce como un promotor central, también conocido como el promotor basal o simplemente el promotor , es una región de ADN que inicia la transcripción de un gen particular. Esta región, en particular para el cuadro CAAT, está ubicada a unas 60–100 bases en sentido ascendente (hacia el extremo 5 '), pero a no menos de 27 pares de bases de distancia, desde el sitio de transcripción inicial o un gen eucariota en el que se encuentra un complejo de Los factores de transcripción se unen a la ARN polimerasa II antes del inicio de la transcripción. [5] [6] Es esencial para la transcripción que estos factores de unión centrales (también denominados factor nuclear Y o NF-Y) puedan unirse al motivo CCAAT. Los experimentos en muchos laboratorios han demostrado que las mutaciones en el motivo CCAAT que causan una pérdida de la unión a CBF también disminuyen la actividad transcripcional en estos promotores, lo que sugiere que los complejos CBF-CCAAT son esenciales para una actividad transcripcional óptima. [3]
Encuadernación [ editar ]
En un experimento realizado con factores de unión al núcleo (CBF) y complejos de ADN, los investigadores pudieron determinar las secuencias preferenciales del promotor en una región sobre e inmediatamente adyacente a la caja CAAT, y dos regiones a cada lado de la caja CAAT. Al utilizar el proceso de selección de unión aleatoria mediado por PCR , los investigadores pudieron demostrar que la secuencia "3 '- (T / C) G ATTGG (T / C) (T / C) (A / G) - 5'" inmediatamente flanqueando la región ATTGG (CCAAT en la cadena complementaria) se seleccionó preferentemente en la cadena codificante (opuesta a la cadena plantilla). [3] [7] [8] Esto se demostró utilizando una secuencia de oligonucleótidos (R1) que contenía 27 nucleótidos aleatorios, flanqueados por una secuencia de 20 nucleótidos definida en cada lado. Si bien no se seleccionó un solo nucleótido en cada clon a cada lado del motivo ATTGG (CCAAT en la cadena complementaria), hubo varios nucleótidos en posiciones seleccionadas con alta frecuencia. Lo más notable de la secuencia anterior fue el residuo G hacia el extremo 5 'del ATTGG. Los otros residuos enumerados también fueron notables, pero hay una división entre dos residuos. Este mismo experimento también produjo la misma secuencia que se muestra arriba cuando se usa un oligonucleótido diferente (R2) que contenía un núcleo de ATTGG y flanqueado por 12 5 'de nucleótidos aleatorios y 10 3' de nucleótidos aleatorios. Ambas secuencias son muy similares y se confirman en múltiples experimentos.[3] En otro experimento realizado con el principal promotor tardío (MLP) de adenovirus de una variedad de especies hospedadoras, se demostró que la mutación de la caja CAAT y la secuencia CCAAT, que se cree desempeña un papel fundamental en la (MLP). ) del subgrupo C adenovirus humanos, en especies con una secuencia CAAT deficiente. El inicio de la transcripción en las especies de MLP mutantes se redujo significativamente en comparación con el tipo salvaje o especie en la que había un mutante CAAT. El hecho de no restaurar los adenovirus normalmente funcionales, exhibidos por una caja CAAT, es consistente con la idea de que la caja CAAT juega un papel vital en la MLP del adenovirus y se prefiere sobre otros elementos transcripcionales. [9]
CCAAT en plantas [ editar ]
Estos factores de unión al núcleo, o factores nucleares (NF-Y), están compuestos por tres subunidades: NF-YA, NF-YB y NF-YC. Mientras que en los animales cada subunidad NF-Y está codificada por un solo gen, ha habido una diversificación en las plantas tanto en su estructura como en su función. Las familias de NF-Y constan de entre ocho y 39 miembros por subunidad. Una gran razón para esta diversificación se debe a las duplicaciones genéticas y las duplicaciones en tándem, que han contribuido a los tamaños familiares más grandes de NF-Y en comparación con los factores nucleares animales codificados de forma única. [10] Cada subunidad contiene una parte conservada evolutivamente: el C-terminal de NF-YA, la parte central de NF-YB y el N-terminalde NF-YC, más del 70% de estos a través de especies permanece conservado. Sin embargo, las regiones vecinas generalmente no se conservan. [6]
Subunidad NF-YA [ editar ]
La familia NF-YA codifica factores de transcripción que son de longitud variable (entre 207 y 347 aminoácidos para M. truncatula ). Las proteínas NF-YA se caracterizan generalmente por dos dominios que están fuertemente conservados en todos los eucariotas superiores investigados hasta la fecha. El primer dominio (A1) contiene 20 aminoácidos que forman una hélice alfaEso parece significativo en sus interacciones con NF-YB y NF-YC. El segundo dominio (A2) es adyacente al dominio A1 por una secuencia enlazadora conservada, es una secuencia de 21 aminoácidos vitales en el ADN específico a la unión de la caja CCAAT. Los dominios A1 y A2 se conservan hacia el extremo C de los mamíferos, pero ocupan una región más central en las subunidades NF-YA de la planta. En las plantas, la subunidad NF-YA ha evolucionado para regular el desarrollo de un órgano radicular facultativo que solo está presente en las plantas leguminosas y se ha demostrado que se expresa en el tejido radicular. Se demostró que tiene propiedades similares a la sequía, que se regulan al alza durante el estrés por sequía en las raíces y hojas de Arabidopsis. Los mutantes NF-YA han demostrado una pérdida de función y una hipersensibilidad a condiciones similares a la sequía, y en contraste, la sobreexpresión de NF-YA ha dado como resultado una resistencia a la sequía. [10]
Subunidad NF-YB [ editar ]
La familia NF-YB es, similar a la subunidad NF-YA, de longitud variable, sin embargo, en promedio es mucho más pequeña que la subunidad NF-YA (90-240 aminoácidos en "M. truncatula"). Se han caracterizado con una estructura y una composición de aminoácidos similar al motivo de plegamiento de histonas (HFM). Se compone de tres hélices alfa separadas por dos dominios de hebra beta. Similar a NF-YA, se ha demostrado que NF-YB también mejora la resistencia a la sequía cuando se sobreexpresa y también la promoción de la floración en Arabidopsis . [10]
Subunidad NF-YC [ editar ]
Las proteínas NF-YC tienen un tamaño intermedio entre las proteínas NF-YA y NF-YB (117–292 aminoácidos en M. truncatula ) y también contienen la HFM que prevalece en las proteínas NF-YB. También se ha demostrado que está involucrado en el tiempo de floración en ciertas plantas (la sobreexpresión conduce a una floración más temprana) donde su influencia está potencialmente regulada por la unión de la proteína CONSTANS (CO) a la subunidad NF-YC. [10]
Complejos NF-Y [ editar ]
Debido al cambio evolutivo en los genes que codifican NF-Y en las plantas, posteriormente tienen una gran variedad de complejos triméricos potenciales. Por ejemplo, en Arabidopsis., Se han identificado 36 subunidades del factor de transcripción NF-Y (incluidas 10 subunidades NF-YA, 13 NF-YB y 13 NF-YC) y que teóricamente podrían formar 1690 complejos únicos (que contienen una de cada tipo de subunidad). Este número, por supuesto, es más alto que lo que realmente sucede, ya que algunas subunidades tienen patrones de enlace específicos. Los análisis funcionales de los genes que codifican para NF-Y en plantas han demostrado que, como resultado de su diversificación evolutiva en relación con sus homólogos animales, han adquirido diversas funciones específicas, como el desarrollo de embriones, el control del tiempo de floración, el estrés ER, el estrés por sequía y los nódulos. y desarrollo de la raíz. También se debe tener en cuenta que esto puede ser solo una pequeña parte de sus capacidades,[10]
Proteínas de unión al potenciador de CCAAT (C / EBPs) [ editar ]
Otro aspecto del motivo de unión CCAAT es las proteínas de unión CCAAT / potenciador (C / EBPs). Son un grupo de factores de transcripción de 6 miembros (α-ζ), que están altamente conservados y se unen al motivo CCAAT. Si bien la investigación sobre estas proteínas de unión es relativamente reciente, se ha demostrado que su función desempeña funciones vitales en la proliferación y diferenciación celular, el metabolismo , la inflamación y la inmunidad en varias células, pero específicamente en hepatocitos , adipocitos y células hematopoyéticas . [11] Por ejemplo, en adipocitos, esto se ha demostrado en una variedad de experimentos con ratones: la expresión ectópica de estos C / EBPs (C / EBPα y C / EBPβ) fue capaz de iniciar los programas de diferenciación de la célula, incluso en ausencia de Hormonas adipogénicas , o la diferenciación de los preadipocitos a adipocitos (o células grasas). Además, un exceso de estos C / EBPs (específicamente, C / EBPδ) causa una respuesta acelerada. Y además, en células que carecen de C / EBP o en ratones deficientes en C / EBP, ambos son incapaces de sufrir adipogénesis. Esto hace que los ratones mueran de hipoglucemia o la acumulación reducida de lípidos en el tejido adiposo. [12] Los C / EBPs siguen un dominio general de cremallera de leucina básica (bZIP) en el término Cy son capaces de formar dímeros con otros C / EBPs u otros factores de transcripción. Esta dimerización permite que los C / EBP se unan específicamente al ADN a través de una secuencia palindrómica en el surco principal del ADN. Están regulados a través de diversos medios, incluidas hormonas , mitógenos , citoquinas , nutrientes y otros factores diversos.
En genética , un centimorgan (abreviado cM ) o unidad de mapa ( mu ) es una unidad para medir el enlace genético . Se define como la distancia entre las posiciones de los cromosomas (también denominados loci o marcadores ) para los cuales el número promedio esperado de cruces cromosómicos intermedios en una sola generación es 0.01. A menudo se utiliza para inferir la distancia a lo largo de un cromosoma. Sin embargo, no es una verdadera distancia física.
Relación con la distancia física [ editar ]
El número de pares de bases a los que corresponde varía ampliamente a lo largo del genoma (las diferentes regiones de un cromosoma tienen diferentes inclinaciones hacia el cruce) y también depende de si la meiosis en la que se produce el cruce es parte de la ovogénesis (formación de gametos femeninos) o espermatogénesis (formación de gametos masculinos).
Un centimorgan corresponde a aproximadamente 1 millón de pares de bases en humanos en promedio. [1] [2] La relación es áspera ya que la distancia cromosómica física correspondiente a un centimorgan varía de un lugar a otro en el genoma, y también varía entre hombres y mujeres, ya que la recombinación durante la formación de gametos en mujeres es significativamente más frecuente que en hombres . Morton et al. calculó que el genoma femenino tiene una longitud de 4782 centimorgans, mientras que el genoma masculino solo tiene una longitud de 2809 centimorgans. [3] Plasmodium falciparum tiene una distancia de recombinación promedio de ~ 15 kb por centímetro de órgano: los marcadores separados por 15 kb de ADN (15,000 nucleótidos) tienen una tasa esperada de cruces cromosómicos de 0.01 por generación. Tenga en cuenta que no syntenic Los genes (genes que residen en diferentes cromosomas) están intrínsecamente desvinculados, y las distancias cM no tienen significado entre ellos.
Relación con la probabilidad de recombinación [ editar ]
Debido a que la recombinación genética entre dos marcadores se detecta solo si hay un número impar de cruces cromosómicos entre los dos marcadores, la distancia en centimorgans no corresponde exactamente a la probabilidad de recombinación genética. Suponiendo que la función del mapa de Haldane , en la que el número de cruces cromosómicos se distribuye de acuerdo con una distribución de Poisson , [4] una distancia genética de d centimorgans conducirá a un número impar de cruces cromosómicos, y por lo tanto una recombinación genética detectable, con probabilidad
donde sinh es la función sinusoidal hiperbólica . La probabilidad de recombinación es aproximadamente d / 100 para valores pequeños de d y se aproxima al 50% a medida que d va al infinito.
La fórmula se puede invertir, dando la distancia en centimorgans en función de la probabilidad de recombinación:
Etimología [ editar ]
El centimorgan fue nombrado en honor del genetista Thomas Hunt Morgan por su estudiante Alfred Henry Sturtevant . Sin embargo, su unidad principal, el morgan, rara vez se utiliza hoy en día.
El centrómero es la secuencia de ADN especializada de un cromosomaque une un par de cromátidas hermanas (una díada). [1] Durante la mitosis , las fibras del huso se adhieren al centrómero a través del cinetocoro . [2] Primero se pensó que los centrómeros eran loci genéticosque dirigían el comportamiento de los cromosomas .
La función física del centrómero es actuar como el sitio de ensamblaje de los cinetocoros , una estructura multiproteína altamente compleja que es responsable de los eventos reales de la segregación de cromosomas , es decir, microtúbulos de unión y señalización a la maquinaria del ciclo celular cuando todos los cromosomas han adoptado correctamente accesorios al huso , de modo que es seguro que la división celularproceda a completarse y que las células ingresen en la anafase . [3]
En términos generales, existen dos tipos de centrómeros. Los "centrómeros puntuales" se unen a proteínas específicas que reconocen secuencias de ADN particulares con alta eficiencia. [4] Cualquier pieza de ADN que contenga la secuencia de ADN de centrómero puntual formará típicamente un centrómero si está presente en la especie apropiada. Los centrómeros puntuales mejor caracterizados son los de la levadura en ciernes, Saccharomyces cerevisiae . "Centromeres regionales" es el término acuñado para describir la mayoría de los centrómeros, que normalmente se forman en regiones de secuencia de ADN preferidas, pero que también pueden formarse en otras secuencias de ADN. [4] La señal para la formación de un centrómero regional parece ser epigenética.. La mayoría de los organismos, que van desde la levadura de fisión Schizosaccharomyces pombe hasta los humanos, tienen centrómeros regionales.
Con respecto a la estructura del cromosoma mitótico, los centrómeros representan una región restringida del cromosoma (a menudo conocida como la constricción primaria) donde dos cromátidas hermanas idénticas están más en contacto. Cuando las células entran en la mitosis, las cromátidas hermanas (las dos copias de cada molécula de ADN cromosómica resultante de la replicación del ADN en forma de cromatina) se unen a lo largo de su longitud por la acción del complejo de cohesina . Ahora se cree que este complejo se libera principalmente de los brazos cromosómicos durante la profase, de modo que para cuando los cromosomas se alinean en el plano medio del huso mitótico (también conocido como placa metafase), el último lugar donde están vinculados El uno con el otro está en la cromatina dentro y alrededor del centrómero.
Posiciones [ editar ]
Cada cromosoma tiene dos brazos, etiquetados p (el más corto de los dos) y q (el más largo). Muchos recuerdan que el brazo corto 'p' se llama así por la palabra francesa "petit" que significa 'pequeño', aunque se demostró que esta explicación es apócrifa. [6] Se pueden conectar de forma metacéntrica, submetacéntrica, acrocéntrica o telocéntrica. [7] [8]
| Categorización de los cromosomas según la longitud relativa de los brazos [8] | ||||||
| Posición centrómera | Relación de longitud de brazos | Firmar | Descripción | |||
| Sensual strictomedial | 1.0 - 1.6 | METRO | Metacéntrico | |||
| Región medial | 1.7 | metro | Metacéntrico | |||
| Submedial | 3.0 | sm | Submetacéntrico | |||
| Subterminal | 3.1 - 6.9 | S t | Subtelocéntrico | |||
| Región terminal | 7.0 | t | Acrocéntrico | |||
| Terminal sensu stricto | ∞ | T | Telocéntrico | |||
| Notas | - | Metacéntrico: M + m | Atelocéntrico : M +m + sm + st + t | |||
Metacéntrico [ editar ]
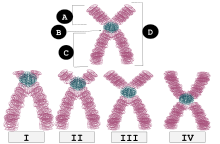
Estos son cromosomas en forma de X, con el centrómero en el medio, de modo que los dos brazos de los cromosomas son casi iguales.
Un cromosoma es metacéntrico si sus dos brazos tienen aproximadamente la misma longitud. En un cariotipo humano normal , cinco cromosomas se consideran metacéntricos: los cromosomas 1, 3, 16, 19 y 20. En algunos casos, un cromosoma metacéntrico se forma por translocación equilibrada: la fusión de dos cromosomas acrocéntricospara formar un cromosoma metacéntrico. [9] [10]
Submetacéntrico [ editar ]
Si la longitud de los brazos es desigual, se dice que el cromosoma es submetacéntrico. Su forma es la forma de L. [11]
Acrocéntrico [ editar ]
Si el brazo p (corto) es tan corto que es difícil de observar, pero aún está presente, entonces el cromosoma es acrocéntrico (el " acro- " en acrocéntrico se refiere a la palabra griega para "pico"). El genoma humano incluye cinco cromosomas acrocéntricos: 13 , 14 , 15 , 21 , 22 . [12] El cromosoma Y también es acrocéntrico. [12]
En un cromosoma acrocéntrico, el brazo p contiene material genético que incluye secuencias repetidas, como las regiones de organización nucleolar, y se puede trasladar sin daño significativo, como en una translocación Robertsonian equilibrada . El genoma del caballo doméstico incluye un cromosoma metacéntrico que es homólogo a dos cromosomas acrocéntricos en el caballo de Przewalski conespecífico pero no domesticado . [13]Esto puede reflejar la fijación de una translocación Robertsoniana equilibrada en caballos domésticos o, a la inversa, la fijación de la fisión de un cromosoma metacéntrico en dos cromosomas acrocéntricos en los caballos de Przewalski. Una situación similar existe entre los genomas humanos y los grandes simios; en este caso, debido a que existen más especies , es evidente que la secuencia evolutiva es una reducción de dos cromosomas acrocéntricos en los grandes simios a un cromosoma metacéntrico en humanos (ver Cariotipo # Aneuploidía ). [11]
Sorprendentemente, las translocaciones dañinas en el contexto de la enfermedad, especialmente las translocaciones desequilibradas en los cánceres de la sangre, implican más frecuentemente cromosomas acrocéntricos que cromosomas no acrocéntricos. [14] Aunque la causa no se conoce, esto probablemente se relaciona con la ubicación física de los cromosomas acrocéntricos dentro del núcleo . Acrocéntricos son generalmente ubicados en y alrededor del nucléolo , por lo que en el centro del núcleo, donde los cromosomas tienden a ser menos densamente poblado de los cromosomas en la periferia nuclear. [15] Consistentemente, las regiones cromosómicas que están menos densamente compactas también son más propensas a las translocaciones cromosómicas en los cánceres. [14]
Telocéntricos [ editar ]
El centrómero de un cromosoma telocéntrico se encuentra en el extremo terminal del cromosoma. Un cromosoma telocéntrico tiene, por lo tanto, un solo brazo. Los telómeros pueden extenderse desde ambos extremos del cromosoma, su forma es similar a la letra "i" durante la anafase. Por ejemplo, el cariotipo de ratón doméstico estándar solo tiene cromosomas telocéntricos. [16] [17] Los humanos no poseen cromosomas telocéntricos.
Subtelocéntricos [ editar ]
Si el centrómero del cromosoma está ubicado más cerca de su extremo que de su centro, puede describirse como subtelocéntrico. [18] [19]
Holocentric [ editar ]
Con los cromosomas holocéntricos , la longitud completa del cromosoma actúa como el centrómero. Se pueden encontrar ejemplos de este tipo de centrómero dispersos en los reinos de plantas y animales [20], siendo el ejemplo más conocido el nematodo Caenorhabditis elegans .
Acéntrico [ editar ]
Si un cromosoma carece de un centrómero, se dice acéntrico . El macronúcleo de los ciliados, por ejemplo, contiene cientos de cromosomas acéntricos. [21] Los eventos de ruptura de cromosomas también pueden generar cromosomas acéntricos o fragmentos acéntricos.
Los cromosomas humanos [ editar ]
| Cromosoma | Posicióncentrómera ( Mbp ) | Categoría | Tamaño delcromosoma (Mbp) | Tamaño decentrómero (Mbp) |
|---|---|---|---|---|
| 1 | 125.0 | metacéntrico | 247.2 | 7.4 |
| 2 | 93.3 | submetacéntrico | 242.8 | 6.3 |
| 3 | 91.0 | metacéntrico | 199.4 | 6.0 |
| 4 | 50.4 | submetacéntrico | 191.3 | - |
| 5 | 48.4 | submetacéntrico | 180.8 | - |
| 6 | 61.0 | submetacéntrico | 170.9 | - |
| 7 | 59.9 | submetacéntrico | 158.8 | - |
| 8 | 45.6 | submetacéntrico | 146.3 | - |
| 9 | 49.0 | submetacéntrico | 140.4 | - |
| 10 | 40.2 | submetacéntrico | 135.4 | - |
| 11 | 53.7 | submetacéntrico | 134.5 | - |
| 12 | 35.8 | submetacéntrico | 132.3 | - |
| 13 | 17.9 | acrocéntrico | 114.1 | - |
| 14 | 17.6 | acrocéntrico | 106.3 | - |
| 15 | 19.0 | acrocéntrico | 100.3 | - |
| dieciséis | 36.6 | metacéntrico | 88.8 | - |
| 17 | 24.0 | submetacéntrico | 78.7 | - |
| 18 | 17.2 | submetacéntrico | 76.1 | - |
| 19 | 26.5 | metacéntrico | 63.8 | - |
| 20 | 27.5 | metacéntrico | 62.4 | - |
| 21 | 13.2 | acrocéntrico | 46.9 | - |
| 22 | 14.7 | acrocéntrico | 49.5 | - |
| X | 60.6 | submetacéntrico | 154.9 | - |
| Y | 12.5 | acrocéntrico | 57.7 | - |
Secuencia [ editar ]
Hay dos tipos de centrómeros. [22] En los centrómeros regionales, las secuencias de ADN contribuyen pero no definen la función. Los centrómeros regionales contienen grandes cantidades de ADN y, a menudo, se empaquetan en heterocromatina . En la mayoría de los eucariotas , la secuencia de ADN del centromero consiste en grandes matrices de ADN repetitivo (por ejemplo , ADN satélite ) donde la secuencia dentro de los elementos de repetición individuales es similar pero no idéntica. En los seres humanos, la unidad de repetición centromérica primaria se llama α-satélite (o alfoide), aunque en esta región se encuentran otros tipos de secuencia. [23]
Los centrómeros de punto son más pequeños y más compactos. Las secuencias de ADN son necesarias y suficientes para especificar la identidad y la función del centrómero en organismos con centrómeros puntuales. En las levaduras en ciernes, la región centromérica es relativamente pequeña (alrededor de 125 pb de ADN) y contiene dos secuencias de ADN altamente conservadas que sirven como sitios de unión para proteínas kinetochore esenciales . [23]
Herencia [ editar ]
Dado que la secuencia de ADN centromérica no es el determinante clave de la identidad centromérica en los metazoos , se cree que la herencia epigenética desempeña un papel importante en la especificación del centrómero. [24] Los cromosomas hijos ensamblarán centrómeros en el mismo lugar que el cromosoma parental, independientemente de la secuencia. Se ha propuesto que la variante de histona H3 CENP-A (proteína A del centrómero) es la marca epigenética del centómero. [25]Se plantea la cuestión de si debe existir todavía alguna forma original en la que se especifica el centrómero, incluso si posteriormente se propaga epigenéticamente. Si el centromero se hereda epigenéticamente de una generación a otra, el problema se remonta al origen de los primeros metazoos.
Estructura [ editar ]
El ADN centromérico está normalmente en un estado de heterocromatina , que es esencial para el reclutamiento del complejo de cohesina que media la cohesión de la cromátida hermana después de la replicación del ADN, así como la coordinación de la separación de la cromátida hermana durante la anafase. En esta cromatina, la histonanormal H3 se reemplaza con una variante específica de centrómero, CENP-A en humanos. [26] Se cree que la presencia de CENP-A es importante para el ensamblaje del cinetocoro en el centrómero. Se ha demostrado que CENP-C se localiza casi exclusivamente en estas regiones de la cromatina asociada a CENP-A. En células humanas, se encuentra que las histonas están más enriquecidas para H4K20me3 y H3K9me3 [27] que son modificaciones heterocromáticas conocidas.
En la levadura Schizosaccharomyces pombe (y probablemente en otros eucariotas), la formación de heterocromatina centromérica está conectada al ARNi . [28] En los nematodos como Caenorhabditis elegans , algunas plantas y los insectos, Lepidoptera y Hemiptera, los cromosomas son "holocéntricos", lo que indica que no hay un sitio primario de uniones de microtúbulos o una constricción primaria, y un ensamblaje cinetocoro difuso a lo largo de toda la longitud del cromosoma.
Aberraciones centroméricas [ editar ]
En casos raros en humanos, los neocentromeros pueden formarse en nuevos sitios en el cromosoma. Actualmente hay más de 90 neocentromeros humanos conocidos identificados en 20 cromosomas diferentes. [29] [30] La formación de un neocentromero se debe combinar con la inactivación del centrómero anterior, ya que los cromosomas con dos centrómeros funcionales ( cromosoma dicéntrico ) provocarán la rotura del cromosoma durante la mitosis. En algunos casos inusuales, se ha observado que los neocentrómeros humanos se forman espontáneamente en cromosomas fragmentados. Algunas de estas nuevas posiciones eran originalmente eucromáticas y carecían completamente de ADN alfa satélite.
Las proteínas centrómeras también son el objetivo autoantigénico de algunos anticuerpos antinucleares , como los anticuerpos anticentrómeros .
La disfunción y la enfermedad [ editar ]
Se sabe que la mala regulación de los centrómeros contribuye a la segregación errónea de los cromosomas, que está fuertemente relacionada con el cáncer y el aborto. En particular, la sobreexpresión de muchos genes centroméricos se ha relacionado con fenotipos malignos de cáncer. La sobreexpresión de estos genes centrómeros puede aumentar la inestabilidad genómica en los cánceres. [31] La inestabilidad genómica elevada, por un lado, se relaciona con fenotipos malignos; por otro lado, hace que las células tumorales sean más vulnerables a terapias adyuvantes específicas, como ciertas quimioterapias y radioterapia. [32] Recientemente se demostró la inestabilidad del ADN repetitivo del centrómero en el cáncer y el envejecimiento. [33]
Etimología y pronunciación [ editar ]
La palabra centrómero ( / s ɛ n t r ə ˌ m ɪər / [34] [35] ) utiliza la combinación de formas de Centro- y -mere , produciendo "parte central", que describen la ubicación del centrómero en el centro del cromosoma.

![\ Pr [{\ text {recombinación}} | {\ text {vínculo de}} d {\ text {cM}}] = \ sum _ {{k = 0}} ^ {{\ infty}} \ Pr [2k +1 {\ text {cruces}} | {\ text {enlace de}} d {\ text {cM}}]](https://wikimedia.org/api/rest_v1/media/math/render/svg/8d2c1615cd0ba257362cdcb70f405eb6cdc821b4)

![d = 50 \ ln \ left ({{\ frac {1} {1-2 \ Pr [{\ text {recombination}}]}}} \ right) \ ,.](https://wikimedia.org/api/rest_v1/media/math/render/svg/d660380310cb8c05941e8d79f1959054450c1f50)

No hay comentarios:
Publicar un comentario