Caudovirales es un orden de virus que comprende a la mayor parte de los bacteriófagos (en torno a un 95%). En el marco del esquema de la Clasificación de Baltimore se integran en el Grupo I, ya que tienen un genoma ADNbicatenario. El tamaño del genoma está comprendido entre 18 y 500 kbp. Las partículas virales tienen una forma distintiva con una cabeza icosaedral que contiene el genoma y conectada a una cola. El orden abarca una amplia gama de virus, muchos de los cuales tienen los mismos o similares genes, mientras que la secuencia de nucleótidos puede variar considerablemente, incluso entre el mismo género. Debido a su característica estructura, se cree que comparten un origen común.
Infección
Al encuentro con una bacteria huésped, la cola del virus se une a los receptores de la superficie celular bacteriana y el virus introduce su ADN en la célula mediante el uso de un mecanismo de inyección. La cola hace un agujero a través de la pared celular y de la membrana plasmática de la bacteria y el genoma pasa por la cola a la célula. Una vez dentro, los genes se expresan mediante la transcripción hecha por la maquinaria huésped, haciendo uso de losribosomas. Típicamente, el genoma se replica haciendo uso de concatémeros.
Ensamblado y maduración
Las proteínas de la cápside se reúnen para formar una protocabeza, en el que se introduce el genoma. Una vez que esto ha ocurrido, la maduración de la protocabeza se realiza por la división de las unidades de la cápside para formar una cabeza icosaedral con simetría 5. Después de la maduración, la cola se añade por una de dos maneras: o bien la cola se construye por separado y se une con la cabeza, o bien se construye directamente sobre la cabeza del fago. La cola consiste en una hélice de proteínas con simetría 6. Después de la maduración de las partículas virales, la célula es lisada usando lisinas, holinas o una combinación de ambas.
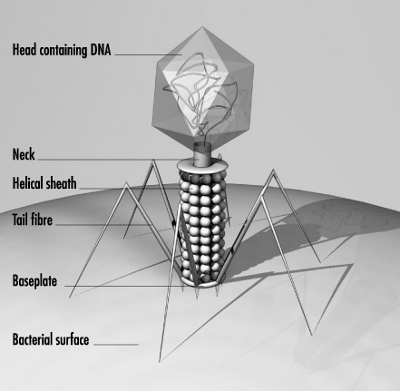

Caulimoviridae es una familia de virus ADN retrotranscritos (o pararetrovirus), esto es, virus que comprenden una etapa de transcripción inversa en su ciclo de replicación. Su genoma consiste en una cadena de ADN bicatenario, por tanto, se incluyen en el Grupo VII de la Clasificación de Baltimore. Todos los virus de esta familia infectan plantas.
Todos los virus de esta familia no presentan envoltura. Las partículas del virus contienen una nucleocápside de dos posibles formas: baciliforme o isométrica. El tipo denucleocápside incorporado en la estructura del virus determina el tamaño de los virus. La nucleocápside de los virus baciliformes mide unos 35-50 nm de diámetro y llega hasta 900 nm de longitud. Los virus de nucleocápside isométrica miden, en promedio, 45-50 nm de diámetro y muestran simetría icosaédrica.
La familia incluye los siguientes géneros:
- Género Badnavirus; especie tipo: Virus del moteado amarillo de Commelina.
- Género Caulimovirus; especie tipo: Virus del mosaico de la coliflor.
- Género Tungrovirus; especie tipo: Virus baciliforme del tungro del arroz.
- Género Soymovirus; especie tipo: Virus del moteado clorótico de la soja.
- Género Cavemovirus; especie tipo: Virus del mosaico veteado de la yuca.
- Género Petuvirus; especie tipo: Virus del veteado claro de la petunia.
El genoma de los virus de esta familia consiste en un único segmento de ADN, ya sea de estructura circular o lineal. El tamaño del genoma es usualmente de 6.000-8.000 pares de bases. Dependiendo del virus, el ADN puede contener un marco abierto de lectura(ORF), como por ejemplo en Pertuvirus, o hasta ocho ORFs como en Soyamovirus. Las proteínas codificadas en el genoma de esta familia incluyen la transcriptasa inversa, proteasas, la nucleocápside y transactivadores. Hay otras proteínas esenciales para la replicación a las que aún no se le ha asignado una función específica.
La replicación se lleva a cabo tanto en el citoplasma como en el núcleo de la célula huésped. En primer lugar, el ADN viral entra en el citoplasma donde forma estructuras mini-cromosómicas altamente empaquetadas que, a continuación, entran en el núcleo de la célula huésped. El ADN viral se transcribe en ARN con poliadenilación que es terminalmente redundante debido a que la transcripción se produce dos veces para algunas partes del ADN. El ARN transcrito entra nuevamente en el citoplasma, en donde puede realizar dos funciones. Puede utilizarse como plantilla para la síntesis de las proteínas virales o puede someterse a la transcripción inversa por la transcriptasa inversa codificada por el virus para fabricar ADN monocatenario. Este ADN puede entonces volver a introducirse el núcleo para repetir el proceso (amplificación).
Puesto que la replicación requiere de ARN intermedio, los virus de la familia Caulimoviridae no son verdaderos virus ADN bicatenarios. En lugar de ello se consideran virus ADN bicatenarios retrotranscritos. Esta característica se da también en los retrovirus, sin embargo, existen varias diferencias importantes. Los virus de la familia Caulimoviridae, a diferencia de los retrovirus, no requieren la integración viral en el genoma del huésped con el fin de replicarse y por esta razón su genoma no codifica la proteína enzimática integrasa.
Caulimovirus ( Caulimoviridae ) son pararetrovirus de ADN que se replican en las plantas a través de un intermediario de ARN evolucionado a partir de LTR retroelements (para más detalles, verBousalem et al. 2008 ). "Pararetrovirus" es el término introducido por Temin (1985) para definir los animales ( Hepadnaviridae ) y los virus de plantas ( Caulimoviridae ) que difieren de los retrovirus, sobre la base de su genoma de ADN y en su integración no regular en el genoma del huésped para la replicación. Hasta la fecha, se sabe sin embargo que las secuencias genómicas de sólo unos pocos pararetrovirus de plantas, tanto como la petunia de la vena para eliminar el virus (PVCV) ( Richert-Pöggeler y Shepherd 1997 ) y badnavirus del rayado del banano (BSV) ( Ndowora et al.1999 ; Harper et al., 2005 ), se integran en sus genomas de acogida, y que los elementos integrados puede dar lugar a virosis episomal ( Ndowora et al. 1999 ). Se da por supuesto que estas secuencias integradas son reliquias de antiguos eventos de infección ( Harper et al. 2005 ) o intermedios de secuencias representativas entre caulimovirus y retrotransposones LTR ( Bousalem et al.2008 ; Llorens et al. 2009 ).
la morfología del virión
Caulimovirus generalmente se forman partículas de virus no envueltos que pueden ser ya sea bacilliform o isométrica. Como se muestra en la figura siguiente, estos son aproximadamente 35 a 50 nm de diámetro y 900 nm de longitud (para los bacilliforms) o 45 a 50 nm de diámetro y de simetría icosaédrica (para los isométricos). El genoma de caulimovirus es un ADN de doble cadena semi circular de aproximadamente 6-8 kb de tamaño, que está flanqueado por repeticiones terminales directos reiterado internamente en una forma invertida (para más detalles ver Fauquet y col ., 2005). El genoma caulimoviral también tiene una región intergénica de poli (A) (que también puede estar ausente), y discontinuidades o huecos de cadena sencilla en sitios específicos de ambas cadenas ( Harper et al. 2002 ).
estructura genómica
Como se ha señalado en el apartado anterior, caulimovirus tienen la ubicación taxonómica oficial dentro de la nomenclatura viral como pararetrovirus pero a pesar de que son virus de ADN y no tienen LTR, que son evolutivamente relacionados con LTR retroelements eucariotas (en particular con los de la Ty3 / gitana familia, Llorens et al. 2009 ) sobre la base de un ancestro gag-pol (sobre este tema, véase también Koonin et al. 1991 ; Bousalem et al. 2008 ; Staginnus et al . 2009 ).
Caulimovirus suelen presentar otros genes, como los que codifican para el Movimiento (MP o MOV) de proteínas, la transmisión del factor de áfidos (ATF), la proteína asociada a virus (VAP) y el / proteína viroplasmin o inclusión proteína de la matriz del cuerpo transactivador (TAV o IBMP) así como diversos genes adicionales (que varían en función del linaje), que son necesarios para su ciclo de vida viral y la transmisión (para más detalles ver Fauquet y col ., 2005 ). Tras esto, las tendencias anteriores corresponden caulimovirus a otros sistemas de virus basados en la acción ordinaria de la proteína de movimiento ( Koonin et al. 1991 ), argumentando que el origen más probable de caulimovirus era quimérico (un híbrido entre retrotransposones LTR y otros virus de ARN).
Replicación
La replicación implica dos etapas: la transcripción de un ARN intermedio a partir del ADN viral en el núcleo y luego invertir la transcripción de este ARN aumentando dsDNA en el citoplasma. El genoma de pararetroviruses también contiene una secuencia complementaria a una planta de tRNA Met que corresponde al sitio de iniciación de la replicación del ADN. Por lo general, este sitio se encuentra en el interior o aguas abajo de la gran región intergénica (región no codificante) y generalmente se designa nucleótidos 1 ( de Kochko et al. 1998 ). En contraste con los retrovirus, pararetroviruses de plantas no requieren su integración en el genoma del huésped para su replicación, por lo tanto su genoma no codifica la proteína integrasa. Aunque esta es una característica de pararetroviruses, las secuencias de ciertos Caulimoviridae especies, denominadas endógena pararetroviruses (EPRVs, Staginnus et al . 2009 ) muestran un dominio de integrasa putativo similares a los encontrados en los retrovirus y retrotransposones de la Ty3 / Gypsy familia ( Richert-Pöggeler y Shepherd 1997 ). En particular, los tres virus, BSV, PVCV y Tabaco virus de compensación vena(TVCV) han demostrado tener infecciones episomales asociados con secuencias integradas ( Harper et al. 2002 ).
distribución biológica y la sintomatología de acogida
Los huéspedes naturales de Caulimoviridae especies pertenecen al reino Plantae (angiospermas de Dicotiledóneas y Monocotyledonae clases). Dependiendo de los géneros, la transmisión del virus natural podría occurr a través de un vector de insecto ( Hemiptera insectos de las familias Aleyrodidae Aphididae , Cicadellidae , Pseudococcidae ) o por el contacto entre la planta alberga así como por las semillas o por el polen. La transmisión también puede llevarse a cabo mediante técnicas tales como la inoculación mecánica e injerto (para más detalles ver Fauquet y col ., 2005 ). Los síntomas característicos de plantas asociadas a la infección por caulimovirus comprenden: la vena de minas, anillado mosaico, manchas necróticas, líneas y manchas cloróticas, retraso en el crecimiento, deformación de las hojas, la hoja de malformación, la torsión de folletos, etc.
la historia evolutiva y taxonomía viral
De acuerdo con el Comité Internacional de Taxonomía de Virus (ICTV, Fauquet y col ., 2005 ), la Caulimoviridae puede dividirse taxonómicamente en seis géneros: caulimovirus , Soymovirus ,Cavemovirus , Tungrovirus , badnavirus y Petuvirus . Los análisis de la reconstrucción filogenética basada en pol recoger estos seis géneros en cuatro clases: Clase 1 incluye los géneroscaulimovirus y Soymovirus ; Clase 2 comprende Tungroviruses y badnavirus (el grupo más abundante de Caulimoviridae especies); Clase 3 representa el Cavemovirus género; y, finalmente, clase 4 comprende el virus de la petunia de la vena de Compensación (PVCV), que hasta la fecha es la única especie molecular conocido de Petuvirus género ( Llorens et al. 2009 ).
| Clase | Anfitrión Phyla | Género | especie tipo |
|---|---|---|---|
| Clase 1 | Las plantas terrestres ( Viridiplantae ) | caulimovirus | Virus del mosaico de la coliflor (CaMV) |
| Clase 1 | Las plantas terrestres ( Viridiplantae ) | Soymovirus | Soja virus del moteado clorótico (SbCMV) |
| Clase 2 | Las plantas terrestres ( Viridiplantae ) | badnavirus | Commelina virus del moteado amarillo (CoYMV) o (ComYMV) |
| Clase 2 | Las plantas terrestres ( Viridiplantae ) | Tungrovirus | Rice virus tungro bacilliform (RTBV) |
| Clase 3 | Las plantas terrestres ( Viridiplantae ) | Cavemovirus | La yuca vena virus del mosaico (CsVMV) |
| clase 4 | Las plantas terrestres ( Viridiplantae ) | Petuvirus | Petunia virus de compensación vena (PVCV) |
Clase 1
Clase 1 incluye dos géneros - caulimovirus y Soymovirus .
caulimovirus
Las especies que pertenecen a este género forman partículas isométricas con un diámetro entre 35 y 50 nm. El genoma viral contiene una sola molécula de ADN de doble cadena circular de aproximadamente 8000 pb de longitud que codifica para 6 o 7 ORF por lo general en el orden tratan en la siguiente figura, que es un consenso de longitud completa idealizada (para más detalles ver Fauquet et col ., 2005 ). Por lo general, los genomas de caulimovirus especies contienen dos grandes secuencias de promotor de la transcripción: uno situado en el extremo 3 'del ORF VI (TAV) y que se extiende en la gran región intergénica que transcribe todo el genoma del virus (una transcripción de longitud completa equivalente a CaMV 35S de transcripción); mientras que la segunda, situada en el extremo 3 'del ORF V (Pol) y que se extiende en la pequeña región intergénica entre ORF V y VI, transcribe solamente el ORF VI, (a sub-genómico transcripción equivalente a CaMV 19S transcripción) ( Bhattacharyya et al. 2002 ).
Los anfitriones naturales de plantas del género caulimovirus son angiospermas de Dicotyledonae clase. Estos virus se transmiten por lo general en forma no persistente o semi-por vectores biológicos, o mediante injerto, la inoculación mecánica o por el contacto entre los hosts. Los vectores de transmisión por lo general son insectos del orden Hemiptera ( Aphididae familia), en la que el virus no se replica. En condiciones experimentales, las especies huéspedes susceptibles se encuentran en las familias Amaranthaceae , Caryophyllaceae , Chenopodiaceae , Compositae ,Convolvulaceae , Cruciferae , Ericaceae , Euphorbiaceae , Leguminosae-Papilionoideae , Nyctaginaceae , Plantaginaceae , Ranunculaceae , Resedaceae , Rosaceae , Scrophulariaceae ,solanáceas . Sin embargo, algunas especies de estas familias inoculados con el virus no muestran signos de susceptibilidad (es decir: Beta vulgaris, Brassica campestris Capsicum annuum, Chenopodium amaranticolor, Chenopodium quinoa, Cucumis sativus, Lycopersicon esculentum, Nicotiana benthamiana, Nicotiana clevelandii, Nicotiana tabacum, Petunia x hybrida , Phaseolus vulgaris, Pisum sativum, Spinacia oleracea, Vicia faba , etc).
De acuerdo con el ICTV ( Fauquet et al 2005. ) Del caulimovirus género contiene especies virales tan diversos como el virus del mosaico de la coliflor (CaMV); Clavel grabado virus de anillo(CERV); Escrofularia virus del mosaico (FMV); el virus del mosaico de la dalia (DMV); fresa virus vein banding (SVBV); rábano picante virus latente (HRLV); el virus del mosaico Mirabilis (MiMV); el virus del moteado del cardo (ThMoV); Aquilegia virus del mosaico necrótico (ANMV, especie provisional).
Soymovirus
Soymoviruses forman partículas virales redondas con simetría icosaédrica. La cápside isométrica tiene un diámetro de 42-50 nm y el genoma viral contiene una sola molécula de ADN de doble cadena de aproximadamente 8200 pb de largo que forma un círculo abierto (para más detalles ver Fauquet y col ., 2005 ). Contiene 8 ORF que codifica para ambas proteínas estructurales y no estructurales (la figura muestra un consenso de longitud completa idealizado). Soymovirus género difiere de otros caulimovirus por la ocurrencia de tres ORFs entre ORF I y ORF IV en lugar de dos (ORF II y III) que muestran poca o ninguna similitud con otros caulimovirus ORFs ( Hasegawa et al 1989. ; Mushegian . et al 1995 ). ORF VII revela similitud de secuencia con el dominio típico de la proteasa, no está claro si esta secuencia es una proteasa funcionalmente activo adicional a la encontrada en pol más que los méritos de características aún más atención, ya que se conserva taxonómicamente en casi (pero no todos) soymoviruses. Por otra parte este género se diferencia en la ubicación del sitio de unión del cebador-putativo (PBS) que en los otros caulimovirus normalmente se encuentra en la región intergénica, mientras que en soymoviruses se ha encontrado dentro de ORF A (PsCV) o ORF Ia (SbCMV) o entre ORF A y B (BRRV) ( Mushegian et al.1995 ; Hasegawa et al 1989. ; Glasheen . et al 2002 ).
Los huéspedes vegetales naturales de soymoviruses son angiospermas de Dicotyledonae clase. En condiciones experimentales especies huéspedes susceptibles se encuentran en la familiaLeguminosae - Papilionoideae . Los virus se transmiten mediante inoculación mecánica, mientras que no se transmiten por las semillas. De acuerdo con el ICTV ( Fauquet et al 2005. ) ElSoymovirus género incluye tres especies - virus de la soja del moteado clorótico (SbCMV), cacahuete virus de la raya cholorotic (PCSV) y arándano virus de la mancha anular de color rojo (BRRV) pero aunque especie provisional ninguno ha sido aún informado , Stavolone et al. (2003) describen la nueva pararetrovirus Cestrum virus amarillo de curling (CmYLCV) estrechamente relacionada con SbCMV.
Clase 2
Clase 2 se compone de otros dos géneros ( Tungrovirus y badnavirus ) y es la rama más abundante de Caulimoviridae elementos.
Este género se caracteriza por las especies con viriones baciliformes que tienen una longitud de 95-130 nm, o 60 a 900 nm, así como una anchura de 24-35 nm (para más detalles ver Fauquet y col., 2005 ). El genoma de badnavirus por lo general contiene una sola molécula de ADN de doble cadena de aproximadamente 7.200-7.600 pb de longitud que forma un círculo abierto interrumpido por discontinuidades específicas de sitio y que podrían contener un poli intergénicas región (A) (es decir CoYMV, Medberry et al . 1990 ) . El genoma del virus por lo general códigos para 3 ORFs, pero en algunos casos pueden presentar una o cuatro ORFs adicionales que funciona ya están en estudio. El producto poliproteína ORF III más grande contiene la proteína de movimiento, la proteína de cubierta del virus (gag), la proteasa aspártico, la transcriptasa inversa y los dominios de la ribonucleasa H ( Medberry et al 1990. ; Bouhida et al. 1993 ; Briddon et al 1999. ) . La capa (gag) de dominio de badnaviral ORF III muestra en el C-terminal de una gran región rica en duplicaciones con dedos de zinc (CCHC) de matriz de manera similar a los de la nucleocápside retroelement LTR ( Bouhida et al. 1993 ; Llorens et al. 2009 ). Además, algunos badnavirus muestran un dominio dUTPase adicional aguas arriba o aguas abajo para el dominio COAT dentro de ORF III similar a la de varios Retroviridae retrovirus ( Elder et al . 1992 ) y varios Ty3 / Gypsy retrotransposones LTR ( Novikova y Blinov 2008 ). La siguiente figura muestra un idealizado Badnavirus-longitud completa del genoma de consenso.
Los huéspedes de plantas por lo general pertenecen a las Dicotiledóneas y Monocotyledonae clases. Dependiendo de las badnavirus especies, el virus puede ser transmitido por inoculación mecánica, por injerto, por semillas o por el polen, pero no por el contacto entre hosts. Badnavirus también pueden transmitirse en un semi o de manera persistente a través de vectores de insectos del orden Hemiptera ( Aleyrodidae , Aphididae , Cicadellidae y Pseudococcidae familias). Además, virus del rayado del banano infecciones (BSV) pueden surgir en plantas sanas de secuencias integradas (como resultado del proceso de in vitro de propagación) durante el cultivo de tejido ( Ndowora et al. 1999 ; Harper et al. 2002 ). De acuerdo con el ICTV ( Fauquet et al. 2005 ) laBadnavirus género contiene numerosas especies y especie provisional, entre los cuales: Commelina virus del moteado amarillo (CoYMV o ComYMV) (especie tipo del género), Aglaonema virus bacilliform (ABV), del rayado del banano virus (BSV), Cacao virus de rodaje hinchada (CSSV), Citrus virus del mosaico amarillo (CMBV), Dioscorea virus alata bacilliform (DaBV), Kalanchoe top virus-manchado (KTSV), caña de azúcar virus bacilliform (ScBV), se han descrito junto con especie provisional como el virus de la bacilliform piña (PBV), virus de la yuca bacilliform (YBV),stilbocarpa virus del mosaico bacilliform (BVSM).
Tungrovirus
Sólo una especie se ha denominado Tungrovirus género: Arroz virus tungro bacilliform (RTBV). La especie se compone de partículas baciliformes largos 110-400 nm y con una anchura de 30-35 nm que encapsidan un ADN de doble cadena circular de aproximadamente 8000 pb de longitud (para más detalles ver Fauquet y col ., 2005 ). El genoma contiene cuatro ORFs que potencialmente codifican para cuatro proteínas (P24, P12, P194, P46, respectivamente, de Hay et al. 1991 ). El mayor ORF (III) codifica para una poliproteína que contiene la proteína de movimiento, así como la proteína de la cubierta, la proteasa, la transcriptasa inversa y la RNasa H dominios característicos de retroelements. Las funciones de los otros tres ORFs tungrovirus son desconocidos o no está bien demostrado aún ( Hay et al. 1991 ; Casco 1996 ), (figura muestra la organización del genoma de la especie tipo del arroz virus tungro bacilliform , RTBV).
Los huéspedes de plantas de RTBV pertenecen a las angiospermas de Monocotyledonae clase. El virus se transmite por vectores de insectos del orden Hemiptera , familia Cicadellidae de una manera semi-persistente y que requiere la presencia de la Rice tungro virus esférico virus helper (RTSV) para la transmisión. RTBV y RTSV son ambos responsables de la "enfermedad del virus del arroz tungro", una de las principales causas de la pérdida de la producción de arroz en el sur y sudeste de Asia ( Casco 1996 ). Secuencias endógenas RTBV similar (ERTBVs) se han encontrado en el genoma del arroz ( Kunii et al. 2004 ). A pesar de que contienen estructuras reordenados y no hay ORFs intactas, las comparaciones de sus secuencias de ADN y de aminoácidos sugieren su relación estrechamente a RTBV y el posible papel de estas secuencias integradas contra la enfermedad viral relacionada ( Kunii et al. 2004 ).
Clase 3
Clase 3 representa el género Cavemovirus .
Cavemovirus
Este género consiste en dos especies virales - yuca vena virus del mosaico (CsVMV) y Tabaco virus de compensación vena (TVCV) - (de acuerdo con el ICTV Fauquet et al . 2005 ) y apoyado por los análisis filogenéticos ( Llorens et al. 2009 ). Estos forman cápsides redondo a alargado con simetría icosaédrica y la longitud y la anchura de 95-130 nm (o 60 a 900 nm) y 24-35 nm, respectivamente (para más detalles ver Fauquet y col ., 2005 ). El genoma viral por lo general consta de una sola molécula de ADN de doble cadena circular de aproximadamente 7.7 a 8.1 kb de longitud que codifica para 4 o 5 ORF (la figura muestra un consenso idealizado de la organización del genoma cavemoviruses).
Cavemoviruses difieren de otros Caulimoviridae géneros, en el orden de sus dominios de la capa y el movimiento ( de Kochko et al. 1998 ; Calvert et al. 1995 ; Lockhart et al. 2000 ). En cavemoviruses, mov está aguas abajo a la capa. Adicionalmente, CsVMV aparentemente codifica para un dominio de proteasa adicional, que se encuentra dentro de ORF I y aguas arriba en el dominio de la proteasa pol convencional. Esta característica se encuentra en otros Caulimoviridae especies, pero no en TVCV. Por otro lado, una observación interesante ha sido propuesto porLockhart et al. (2000) que sugieren que la forma episomal de TVCV encontró en los infectados híbrido especies tabaco N. edwardsonii ( N. clevelandii x N. glutinosa ), probablemente surge a partir de secuencias integradas pararetrovirales presentes en el genoma de la planta huésped y que se heredan del padre masculino ( glutinosa N. ).
clase 4
Esta clase describe el género Petuvirus , que cae en la posición más profunda de la Caulimoviridae filogenia.
Petuvirus
Este es un género hasta la fecha compuesto únicamente por un solo representante secuencia, la petunia virus de compensación vena (PVCV), lo que podría constituir la posición más basal en elCaulimoviridae filogenia ( Bousalem et al. 2008 ; Llorens et al. 2009 ). Esta especie viral consiste en partículas isométricas no envueltas de 43-46 nm de diámetro (para más detalles ver Fauquet et al . 2005 ). El genoma no está segmentado y se compone de una molécula de circular dsDNA 7206 pb de largo, organizado en un gran ORF que contiene la proteína de movimiento típico de caulimovirus, un putativo [HHCC y DD (35) E] integrasa similares a los codificados por retrotransposones LTR, y la capa típica (gag) y pol dominios (transcriptasa-RNaseH proteasa inversa) (Richert-Pöggeler y Shepherd 1997 ; Harper et al. 2002 ). La presencia de una función de la integrasa probable en el genoma de este virus sugiere que PVCV existe como un retroelement viral que puede también tiene el potencial para transponer en Petunia ( Richert-Pöggeler y Shepherd 1997 ).
Los huéspedes de plantas de PVCV pertenecen a las solanáceas familia (angiospermas de Dicotyledonae clase). El virus se ha encontrado en Petunia x hybrida (petunia jardín) en la que los síntomas característicos son vena compensación y la hoja de malformación, y es muy transmite por semilla y el injerto pero no por inoculación mecánica o por insectos vectores (áfidos) ( Harper et al . 2002 ).
No hay comentarios:
Publicar un comentario