El aprendizaje automático, un subcampo de las ciencias de la computación que implica el desarrollo de algoritmos que aprenden cómo hacer predicciones basadas en datos, tiene una serie de aplicaciones emergentes en el campo de la bioinformática. La bioinformática trata con enfoques computacionales y matemáticos para comprender y procesar datos biológicos. Antes de la aparición de los algoritmos de aprendizaje automático, los algoritmos bioinformáticos tenían que programarse a mano, lo que para problemas como la predicción de la estructura de las proteínas resulta extremadamente difícil. Las técnicas de aprendizaje automático como el aprendizaje profundo permiten al algoritmo utilizar el aprendizaje de características, lo que significa que, basándose solo en el conjunto de datos, el algoritmo puede aprender cómo combinar múltiples características de los datos de entrada en un conjunto más abstracto de características para realizar aprendizaje adicional. Este enfoque multicapa de los patrones de aprendizaje en los datos de entrada permite que dichos sistemas hagan predicciones bastante complejas cuando se entrena con grandes conjuntos de datos. En los últimos años, el tamaño y la cantidad de conjuntos de datos biológicos disponibles se han disparado, lo que permite a los investigadores en bioinformática hacer uso de estos sistemas de aprendizaje automático. El aprendizaje automático se ha aplicado a seis subcampos principales de la bioinformática: genómica, proteómica, microarrays, biología de sistemas, evolución y minería de textos.
Aplicaciones[editar]
Genómica[editar]
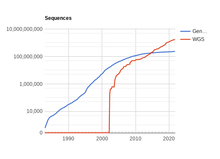
La genómica involucra el estudio del genoma, la secuencia completa de ADN de los organismos. Si bien los datos de la secuencia genómica han sido históricamente escasos debido a la dificultad técnica para secuenciar un fragmento de ADN, el número de secuencias disponibles está creciendo exponencialmente.1 Sin embargo, aunque los datos en bruto están cada vez más disponibles y accesibles, la interpretación biológica de estos datos está ocurriendo a un ritmo mucho más lento.2 Por lo tanto, existe una necesidad creciente de desarrollar sistemas de aprendizaje automático que puedan determinar automáticamente la ubicación de los genes que codifican proteínas dentro de una secuencia de ADN dada. Este problema es conocido en la biología computacional como predicción de gen.
La predicción de genes se realiza comúnmente a través de una combinación de lo que se conoce como búsquedas extrínsecas e intrínsecas. Para la búsqueda extrínseca, la secuencia de entrada de ADN se ejecuta a través de una gran base de datos de secuencias cuyos genes han sido previamente descubiertos y sus ubicaciones anotadas. Se pueden identificar varios genes de la secuencia determinando qué cadenas de bases dentro de la secuencia son homólogas a las secuencias de genes conocidas. Sin embargo, dada la limitación en el tamaño de la base de datos de secuencias génicas conocidas y anotadas, no todos los genes en una secuencia de entrada dada pueden identificarse a través de la homología por sí sola. Por lo tanto, se necesita una búsqueda intrínseca cuando un programa de predicción de genes intenta identificar los genes restantes de la secuencia de ADN sola.
El aprendizaje de máquina es también utilizado para el problema de alineación de secuencia múltiple qué implica alinear muchas secuencias de ADN o aminoácidos para determinar regiones de semejanza que podría indicar una historia evolutiva compartida. También suele detectar y visualizar reordenamientos genómicos.3
Proteómica[editar]
Las proteínas, cadenas de aminoácidos, obtienen gran parte de su función del plegamiento de proteínas en el que se conforman en una estructura tridimensional. Esta estructura está compuesta de varias capas de plegado incluyendo la estructura primaria (i.e. la cadena plana de aminoácidos), la estructura secundaria (helices alfa y láminas beta), la estructura terciaria, y la estructura cuaternaria.
La predicción de la estructura secundaria de proteínas es uno de los focos principales de este subcampo ya que los pliegues adicionales de proteínas (estructuras terciarias y cuaternarias) se determinan con base en la estructura secundaria. Resolver la verdadera estructura de una proteína es un proceso increíblemente costoso y que requiere mucho tiempo, promoviendo la necesidad de sistemas que puedan predecir con precisión la estructura de una proteína mediante el análisis directo de la secuencia de aminoácidos. Antes del aprendizaje automático, los investigadores debían realizar esta predicción manualmente. Esta tendencia comenzó en 1951 cuando Pauling y Corey publicaron su trabajo sobre la predicción de las configuraciones de enlaces de hidrógeno de una proteína de una cadena polipeptídica.4 Hoy en día, mediante el uso del aprendizaje automático de características, las mejores técnicas de aprendizaje automático pueden alcanzar una precisión del 82-84%. El estado actual de la técnica en la predicción de estructuras secundarias utiliza un sistema llamado DeepCNF (campos neuronales convolucionales profundos) que se basa en el modelo de aprendizaje automático de redes neuronales artificialespara lograr una precisión de aproximadamente 84% cuando se clasifican los aminoácidos de una secuencia de proteína en una de las tres clases estructurales (hélice, lámina o espiral).5 El límite teórico para la estructura secundaria de proteínas de tres estados es 88-90%.
El aprendizaje de máquina también ha sido aplicado a problemas de proteómica como la predicción de cadenas laterales de proteínas, el modelado de bucles de proteínas, y la predicción de mapas de contacto de proteína.
Microarrays[editar]
Microarrays, es un tipo de "lab on a chip" utilizado para recopilar datos de forma automática sobre grandes cantidades de material biológico. El aprendizaje automático puede ayudar en el análisis de estos datos, y se ha aplicado a la identificación del patrón de expresión, la clasificación y la inducción de la red genética.
Esta tecnología es especialmente útil para controlar la expresión de genes dentro de un genoma, ayudando a diagnosticar diferentes tipos de cáncer con base en qué genes se expresan.6 Uno de los principales problemas en este campo es identificar qué genes son expresados basándose en los datos recopilados. Además, debido a la gran cantidad de genes en los que los datos son recolectados por los microarrays, existe una gran cantidad de datos irrelevantes para la tarea de identificación genética expresada, lo que complica aún más este problema. El aprendizaje automático presenta una posible solución a este problema, ya que se pueden usar varios métodos de clasificación para realizar esta identificación. Los métodos más comúnmente utilizados son redes radiales de funciones básicas, aprendizaje profundo, clasificación bayesiana, árboles de decisión, y bosque aleatorio.
Biología de sistemas[editar]
La biología de sistemas se centra en el estudio de los comportamientos emergentes a partir de interacciones complejas de componentes biológicos simples en un sistema. Dichos componentes pueden incluir moléculas como ADN, ARN, proteínas y metabolitos.7
El aprendizaje automático se ha utilizado para ayudar a modelar estas interacciones complejas en sistemas biológicos en dominios tales como redes genéticas, redes de transducción de señales y vías metabólicas. Los modelos gráficos probabilistas, una técnica de aprendizaje automático para determinar la estructura entre diferentes variables, son uno de los métodos más utilizados para modelar redes genéticas. Además, el aprendizaje automático se ha aplicado a problemas de biología de sistemas tales como la identificación de sitios de unión del factor de transcripción utilizando una técnica conocida como optimización de cadena de Markov. Los algoritmos genéticos, técnicas de aprendizaje automático que se basan en el proceso natural de la evolución, se han utilizado para modelar redes genéticas y estructuras reguladoras.
Otras aplicaciones de biología de sistemas de aprendizaje automático incluyen la tarea de predicción de función enzimática, análisis de datos de micromatriz de alto rendimiento, análisis de estudios de asociación de genoma completo para comprender mejor los marcadores de esclerosis múltiple, predicción de función de proteína e identificación de sensibilidad de genes NCR en levadura.8
Minería de textos[editar]
El aumento de las publicaciones biológicas disponibles provocó un aumento de la dificultad en la búsqueda y la compilación de toda la información relevante disponible sobre un tema determinado en todas las fuentes. Esta tarea se conoce como extracción de conocimiento. Esto es necesario para la recopilación de datos biológicos que a su vez puede alimentar algoritmos de aprendizaje automático para generar nuevos conocimientos biológicos.9 El aprendizaje automático se puede utilizar para esta tarea de extracción de conocimiento usando técnicas tales como el procesamiento del lenguaje natural para extraer la información útil de los informes generados por humanos en una base de datos.
Esta técnica se ha aplicado a la búsqueda de nuevas dianas farmacológicas, ya que esta tarea requiere el examen de la información almacenada en bases de datos y revistas biológicas. Las anotaciones de proteínas en las bases de datos de proteínas a menudo no reflejan el conjunto de conocimiento conocido completo de cada proteína, por lo que se debe extraer información adicional de la literatura biomédica. El aprendizaje de máquina ha sido aplicado a anotación automática de la función de genes y proteínas, determinación de la localización subcelular de una proteína, análisis de análisis de matrices de expresión de ADN, análisis de interacción de proteínas a gran escala y análisis de interacción de moléculas.
Otra aplicación es la detección y visualización de regiones que comparten un alto grado de semejanza o son nuevos según una referencia.
La atracción de ramas largas es un error metodológico que se produce en los análisis de filogenia molecular cuando grupos que han evolucionado rápidamente son erróneamente colocados en la base de los árboles filogenéticos. El error es muy común cuando la evolución de un gen no ha seguido un modelo de reloj moleculary tiende a registrarse cuando se mide con el método de máxima parsimonia. Este método, como otros que se rigen por un sistema de puntuación, no es capaz de diferenciar un gran cambio repentino de una divergencia evolutiva ancestral.
Así, por ejemplo, los microsporidios han simplificado extremadamente su ARNr haciendo que sus secuencias parezcan más divergentes comparadas con las de sus parientes próximos. Esto provoca que en los árboles filogenéticos realizados con este gen aparezcan como un grupo eucariota muy primitivo mientras que los demás análisis los muestran como un grupo derivado de los hongos. Así, la interpretación de los datos tiene que realizarse con precaución.
Un ejemplo muy conocido de este error es el que hizo que Carl Woese sugiriera en su sistema de los tres dominios que los eucariotas eran uno de los clados más antiguos de la naturaleza junto con otros dos clados bacterianos, lo que se sabe que es posible tanto sobre la base de análisis morfológicos como paleontológicos, pero que debido a los grandes cambios que experimentó evolutivamente este grupo aparece así invariablemente en los análisis moleculares de la mayoría de genes.
Este error se encuentra sobre todo si se usa sólo una molécula para los análisis (por ejemplo con los árboles filogenéticos con sólo ARNr). Se puede intentar rectificar comparando muchos datos a la vez, comprobando así la robustez del árbol. Usando métodos que incorporen ritmos diferentes de sustitución entre linajes (máxima probabilidad, por ejemplo), acompañando los análisis genéticos con análisis morfológicos, o rompiendo las ramas largas añadiendo taxones que estén relacionados con los que tienen estos tipos de ramas.
base de datos biológica es una colección de información sobre ciencias de la vida, recogida de experimentos científicos, literatura publicada, tecnología de experimentación de alto rendimiento, y análisis computacional.1 Contiene información de áreas de investigación incluyendo genómica, proteómica, metabolómica, expresión génica mediante microarrays, y filogenética.2 La información contenida en bases de datos biológicas incluye funciones, estructura y localización (tanto celular como cromosómica) de genes, efectos clínicos de mutaciones, así como similitudes de secuencias y estructuras biológicas.
En los últimos años, debido a la rápida evolución de las técnicas experimentales de alto rendimiento (Secuenciación del ADN, Cristalografía de rayos X, Microarreglo de ADN) se generó un crecimiento exponencial en la cantidad de datos biológicos (secuencias genómicas y de proteínas, estructuras de proteínas, expresión génica, mutaciones, etc) que generaron la necesidad de contar con formas eficientes de almacenar la información.
Descripción[editar]
Las bases de datos biológicas constituyen una herramienta esencial para almacenar, estructurar, organizar, actualizar y manipular datos biológicos. La variedad de éstos datos, así como también su rápido crecimiento, hacen a las bases de datos una herramienta clave. Se han convertido en un instrumento indispensable para los científicos experimentales del campo de la biología, como para aquellos científicos del área de la bioinformáticaque desarrollan experimentos in silico.
Las bases de datos biológicas surgen a partir de los conceptos de bases de datos relacionales de las ciencias de la computación, y los conceptos de recuperación de información de las bibliotecas digitales. El diseño de estas bases de datos, su desarrollo y su gestión a largo plazo, forman un área nuclear dentro de la bioinformática.3 El contenido de los datos incluye secuencias génicas, descripciones textuales, atributos y clasificaciones ontológicas, estructuras de proteínas, anotaciones, entre otras. Estos son descritos a menudo como datos semi-estructurados, y se pueden representar como tablas, registros delimitados por claves y estructuras XML. Son comunes las referencias cruzadas entre las diferentes bases de datos biológicas usando los números de acceso (identificadores únicos de los registros en una base de datos, o también conocidos como Clave primaria).
Las bases de datos para ayudan a los científicos a comprender y explicar una serie de fenómenos biológicos desde la estructura biomolecular de una proteína y su interacción, hasta el metabolismo completo de los organismos y a la comprensión de la evolución de las especies.
Un recurso importante para la búsqueda de bases de datos biológicos es la edición anual de la revista Nucleic Acids Research (NAR). Una edición de bases de datos en NAR está disponible gratuitamente todos los años, donde se publican nuevas base de datos y algunas actualizaciones de las ya conocidas. Se encuentran clasificadas de acuerdo a su temática y están en línea a disposición de toda la comunidad científica.
Clasificación de bases de datos biológicas[editar]
Las bases de datos biológicas se han desarrollado para diversos propósitos, almacenan varios tipos de datos heterogéneos y son curadas a distintos niveles con diferentes métodos, por lo tanto hay diferentes criterios para su clasificación.45
Alcance y cobertura de los datos[editar]
De acuerdo a este criterio, las bases de datos pueden clasificarse en exhaustivas o especializadas:
- Exhaustivas: abarcan diferentes tipos de datos de muchas especies, ejemplos típicos son GenBank la base de datos moleculares mantenidos por el European Bioinformatics Institute European Molecular Biology Laboratory (EMBL-EBI) y DNA Data Bank of Japan (DDJB). Estas tres bases de datos fueron establecidas como una Colaboración Internacional de Bases de Datos de Secuencias de Nucleótidos en 1988, para colectar y compartir secuencias de ADN y ARN
- Especializadas: contienen información específica o de especies particulares. Por ejemplo WormBase que contiene información biológica y genómica de nemátodos.
Según la fuente de los datos[editar]
De acuerdo a este criterio, las bases de datos pueden clasificarse como primarias, secundarias y combinadas:
- Primarias: contienen información solamente de la secuencia o la estructura, es decir que los datos experimentales son directamente subidos a la base de datos. En esta categoría encontramos las bases de datos GenBank, DNA Data Bank of Japan (DDJB)], UniProtKB/TrEMBL y Protein Data Bank (PDB)
- Secundarias: contienen información derivada de las bases de datos primarias. Una base de datos secundaria de secuencia contiene información de la conservación de la secuencia, patrones de secuencia y residuos del sitio activo de familias de proteínas derivados de alineamientos múltiples entre secuencias evolutivamente relacionadas. Una base de datos secundaria de estructuras organiza las entradas de PDB clasificándolas, por ejemplo, de acuerdo a su estructura como todas alfa, todas beta, alfa-beta, etc. Algunos ejemplos de éstas bases de datos son: CATH y SCOP.
- Compuestas: combinan una variedad de fuentes primarias de datos, como por ejemplo, el National Center for Biotechnology Information (NCBI) que alberga un conjunto de bases de datos de secuencia, taxonomía, genomas, mutaciones, entre otras y además herramientas como BLAST para búsquedas por similitud de secuencia.
Nivel de curación[editar]
De acuerdo al nivel de curación, pueden clasificarse en bases de datos primarias, secundarias o derivadas:
- Primarias: contienen datos “crudos” a modo de repositorio de archivos como [[NCBI Sequence Read Archive] (SRA)'
- Secundarias o derivadas: almacena información que tiene un valor agregado por ser curada, por ejemplo NCBI RefSeq
Método de conservación[editar]
El crecimiento explosivo de la cantidad de datos disponibles requiere de curación, integración y anotación, que se logra mediante la colaboración colectiva. Desde este punto de vista, las bases de datos biológicas pueden clasificarse como:
- Conservadas por expertos, por ejemplo RefSeq y [The Arabidopsis Information Resource]] (TAIR)
- Conservadas por una comunidad de investigadores, de forma colectiva y colaborativa, por ejemplo LncRNA Wiki y GeneWiki
Tipo de datos almacenados[editar]
De acuerdo al tipo de datos almacenados en cada base de datos, las bases de datos biológicas pueden clasificarse de forma genérica en alguna de las siguientes categorías (se listan algunos ejemplos de bases de datos):
- Secuencias nucleotídicas (ADN y ARN): la colaboración de las tres bases de datos más importantes hace posible acceder a casi toda la información de secuencias de nucleótidos desde cualquiera de sus tres sedes
Bases de datos de EMBL en el European Bioinformatics Institute (EMBL-EBI). Enlace externo base de datos de nucleótidos de EMBL-EBI
DNA Data Bank of Japan (DDJB). Enlace externo DDJB
Si bien son mantenidas por distintos organismos en distintos países, existe una coordinación entre las distintas bases. Una secuencia enviada a cualquiera de las bases se verá reflejada en las otras dos en aproximadamente una semana, ya que esa es la frecuencia de actualización entre las distintas bases genéticas. Por este motivo es indistinto que base se use para enviar nuevas secuencias, aunque normalmente los europeos utilizan EMBL y los americanos GenBank.
- Proteínas: bases de datos de secuencias, estructuras, e información relacionada
UniProtKB/Swiss-Prot contiene secuencias anotadas o comentadas, es decir, cada secuencia ha sido revisada, documentada y enlazada a otras bases de datos. Enlaces externos UniProtKB, Swissprot en el EBIUniProtKB/TrEMBL por Translation of EMBL Nucleotide Sequence Database incluye la traducción de todas las secuencias codificantes derivadas del (EMBL) y que todavía no han podido ser anotadas en Swiss-Prot. Enlaces externos TrEMBL, UniProtKB 'PIR por Protein Information Resource está dividida en cuatro sub-bases que tienen un nivel de anotación decreciente. Enlace externo PIR
'ENZYME enlaza la clasificación de actividades enzimáticas completa a las secuencias de Swiss-Prot. Enlace externo ENZYME
'Protein Data Bank (PDB) es la base de datos de estructura terciaria 3D de proteínas que han sido cristalizadas. Enlace externo PDB
El portal de EMBL-EBI ofrece una variedad de bases de datos de expresión génica. Enlace externo a bases de datos de expresión de EMBL-EBI
- Interactomas, reactomas y rutas metabólicas
- Variación genética (SNPs) y enfermedad
COSMIC es un catálogo de mutaciones somáticas en cáncer, mantenida por el Wellcome Trust Sanger Institute. Enlace externo a COSMIC
- Literatura
- Ontología
El proyecto de Ontología Génica (GO) es un esfuerzo colaborativo que surgió de la necesidad de tener descriptores consistentes de los productos de genes depositados en distintas bases de datos. Enlace externo a Gene Ontology Consortium
Genomes server y TIGR son portales con información o enlaces de todos los genomas secuenciados por el momento, desde virus a humanos. Enlace externo Genome Server, enlace externo TIGR
- Otras
TAIR (The Arabidopsis Information Resource) es el portal de la planta modelo Arabidopsis thaliana. Enlace externo Arabidopsis
GYPSY, base de datos de elementos genéticos móviles. Enlace externo The GYPSY Database of Mobile Genetic Elements
Problemas por los formatos de entrada[editar]
Un problema fundamental en todas las grandes bases de datos genómicas es que los registros provienen de una gran variedad de fuentes, desde investigadores individuales hasta grandes centros de secuenciación. Como resultado, las secuencias mismas y principalmente las anotaciones biológicas adjuntas a estas secuencias, varían notablemente en calidad. También hay mucha redundancia ya que muchos laboratorios ingresan a menudo secuencias que son idénticas o muy similares a otras en la base de datos.
Muchas anotaciones no están basadas en experimentos de laboratorio sino en resultados de búsquedas de secuencias similares de secuencias previamente anotadas. Por supuesto, una vez que una secuencia es anotada basándose en su similitud con otra, puede servir como base para futuras anotaciones. Esto conduce al problema de las anotaciones transitivas, porque puede haber varias de esas secuencias transferidas por similitud de secuencia entre una base de datos de registro real y la información experimental de laboratorio. Por lo tanto, siempre hay observar el sentido biológico de las anotaciones en las principales bases de datos de secuencias con un considerable grado de escepticismo, a menos que pueda ser verificada por referencias a artículos publicados con la descripción de la alta calidad de los datos experimentales, o al menos por referencia a una secuencia de la base de datos arreglada por un humano.

No hay comentarios:
Publicar un comentario