La fotosíntesis anoxigénica bacteriana se distingue de la fotosíntesis oxigenada de la planta terrestre más familiar por la naturaleza del reductor terminal (por ejemplo, sulfuro de hidrógeno en lugar de agua) y en el subproducto generado (por ejemplo, azufre elemental en lugar de oxígeno molecular). Como su nombre lo indica, la fotosíntesis anoxigénica no produce oxígeno como un subproducto de la reacción. Varios grupos de bacterias pueden realizar la fotosíntesis anoxigénica: bacterias azufradas verdes (GSB), fotótrofos filamentosos rojos y verdes (FAP, por ejemplo, cloroflexi ), bacterias púrpuras , acidobacterias y heliobacterias . [1] [2]
Los pigmentos utilizados para llevar a cabo la fotosíntesis anaeróbica son similares a la clorofila, pero difieren en el detalle molecular y la longitud de onda máxima de la luz absorbida. Las bacterioclorofilas a través de gabsorben fotones electromagnéticos al máximo en el infrarrojo cercanodentro de su medio de membrana natural. Esto difiere de la clorofila a, la planta predominante y el pigmento de cianobacterias , que tiene una longitud de onda de absorción máxima aproximadamente 100 nanómetros más corta (en la porción roja del espectro visible).
Algunas arqueas (por ejemplo, Halobacterium ) capturan energía luminosa para la función metabólica y, por lo tanto, son fototróficas, pero se sabe que ninguna "fija" carbono (es decir, es fotosintética). En lugar de un receptor de tipo clorofila y una cadena de transporte de electrones, las proteínas como la halorrodopsina capturan la energía de la luz con la ayuda de diterpenospara mover los iones contra el gradiente y producir ATP a través de la quimiosmosis a la manera de las mitocondrias.
Hay dos tipos principales de cadenas de transporte de electrones fotosintéticos anaeróbicos en las bacterias. Los centros de reacción de tipo I que se encuentran en GSB, Chloracidobacterium y Heliobacteria y los centros de reacción de tipo II que se encuentran en FAP y bacterias púrpuras
Centros de reacción tipo I
La cadena de transporte de electrones de las bacterias de azufre verde, como la presente en el organismo modelo Chlorobaculum tepidum , utiliza el par de bacterioclorofila del centro de reacción , P840. Cuando la luz es absorbida por el centro de reacción, el P840 entra en un estado excitado con un gran potencial de reducción negativa y, por lo tanto, dona el electrón a la bacterioclorofila 663 que la pasa por la cadena de electrones. El electrón se transfiere a través de una serie de portadores y complejos de electrones hasta que se usa para reducir NAD + . La regeneración de P840 se realiza con la oxidación del ion sulfuro a partir de sulfuro de hidrógeno (o hidrógeno o hierro ferroso) mediante el citocromo C 555 [ citación necesaria ] .
Centros de reacción tipo II
Aunque los centros de reacción de tipo II son estructuralmente y secuencialmente análogos al fotosistema II(PSII) en cloroplastos y cianobacterias de plantas, los organismos conocidos que presentan fotosíntesis anoxigénica no tienen una región análoga al complejo de desarrollo de oxígeno de PSII.
La cadena de transporte de electrones de las bacterias púrpuras sin azufre comienza cuando el par de bacterioclorofilas del centro de reacción , P870, se excita por la absorción de la luz. El P870 emocionado donará un electrón a la bacteriofeofitina , que luego lo pasa a una serie de portadores de electrones a lo largo de la cadena de electrones . En el proceso, generará un gradiente electroquímico que luego se puede usar para sintetizar ATP por quimiosmosis . El P870 debe regenerarse (reducirse) para que esté disponible nuevamente para que un fotón llegue al centro de reacción y comience nuevamente el proceso. El hidrógeno molecular en el ambiente bacteriano es el donante de electrones habitual.
| ATP sintasa | |||
|---|---|---|---|
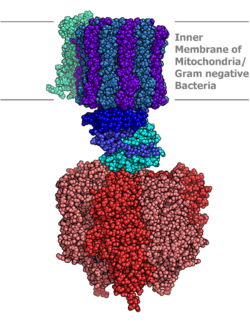
Modelo molecular de la ATP sintasa determinado por cristalografía de rayos X . El estator no se muestra aquí.
| |||
| Identificadores | |||
| Numero de EC | 7.1.2.2 | ||
| número CAS | 9000-83-3 | ||
| Bases de datos | |||
| IntEnz | Vista IntEnz | ||
| Brenda | BRENDA entrada | ||
| ExPASY | Vista de NiceZyme | ||
| KEGG | Entrada KEGG | ||
| MetaCyc | camino metabólico | ||
| PRIAM | perfil | ||
| Estructuras dePDB | RCSB PDB PDBePDBsum | ||
| Ontología de genes | AmiGO / QuickGO | ||
| |||
La ATP sintasa es una enzima que crea la molécula de almacenamiento de energía trifosfato de adenosina (ATP). El ATP es la "moneda de energía" más utilizada de las células para todos los organismos. Se forma a partir de difosfato de adenosina (ADP) y fosfato inorgánico (P i ). La reacción global catalizada por la ATP sintasa es:
- ADP + P i + H + out ⇌ ATP + H 2 O + H + in
La formación de ATP a partir de ADP y P i es desfavorable desde el punto de vista energético y normalmente se desarrollaría en sentido inverso. Para impulsar esta reacción, la ATP sintasa combina la síntesis de ATP durante la respiración celular con un gradiente electroquímico creado por la diferencia en la concentración de protones (H + ) a través de la membrana mitocondrial en eucariotaso la membrana plasmática en bacterias. Durante la fotosíntesis en las plantas, la ATP sintasa sintetiza el ATP utilizando un gradiente de protones creado en el lumen de los tilacoides a través de la membrana del tilacoide y en laEstroma de cloroplasto .
La ATP sintasa consta de dos subunidades principales, F O y F 1 , que tiene un mecanismo de motor rotativo que permite la producción de ATP. [1] [2] Debido a su subunidad giratoria, la ATP sintasa es una máquina molecular .
Nomenclatura [ editar ]
La fracción F 1 deriva su nombre del término "Fracción 1" y F O (escrita como una letra de subíndice "o", no "cero") deriva su nombre de ser la fracción de unión para la oligomicina , un tipo de antibiótico de origen natural. que es capaz de inhibir la unidad F O de la ATP sintasa. [3] [4] Estas regiones funcionales consisten en diferentes subunidades de proteínas; consulte las tablas. Esta enzima se utiliza en la síntesis de ATP a través de la respiración aeróbica.
Estructura y función [ editar ]
Ubicada dentro de la membrana tilacoide y la membrana mitocondrial interna , la ATP sintasa consta de dos regiones F O y F 1 . F O causa la rotación de F 1 y está formado por un anillo en C y las subunidades a, b, d, F6. F 1 está hecho de subunidades F 1 tiene una parte soluble en agua que puede hidrolizar el ATP. Por otra parte, F O tiene regiones principalmente hidrófobas. F O F 1 crea un camino para el movimiento de protones a través de la membrana. [7]
Región F 1 [ editar ]
La porción F 1 de la ATP sintasa es hidrófila y responsable de la hidrólisis del ATP. La unidad F 1 sobresale en el espacio de la matriz mitocondrial. Subunidades y Hacer un hexámero con 6 sitios de unión. Tres de ellos son catalíticamente inactivos y se unen a ADP.
Otras tres subunidades catalizan la síntesis de ATP. Las otras subunidades F 1 Son parte de un mecanismo de motor rotativo. la subunidad permite pasar por cambios conformacionales, es decir, estados cerrados, medio abiertos y abiertos permite que el ATP se vincule y libere una vez que se sintetice. La partícula F 1 es grande y se puede ver en el microscopio electrónico de transmisión por tinción negativa. [8] Estas son partículas de 9 nm de diámetro que salpican la membrana mitocondrial interna.
| Subunidad | Gen humano |
|---|---|
| alfa | ATP5A1 , ATPAF2 |
| beta | ATP5B , ATPAF1 , C16orf7 |
| gama | ATP5C1 |
| delta | ATP5D |
| épsilon | ATP5E |
F O región [ editar ]
F O es una proteína insoluble en agua con ocho subunidades y un anillo transmembrana. El anillo tiene una forma de tetrámero con una proteína helicoidal de bucle helicoidal que pasa por cambios conformacionales cuando está protonado y desprotonado, lo que empuja a las subunidades vecinas a rotar, lo que provoca el giro de F O que también afecta a la conformación de F 1 , lo que da como resultado el cambio de estados alfa y subunidades beta. La región F O de la ATP sintasa es un poro de protones que está incrustado en la membrana mitocondrial. Consiste en tres subunidades principales, a, byc, y (en humanos) seis subunidades adicionales, d , e , f ,g , F6 y 8 (o A6L). Se determinó un modelo atómico para la región F O de la levadura dimérica mediante crio-EM a una resolución global de 3,6 Å. [10] Esta parte de la enzima se encuentra en la membrana interna mitocondrial y acopla la translocación de protones a la rotación, lo que causa la síntesis de ATP en la región F 1 .
| Subunidad | Gen humano |
|---|---|
| una | MT-ATP6 , MT-ATP8 |
| segundo | ATP5F1 |
| do | ATP5G1 , ATP5G2 , ATP5G3 |
Modelo de unión [ editar ]
En los años sesenta y setenta, Paul Boyer , un profesor de UCLA , desarrolló la teoría del mecanismo de cambio vinculante o flip-flop, que postulaba que la síntesis de ATP depende de un cambio conformacional en la ATP sintasa generado por la rotación de la subunidad gamma. El grupo de investigación de John E. Walker , entonces en el Laboratorio de Biología Molecular MRC en Cambridge , cristalizó el dominio catalítico F 1 de la ATP sintasa. La estructura, en el momento en que se conoce la estructura proteica asimétrica más grande, indica que el modelo de catálisis rotatoria de Boyer era, en esencia, correcto. Para dilucidar esto, Boyer y Walker compartieron la mitad del Premio Nobel de Química de1997 .
La estructura cristalina de la F 1 mostraba subunidades alfa y beta alternas (3 de cada una), dispuestas como segmentos de una naranja alrededor de una subunidad gamma asimétrica giratoria. De acuerdo con el modelo actual de síntesis de ATP (conocido como el modelo catalítico alternativo), el potencial transmembrana creado por los cationes de protones (H +) suministrados por la cadena de transporte de electrones, impulsa los cationes de protones (H +) desde el espacio intermembrana a través de la membrana a través de la membrana. F Oregión de la ATP sintasa. Una porción de la F O (el anillo de las subunidades c ) gira cuando los protones pasan a través de la membrana. El anillo cestá fuertemente unido al tallo central asimétrico (que consiste principalmente en la subunidad gamma), lo que hace que gire dentro del alfa 3 beta 3 de F 1, lo que hace que los 3 sitios de unión a los nucleótidos catalíticos pasen por una serie de cambios conformacionales que conducen a la síntesis de ATP . Los principales F 1 subunidades se impide que gire en simpatía con el rotor central tallo por un tallo periférico que se une a la alfa 3 beta 3 a la porción no giratoria de F O . La estructura de la ATP sintasa intacta se conoce actualmente a baja resolución a partir de la crio-microscopía electrónica.(crio-EM) estudios del complejo. El modelo de la crio-EM de la ATP sintasa sugiere que el tallo periférica es una estructura flexible que se envuelve alrededor del complejo, ya que se une a F 1 a F O . En las condiciones adecuadas, la reacción de la enzima también se puede llevar a cabo en sentido inverso, con la hidrólisis de ATP conduciendo el bombeo de protones a través de la membrana.
El mecanismo de cambio de enlace involucra el sitio activo de un ciclo de una subunidad β entre tres estados. [11]En el estado "suelto", el ADP y el fosfato entran al sitio activo; en el diagrama adyacente, esto se muestra en rosa. Luego, la enzima experimenta un cambio en la forma y fuerza estas moléculas juntas, con el sitio activo en el estado "apretado" resultante (que se muestra en rojo) uniéndose a la molécula de ATP recién producida con una afinidad muy alta . Finalmente, el sitio activo regresa al estado abierto (naranja), liberando ATP y uniendo más ADP y fosfato, listo para el próximo ciclo de producción de ATP. [12]
Papel fisiológico [ editar ]
Al igual que otras enzimas, la actividad de F 1 F O ATP sintasa es reversible. Cantidades suficientemente grandes de ATP hacen que cree un gradiente de protones transmembrana , esto se usa para fermentar bacterias que no tienen una cadena de transporte de electrones, sino que hidrolizan el ATP para formar un gradiente de protones, que utilizan para impulsar los flagelos y el transporte de nutrientes en la célula.
En la respiración de bacterias en condiciones fisiológicas, la ATP sintasa, en general, se ejecuta en la dirección opuesta, creando ATP mientras se usa la fuerza motriz de protones creada por la cadena de transporte de electrones como fuente de energía. El proceso general de creación de energía de esta manera se denomina fosforilación oxidativa . El mismo proceso se lleva a cabo en las mitocondrias , donde la ATP sintasa se encuentra en la membrana mitocondrial interna y los proyectos de la parte F 1 en la matriz mitocondrial . El consumo de ATP por la ATP-sintasa bombea los cationes de protones a la matriz.
Evolución [ editar ]
Se cree que la evolución de la ATP sintasa ha sido modular, por lo que dos subunidades funcionalmente independientes se asociaron y obtuvieron nuevas funcionalidades. [13] [14] Esta asociación parece haber ocurrido al principio de la historia evolutiva, porque esencialmente la misma estructura y actividad de las enzimas ATP sintasas están presentes en todos los reinos de la vida. [13] La F-ATP sintasa muestra una gran similitud funcional y mecánica con la V-ATPasa . [15] Sin embargo, mientras que la F-ATP sintasa genera ATP utilizando un gradiente de protones, la V-ATPasa genera un gradiente de protones a expensas del ATP, generando valores de pH tan bajos como 1. [16]
La región F 1 también muestra una similitud significativa con las helicasas de ADN hexameric , y la región F Omuestra cierta similitud con H +
-Complejos de motor flagelar . [15] El hexámero α 3 β 3 de la región F 1 muestra una similitud estructural significativa con las helicasas de ADN hexaméricas; ambos forman un anillo con una simetría rotacional triple con un poro central. Ambos tienen roles que dependen de la rotación relativa de una macromolécula dentro del poro; las helicasas de ADN utilizan la forma helicoidal del ADN para impulsar su movimiento a lo largo de la molécula de ADN y para detectar el supercoldeo, mientras que el hexámero α 3 β 3utiliza los cambios conformacionales a través de la rotación de la subunidad γ para conducir una reacción enzimática. [17]
-Complejos de motor flagelar . [15] El hexámero α 3 β 3 de la región F 1 muestra una similitud estructural significativa con las helicasas de ADN hexaméricas; ambos forman un anillo con una simetría rotacional triple con un poro central. Ambos tienen roles que dependen de la rotación relativa de una macromolécula dentro del poro; las helicasas de ADN utilizan la forma helicoidal del ADN para impulsar su movimiento a lo largo de la molécula de ADN y para detectar el supercoldeo, mientras que el hexámero α 3 β 3utiliza los cambios conformacionales a través de la rotación de la subunidad γ para conducir una reacción enzimática. [17]
El H +
El motor de la partícula F O muestra una gran similitud funcional con el H +.
Motores que impulsan los flagelos. [15] Ambos presentan un anillo de muchas proteínas alfa-helicoidales pequeñas que giran en relación con las proteínas estacionarias cercanas, utilizando un H +
gradiente potencial como fuente de energía. Sin embargo, este enlace es tenue, ya que la estructura general de los motores flagelares es mucho más compleja que la de la partícula F O y el anillo con aproximadamente 30 proteínas rotativas es mucho más grande que las 10, 11 o 14 proteínas helicoidales en la F O complejo.
El motor de la partícula F O muestra una gran similitud funcional con el H +.
Motores que impulsan los flagelos. [15] Ambos presentan un anillo de muchas proteínas alfa-helicoidales pequeñas que giran en relación con las proteínas estacionarias cercanas, utilizando un H +
gradiente potencial como fuente de energía. Sin embargo, este enlace es tenue, ya que la estructura general de los motores flagelares es mucho más compleja que la de la partícula F O y el anillo con aproximadamente 30 proteínas rotativas es mucho más grande que las 10, 11 o 14 proteínas helicoidales en la F O complejo.
La teoría de la evolución modular para el origen de la ATP sintasa sugiere que dos subunidades con función independiente, una helicasa de ADN con actividad ATPasa y una H +
motor, fueron capaces de unirse, y la rotación del motor impulsó la actividad ATPasa de la helicasa en sentido inverso. [13] [17] Este complejo luego evolucionó a una mayor eficiencia y eventualmente se convirtió en las complejas sintasas ATP de hoy. Alternativamente, la ADN helicasa / H +.
complejo motor puede haber tenido H +
Actividad de la bomba con la actividad ATPasa de la helicasa que impulsa el H +.
Motor a la inversa. [13] Esto puede haber evolucionado para llevar a cabo la reacción inversa y actuar como una ATP sintasa. [14] [18] [19]
motor, fueron capaces de unirse, y la rotación del motor impulsó la actividad ATPasa de la helicasa en sentido inverso. [13] [17] Este complejo luego evolucionó a una mayor eficiencia y eventualmente se convirtió en las complejas sintasas ATP de hoy. Alternativamente, la ADN helicasa / H +.
complejo motor puede haber tenido H +
Actividad de la bomba con la actividad ATPasa de la helicasa que impulsa el H +.
Motor a la inversa. [13] Esto puede haber evolucionado para llevar a cabo la reacción inversa y actuar como una ATP sintasa. [14] [18] [19]
En diferentes especies [ editar ]
E. coli [ editar ]
La ATP sintasa de E. coli es la forma más simple conocida de la ATP sintasa, con 8 tipos de subunidades diferentes. [20]
Levadura [ editar ]
La levadura ATP sintasa es una de las ATP sintasas eucarióticas mejor estudiadas; y se han identificado cinco subunidades F 1 , ocho subunidades F O y siete proteínas asociadas. [7] La mayoría de estas proteínas tienen homólogos en otros eucariotas. [21] [22] [23] [24]
Planta [ editar ]
En plantas, la ATP sintasa también está presente en los cloroplastos (CF 1 F O -ATP sintasa). La enzima está integrada en la membrana tilacoide ; La parte CF 1 se adhiere al estroma , donde se producen las reacciones oscuras de la fotosíntesis (también llamadas reacciones independientes de la luz o el ciclo de Calvin ) y la síntesis de ATP. La estructura general y el mecanismo catalítico de la ATP sintasa de cloroplasto son casi los mismos que los de la enzima bacteriana. Sin embargo, en los cloroplastos, la fuerza motriz del protón se genera no por la cadena de transporte de electrones respiratorios sino por las proteínas fotosintéticas primarias.
Bovino [ editar ]
La ATP sintasa aislada de las mitocondrias del corazón bovino ( Bos taurus ) es, en términos de bioquímica y estructura, la ATP sintasa mejor caracterizada. El corazón de res se utiliza como fuente de la enzima debido a la alta concentración de mitocondrias en el músculo cardíaco. [25] [26] [27]
Humano [ editar ]
Genes humanos que codifican componentes de las sintasas ATP:











No hay comentarios:
Publicar un comentario