Replicación de ADN
El modelo líder de replicación de cpDNA.
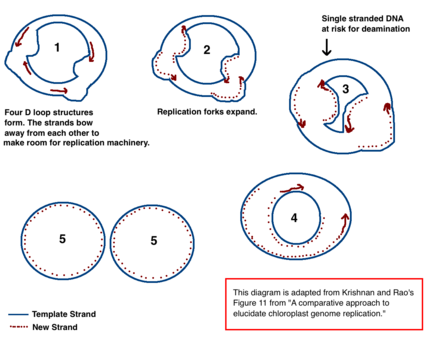
El mecanismo para la replicación del ADN del cloroplasto (ADNcp) no se ha determinado de manera concluyente, pero se han propuesto dos modelos principales. Los científicos han intentado observar la replicación del cloroplasto a través de microscopía electrónica desde la década de 1970. [71] [72] Los resultados de los experimentos de microscopía llevaron a la idea de que el ADN del cloroplasto se replica utilizando un bucle de doble desplazamiento (bucle D). A medida que el bucle D se mueve a través del ADN circular, adopta una forma intermedia theta, también conocida como un intermediario de replicación de Cairns, y completa la replicación con un mecanismo de círculo rodante. [71] [73]La transcripción se inicia en puntos específicos de origen. Se abren múltiples horquillas de replicación, lo que permite que la maquinaria de replicación transcriba el ADN. A medida que continúa la replicación, las horquillas crecen y eventualmente convergen. Las nuevas estructuras de cpDNA se separan, creando cromosomas hijas de cpDNA.
Además de los primeros experimentos de microscopía, este modelo también está respaldado por las cantidades de desaminación observadas en cpDNA. [71] La desaminación ocurre cuando se pierde un grupo amino y es una mutación que a menudo produce cambios en la base. Cuando la adenina se desamina, se convierte en hipoxantina . La hipoxantina se puede unir a la citosina, y cuando el par de bases XC se replica, se convierte en un GC (por lo tanto, un cambio de base A → G). [74]
Desminación
En cpDNA, hay varios gradientes de desaminación A → G. El ADN se vuelve susceptible a los eventos de desaminación cuando es monocatenario. Cuando se forman las horquillas de replicación, la hebra que no se copia es de una sola hebra y, por lo tanto, está en riesgo de desaminación A → G. Por lo tanto, los gradientes en la desaminación indican que las horquillas de replicación estaban más probablemente presentes y la dirección en la que se abrieron inicialmente (el gradiente más alto es más probable que esté más cerca del sitio de inicio debido a que fue de una sola hebra por el mayor tiempo). [71]Este mecanismo sigue siendo la teoría principal hoy en día; sin embargo, una segunda teoría sugiere que la mayoría de los ADNcp es en realidad lineal y se replica a través de una recombinación homóloga. Además, sostiene que solo una minoría del material genético se mantiene en cromosomas circulares, mientras que el resto está en estructuras ramificadas, lineales u otras complejas. [71] [73]
Modelo alternativo de replicación.
Uno de los modelos competidores para la replicación del ADNcp afirma que la mayoría de los ADNcp es lineal y participa en estructuras de replicación y recombinación homólogas similares a las estructuras de ADN lineal y circular del bacteriófago T4 . [73] [75] Se ha establecido que algunas plantas tienen ADNcp lineal, como el maíz, y que más especies aún contienen estructuras complejas que los científicos aún no comprenden. [73] Cuando se realizaron los experimentos originales en cpDNA, los científicos notaron estructuras lineales; sin embargo, atribuyeron estas formas lineales a círculos rotos. [73]Si las estructuras ramificadas y complejas que se ven en los experimentos con ADNcp son reales y no son artefactos de ADN circular concatenado o círculos rotos, entonces el mecanismo de replicación del bucle D es insuficiente para explicar cómo se replicarían esas estructuras. [73] Al mismo tiempo, la recombinación homóloga no expande los múltiples gradientes A -> G observados en los plastomas. [71] Debido a la falta de explicación del gradiente de desaminación, así como a las numerosas especies de plantas que se ha demostrado que tienen cpDNA circular, la teoría predominante continúa sosteniendo que la mayoría de cpDNA es circular y lo más probable es que se replique a través de un mecanismo de D loop.
Contenido de genes y síntesis de proteínas.
El genoma del cloroplasto incluye más comúnmente alrededor de 100 genes [24] [41] que codifican una variedad de cosas, principalmente relacionadas con la línea de proteínas y la fotosíntesis . Como en los procariotas , los genes en el ADN del cloroplasto se organizan en operones . [24] A diferencia de las moléculas de ADN procarióticas , las moléculas de ADN de cloroplasto contienen intrones ( los ADN mitocondriales de las plantas también, pero no los ADNmt humanos). [76]
Entre las plantas terrestres, el contenido del genoma del cloroplasto es bastante similar. [42]
Reducción del genoma del cloroplasto y transferencia de genes.
Con el tiempo, muchas partes del genoma del cloroplasto se transfirieron al genoma nuclear del huésped, [40] [41] [77] un proceso llamado transferencia de genes endosimbióticos . Como resultado, el genoma del cloroplasto está muy reducido en comparación con el de las cianobacterias de vida libre. Los cloroplastos pueden contener entre 60 y 100 genes, mientras que las cianobacterias suelen tener más de 1500 genes en su genoma. [78] Recientemente, se encontró un plástido sin un genoma, lo que demuestra que los cloroplastos pueden perder su genoma durante el proceso de transferencia de genes endosimbióticos. [79]
La transferencia de genes endosbióticos es lo que sabemos acerca de los cloroplastos perdidos en muchos linajes CASH. Incluso si un cloroplasto se pierde finalmente, los genes que donó al núcleo del huésped anterior persisten, lo que proporciona evidencia de la existencia del cloroplasto perdido. Por ejemplo, mientras que las diatomeas (un heterocontofito ) ahora tienen un cloroplasto derivado de algas rojas , la presencia de muchos genes de algas verdes en el núcleo de diatomeas evidencia que el ancestro de diatomeas tuvo un cloroplasto derivado de algas verdes en algún momento, que posteriormente fue reemplazado por cloroplasto rojo. [44]
En las plantas terrestres, entre el 11 y el 14% del ADN en sus núcleos se remonta al cloroplasto, [39] hasta el 18% en Arabidopsis , que corresponde a unos 4.500 genes codificantes de proteínas. [80] Ha habido algunas transferencias recientes de genes del ADN del cloroplasto al genoma nuclear en plantas terrestres. [41]
De las aproximadamente 3000 proteínas encontradas en los cloroplastos, alrededor del 95% de ellas están codificadas por genes nucleares. Muchos de los complejos de proteínas del cloroplasto consisten en subunidades del genoma del cloroplasto y del genoma nuclear del huésped. Como resultado, la síntesis de proteínas debe coordinarse entre el cloroplasto y el núcleo. El cloroplasto se encuentra principalmente bajo control nuclear, aunque los cloroplastos también pueden emitir señales que regulan la expresión génica en el núcleo, lo que se denomina señalización retrógrada . [81]
Síntesis de proteínas
La síntesis de proteínas dentro de los cloroplastos se basa en dos ARN polimerasas . Uno está codificado por el ADN del cloroplasto, el otro es de origen nuclear . Las dos ARN polimerasas pueden reconocer y unirse a diferentes tipos de promotores dentro del genoma del cloroplasto. [82] Los ribosomas en los cloroplastos son similares a los ribosomas bacterianos. [83]
Orientación e importación de proteínas
Debido a que tantos genes de cloroplastos se han trasladado al núcleo, muchas proteínas que originalmente se habrían traducido en el cloroplasto ahora se sintetizan en el citoplasma de la célula vegetal. Estas proteínas deben dirigirse de nuevo al cloroplasto e importarse a través de al menos dos membranas de cloroplasto. [84]
Curiosamente, alrededor de la mitad de los productos proteicos de los genes transferidos ni siquiera están dirigidos al cloroplasto. Muchos se convirtieron en exaptaciones , asumiendo nuevas funciones como participar en la división celular , enrutamiento de proteínas e incluso resistencia a enfermedades . Unos pocos genes de cloroplastos encontraron nuevos hogares en el genoma mitocondrial, la mayoría se convirtieron en pseudogenesno funcionales , aunque algunos genes de ARNt aún funcionan en la mitocondria . [78] Algunos productos de proteína de ADN de cloroplasto transferidos se dirigen a la vía secretora [78] a través de muchos plastos secundariosestán limitados por una membrana más externa derivada de la membrana celular del huésped y, por lo tanto, topológicamente fuera de la célula, porque para alcanzar el cloroplasto desde el citosol , debe cruzar la membrana celular , como si se dirigiera al espacio extracelular . En esos casos, las proteínas dirigidas a los cloroplastos viajan inicialmente a lo largo de la vía secretora. [32]
Debido a que la célula que adquirió un cloroplasto ya tenía mitocondrias (y peroxisomas , y una membrana celular para la secreción), el nuevo huésped del cloroplasto tuvo que desarrollar un sistema único de direccionamiento de proteínas para evitar que las proteínas del cloroplasto se enviaran al orgánulo equivocado . [84]
En la mayoría, pero no en todos los casos, las proteínas de cloroplasto codificadas en el núcleo se traducen con un péptido de tránsito escindible que se agrega al extremo N de la proteína precursora. A veces, la secuencia de tránsito se encuentra en el extremo C de la proteína, [86] o dentro de la parte funcional de la proteína. [84]
Proteínas de transporte y translocones de membrana.
Después de que un polipéptido de cloroplasto se sintetiza en un ribosoma en el citosol , una enzima específica de las proteínas de cloroplasto [87] fosforila , o agrega un grupo de fosfato a muchos (pero no a todos) de ellos en sus secuencias de tránsito. [84] La fosforilación ayuda a muchas proteínas a unirse al polipéptido, evitando que se repliegue prematuramente. [84] Esto es importante porque evita que las proteínas cloroplásticas asuman su forma activa y realicen sus funciones de cloroplasto en el lugar equivocado: el citosol . [88] [89]Al mismo tiempo, tienen que mantener la forma suficiente para que puedan ser reconocidos por el cloroplasto. [88] Estas proteínas también ayudan al polipéptido a importarse en el cloroplasto. [84]
Desde aquí, las proteínas de cloroplastos con destino a la estroma deben pasar a través de dos complejos de la proteína complejos TOC , o t ranslocon en la o uter c hloroplast membrana , y la translocon TIC , o t ranslocon en el i nner c hloroplast membrana translocon . [84] Las cadenas de polipéptidos de cloroplastos a menudo viajan a través de los dos complejos al mismo tiempo, pero el complejo TIC también puede recuperar preproteínas perdidas en el espacio intermembrana .

![Los dos extremos de un polipéptido se denominan el término N, o extremo amino, y el extremo C, o extremo carboxilo. [85] Este polipéptido tiene cuatro aminoácidos unidos entre sí. A la izquierda está el término N, con su grupo amino (H2N) en verde. El término C azul, con su grupo carboxilo (CO2H) está a la derecha.](https://upload.wikimedia.org/wikipedia/commons/thumb/c/c0/Tetrapeptide_structural_formulae.svg/350px-Tetrapeptide_structural_formulae.svg.png)
No hay comentarios:
Publicar un comentario