El conteo de bacterioplancton es la estimación de la abundancia de bacterioplancton en un cuerpo de agua específico, que es una información útil para los microbiólogos marinos. Se han desarrollado varias metodologías de conteo a lo largo de los años para determinar el número presente en el agua que se observa. Los métodos utilizados para el recuento de bacterioplancton incluyen microscopía de epifluorescencia , citometría de flujo , medidas de productividad a través de la frecuencia de células en división (FDC), incorporación de timina e incorporación de leucina .
Factores como la salinidad , la temperatura, la latitud , diversos niveles de nutrientes, el movimiento del agua y la presencia de otros organismos pueden afectar la enumeración de bacterioplancton. [1] [2] [3] [4] [5] Los cambios en estos factores afectan el recuento de bacterioplancton, causando que varíe según el cuerpo de agua, la ubicación, la distancia desde la costa y la estación. [6] [7] [8]
El recuento de bacterioplancton generalmente se expresa en células por ml (células ml- 1 ).
Usos [ editar ]
Para comprender la microbiología marina y el ecosistema acuático, los recuentos de bacterioplancton pueden ser útiles. La observación del número de bacterioplancton puede proporcionar más información en lo siguiente:
- Procesos implicados en diversos ciclos de nutrientes en los sistemas acuáticos [9] [10]
- Para la productividad acuática [11]
- Para determinar cambios ambientales, especialmente extremos [12]
- Composición de nutrientes en ecosistemas acuáticos [16]
- Abundancia y condiciones de otros organismos acuáticos (es decir, camarones) [17]
Epifluorescencia microscopía [ editar ]
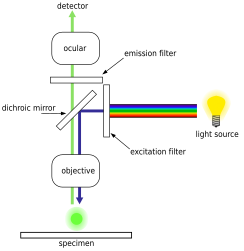
La microscopía de epifluorescencia es una técnica avanzada de microscopio óptico que se basa en el uso de tintes fluorescentes que se unen a marcadores biológicos específicos, que luego emiten un espectro de emisión distintivo que se identifica a través de la lente. Los tintes fluorescentes incluyen DAPI , naranja de acridina , SYBR Green 1 y YO-PRO-1, todos los cuales son capaces de teñir las estructuras de ADN y ARN en muestras biológicas como bacterias y virus. [18] [19] [20] [21]Sin embargo, la tinción de ADN se utiliza principalmente para la identificación de células bacterianas. Con la microscopía de epifluorescencia moderna, el estándar de la industria para estimar y contar las cantidades de células bacterianas es mediante el uso de una tinción DAPI . [22] Esta técnica se puede realizar para muestras de una amplia gama de entornos y ubicaciones, como agua de mar, diversas fuentes de agua dulce, suelos y sedimentos. [22]
Técnica de enumeración [ editar ]
En un experimento estándar, las muestras bacterianas preparadas se colocan en portaobjetos de recuento y luego se observan bajo un microscopio de epifluorescencia. La ampliación se establece en un nivel donde las unidades cuadradas de 0.1 X 0.1 mm en la diapositiva de conteo son claramente visibles. [23] Para cuantificar las bacterias, las células se cuentan en 5-30 campos de visión de unidades cuadradas aleatorias y se tabula un recuento promedio de bacterias por campo. [22] Este valor se extrapola luego para estimar el recuento total de células bacterianas por ml al determinar el número total de campos de visión en el área de deposición de portaobjetos y multiplicarlo por el recuento promedio de bacterias por unidad de recuento. [23]
Fiabilidad [ editar ]
Para enumerar las cantidades de células bacterianas, solo se cuentan físicamente pequeñas porciones de bacterias en una muestra por razones logísticas, sobre las cuales las abundancias totales se estiman mediante extrapolación. Los valores medios se utilizan para la comparación entre muestras. Sin embargo, la precisión de esta técnica, donde se usa la tabulación de solo un pequeño subconjunto para estimar las cantidades totales de abundancia, ha sido cuestionada. [22] Principalmente, se ha demostrado que la distribución de las células bacterianas en el conteo de las diapositivas puede ser desigual e inconsistente. [22] Además, para obtener una estimación legítima de los recuentos de bacterias mediante el uso de esta técnica, se ha sugerido que se deben medir más de 350 células individuales, desde 20 campos de visión. [22] Esto no solo puede llevar mucho tiempo, sino que es difícil de lograr en ciertas muestras.
Citometría de Flujo [ editar ]
El análisis de citometría de flujo (o citometría de flujo ) es un procedimiento común en muchas aplicaciones clínicas. Sin embargo, a pesar de su descubrimiento hace más de tres décadas, su adopción por la ecología microbiana acuática en la enumeración de bacterioplancton, ha sido relativamente lenta. [24] Su uso aún está por superar la microscopía de epifluorescencia . [25] A pesar de que ambas técnicas de estimación de la abundancia son relativamente precisas, la citometría de flujo es menos propensa al error humano, más precisa, tiene una resolución más alta y es capaz de examinar decenas de miles de células en cuestión de minutos. [24] La citometría de flujo también puede proporcionar información sobre el tamaño, la actividad y la morfología de las células, además de la abundancia de células. [26]
La citometría de flujo se puede utilizar para distinguir y cuantificar el bacterioplancton tanto fotosintético como no fotosintético. [26] La cuantificación de procariotas fotosintéticos como las cianobacterias y las algas picoeucariota es posible gracias a la capacidad de los pigmentos fotosintéticos para emitir fluorescencia. [27] Por ejemplo, la diferente formación de pigmentos fotosintéticos en los dos procariotas fotosintéticos principales, el proclorococo y el sinechococo , permiten su distinción. [28] [29] [30] Prochlorococcus contiene divinil-clorofilas a y bque muestran únicamente fluorescencia roja bajo excitación por luz azul o UV, mientras que synechococcus emite fluorescencia naranja y roja; Naranja de las ficobilinas y rojo de la clorofila . Además de la fluorescencia, prochlorococcus y synechococus son de tamaños significativamente diferentes y por lo tanto ofrecen diferentes señales de dispersión en el análisis de citometría de flujo. Esto ayuda aún más en su diferenciación. [31] La cuantificación de prochlorococcus se considera un gran avance, ya que casi solo ha sido posible a través de la citometría de flujo. Esto se debe a la incapacidad de la microscopía de epifluorescencia para detectar la autofluorescencia baja en clorofila presente en el proclorococo. [26]
Además del bacterioplancton fotosintético, el bacterioplancton no fotosintético también se puede enumerar mediante citometría de flujo. Esto se hace a través de la tinción de ADN o vacuola de alimentos. [27] La citometría de flujo ha sido especialmente exitosa en la diferenciación de prochloroccocus de bacterias heterótrofas, cuyos recuentos se confundieron inicialmente debido a su tamaño similar.
El uso de la microscopía de epifluorescencia sobre la citometría de flujo en muchos laboratorios de ecología microbiana puede atribuirse a una serie de factores económicos y prácticos. Primero, el uso de citómetros de flujo comerciales requiere la experiencia de un técnico rigurosamente capacitado. En segundo lugar, los citómetros de flujo son bastante caros en comparación con los aparatos de microscopía de epi-fluorescencia. En tercer lugar, muchos citómetros de flujo están diseñados para examinar las células sanguíneas; Las bacterias oceánicas son relativamente pequeñas y, por lo tanto, se acercan al límite de resolución en muchos citómetros de flujo comerciales. [32]
Proceso de enumeración [ editar ]
La cuantificación por citometría de flujo de bacterioplancton implica cuatro pasos: fijación , tinción , procesamiento de datos e interpretación de datos.
Fijación [ editar ]
La fijación se realiza no solo para preservar la muestra, sino también para aumentar la permeabilidad de las células a las manchas. [24] Sin embargo, la mayoría de los agentes de fijación comunes tienen la capacidad de alterar las células al cambiar ciertos aspectos, como el tamaño, la forma en que se dispersa la luz, la autofluorescencia y los ácidos nucleicos . Esto es problemático, ya que la distinción citométrica de flujo de las células se basa en estas cualidades. Algunos fijadores también conducen a la pérdida completa de células. [24]Actualmente, algunos de los agentes utilizados en el proceso de fijación incluyen dos variaciones de formaldehído (formalina y paraformaldehído), etanol al 70%, glutaraldehído y TCA. [33]Se presume que el mejor agente de fijación de proteínas y ácidos nucleicos es el paraformaldehído debido a su capacidad para ingresar rápidamente a las células. [24]
Tinción [ editar ]
En la citometría de flujo, la tinción permite distinguir el bacterioplancton de las partículas no bacterianas. Implica la incubación de la muestra en la amplia gama de fluorocromos , como los colorantes UV-excitados ( DAPI y Hoechst 33342) y los colorantes de ácidos nucleicos excitados con luz azul (TO-PRO-1, TOTO-1, SYBR Green I). [31] Durante mucho tiempo, los citómetros de flujo utilizaron tintes con radiación UV para examinar el bacterioplanctonque podrían usarse en citómetros de flujo de bajo costo con sensibilidad limitada, o en citómetros de flujo costosos con la alta sensibilidad necesaria para distinguir las bacterias heterótrofas de los autótrofos. La introducción de colorantes azul excitados, como SYBR Green I, permitió el análisis citométrico de flujo de alta calidad del bacterioplancton en citómetros de flujo de alta sensibilidad y bajo costo. [31]
Los tiempos de incubación para una tinción óptima varían de un compuesto a otro. Los tintes con radiación UV pueden requerir una hora o más, mientras que los tintes con luz azul requieren solo 15 minutos. [24]
La tinción puede ir acompañada de tampones como Triton X-100 que hacen que las células sean más permeables a las manchas. Se utilizan especialmente en tintes impermeables a las células como TO-PRO-1. Los tampones también se utilizan para diluir tintes sensibles a la fuerza iónica, como Picogreen, YO-PRO-1 y YOYO-1. Sin embargo, el uso de tampones puede ser perjudicial para las células, ya que los tampones como Triton-X-100 no solo pueden extinguir la fluorescencia de la clorofila, sino que también crean una fluorescencia de fondo no deseada. Esto puede aumentar la dificultad de distinguir entre bacterias heterótrofas y procariotas autótrofos . [24]
Contando [ editar ]
En un análisis de citometría de flujo, más de 200 células pasan frente a un rayo láser o una lámpara de mercurio cada segundo, una célula a la vez. Los fotomultiplicadores reúnen la cantidad de luz que dispersa cada partícula y la fluorescencia emitida durante la excitación. Esta información es luego internalizada e interpretada por el sistema como un evento. Sin embargo, a pesar de la capacidad de los citómetros de flujo para contar células con muy poco esfuerzo, la mayoría no tiene forma de determinar la concentración real de las células. Esto se puede determinar a través de una variedad de métodos que incluyen el uso de perlas de referencia cuya cantidad está predeterminada (ayuda a determinar la proporción de bacterias a perlas), mediciones de peso antes y después del experimento y calibración diaria del flujo. [24]
Una gran ventaja de los citómetros de flujo es su capacidad para identificar diferentes poblaciones de bacterioplanton. Esta discriminación se realiza a través del análisis de cuatro factores; dispersión de luz , fluorescencia verde, fluorescencia azul y fluorescencia roja. El análisis de dispersión de la luz es inadecuado solo y, a menudo, se examina junto con la fluorescencia por varias razones; Primero, el agua de mar contiene muchas partículas que dispersan la luz como bacterias. Segundo, los tamaños de muchas bacterias oceánicas se acercan al límite de resolución. La cantidad de luz dispersada por las células se determina no solo por el tamaño de las células, sino también por la estructura interna, el índice de refracción, la forma y la orientación de las partículas. La luz dispersada se clasifica en dispersión hacia adelante(FSC) o dispersión lateral (SSC). El primero se ha asociado con el volumen y la masa celular, mientras que el último se ha asociado con el índice de refracción, contenido y granularidad de las células [24]
Cuando las concentraciones de células son superiores a 2,5 × 10 6 células por ml, la probabilidad de que más de una vez la célula pase cerca y se registre como un solo evento se magnifica. Esto se conoce como coincidencia y se puede evitar fácilmente diluyendo la muestra de antemano [31]
Medidas de productividad [ editar ]
La frecuencia de las células en división [ editar ]
La frecuencia de las células en división (FDC) es un método utilizado para predecir la tasa de crecimiento promedio de una comunidad bacteriana heterótrofa acuática. [34] El método utiliza la división celular, específicamente la formación de septo , como un proxy para la tasa de crecimiento. [34] Las células se consideran divididas, cuando se observan cavidades entre células individuales ( invaginación ) bajo microscopía de epifluorescencia . [34] FDC se basa en el supuesto de que existe una relación entre la proporción de células que se dividen actualmente y la tasa de crecimiento en una comunidad bacteriana. [35]
De incorporación de timidina [ editar ]
La incorporación de timidina es uno de los métodos más utilizados para estimar el crecimiento bacteriano. [36] La timidina es un precursor del ADN , y la síntesis de ADN se puede medir mediante la incorporación de timidina tritiada en ácido tricloroacético (TCA), material insoluble a través de vías de recuperación . [37] La incorporación de timidina mide el crecimiento en función de las tasas de síntesis de ADN, asumiendo que solo las células en crecimiento pueden incorporar la timidina radiactiva para sintetizar el ADN. [38]
Las debilidades de este procedimiento incluyen el marcado de otras moléculas además del ADN cuando se agrega timidina tritiada a una muestra. [36] En casos de limitación de carbono, la timidina también se puede usar como fuente de carbono en lugar de como un precursor de ADN. [36] Los resultados de los experimentos de incorporación de timidina pueden ser confusos cuando se desconoce la proporción de timidina incorporada en el ADN en comparación con otras moléculas. [36]
Leucina incorporación [ editar ]
La incorporación de leucina se usa como una medida de la síntesis de proteínas en comunidades de bacterias acuáticas. [39] La leucina radiomarcada se agrega a las muestras y se determina su acumulación en partes de la célula insolubles en ácido tricloroacético (CA) calientes. [39] Las muestras se recolectan en un filtro de membrana. [39] La proteína leucina es absorbida por más del 50% de las poblaciones de bacterias acuáticas, y la incorporación de leucina se puede usar para estimar la utilización de nitrógeno en la comunidad bacteriana. [39]
La dinámica de sucesión de temporada marinos [ editar ]
Como las poblaciones bacterianas tienen metabolismos y preferencias de recursos únicos, el uso de análisis de series de tiempo de composiciones bacterianas de alta resolución permite la identificación de patrones en la sucesión bacteriana estacional. [40] Las diferencias en las composiciones de la comunidad bacteriana dan lugar a permutaciones particulares de las interacciones bacterianas interespecies con el plancton fotosintético, los pastores protistas y los fagos, lo que afecta la dinámica de la estacionalidad. Se ha demostrado que los métodos estadísticos utilizados para verificar los patrones en la dinámica y composición de la población son replicables durante algunos años, y los factores ambientales sirven como factores predictivos de estos patrones temporales. [41]La mayor parte de la actividad de investigación para el bacterioplancton se produce actualmente en las aguas templadas del hemisferio norte desde 30 ° N hasta el Círculo Ártico a 66 ° N. [40]
Sucesión estacional en regiones templadas [ editar ]
Como las sucesiones estacionales de las poblaciones de fitoplancton siguen un patrón recurrente consistente, la dinámica bacteriana y la sucesión de fitoplancton pueden correlacionarse. [40] En general, los cambios estacionales en la composición bacteriana siguen a los cambios en la temperatura y la clorofila a , mientras que la disponibilidad de nutrientes limita las tasas de crecimiento de bacterioplancton. [42] [43] [44] [45] [6] [46] Durante la mezcla de la columna de agua a fines del otoño / invierno, los nutrientes traídos a la superficie inician una clara floración de primavera de diatomeas seguida de dinoflagelados. [40] Después de la floración de primavera.La producción y el crecimiento de bacterias se elevan debido a la liberación de materia orgánica disuelta (DOM) a partir de la desintegración del fitoplancton. [47] [48] En esta etapa temprana de sucesión, los miembros de la clase Flavobacteria (Bacteroidetes) son típicamente los componentes dominantes de la comunidad bacteriana. [49] [50]El análisis del genoma y la meta-transcriptómica han descubierto la presencia de bacterias que contienen múltiples enzimas hidrolíticas que facilitan la degradación y asimilación de la DOM. [51] [52] [53] [54] Durante las floraciones primaverales, algunos miembros del clado Roseobacter ( Alphaproteobacteria ) y algunosLas gammaproteobacterias se asocian generalmente con la degradación de DOM. [48] [49] A medida que aumentan las temperaturas y se agotan los nutrientes de la floración de primavera, crecen fitoplancton y cianobacterias más pequeñas en las aguas ahora oligotróficas. [40]
A medida que las aguas se estratifican en verano, Roseobacter, SAR86 (Gammaproteobacteria) y SAR11 (Alphaproteobacteria) clados de bacterias aumentan en abundancia. [55] [56] Las floraciones frecuentes de diatomeas y dinoflagelados de otoño se correlacionan con aportes de nutrientes suplementarios y el muestreo de alta frecuencia en el Mar Báltico encontró que en otoño, las Actinobacterias generalmente aumentan seguidas de diferentes Flavobacterias específicas de otoño , SAR11 y Planctomicetos . [49]
En el mar Mediterráneo , la mezcla en invierno profundo permite a los miembros del clado SAR11 lograr una mayor diversidad a medida que las poblaciones oligotróficas que una vez dominaron durante la estratificación de verano mueren lentamente. [57] Entre las arqueas en el Mar Mediterráneo, las poblaciones de ThaumarchaeotaMarine Group I (MGI) y Euryarchaeota Marine Group II (MGII.B) se hicieron dominantes en invierno. [58] Mientras que en el Mar Báltico , la mezcla de invierno trae a la superficie a las poblaciones de Epsilon-proteobacterias y arqueas de su hábitat profundo.


No hay comentarios:
Publicar un comentario