En enzimología , una [acil-portador-proteína] fosfodiesterasa ( EC 3.1.4.14 ) es una enzima que cataliza la reacción química
- holo [acil-carrier-proteína] + H 2 O 4'-fosfopanteteína + apo- [proteína portadora de acilo]
Así, los dos sustratos de esta enzima son [holo-acil-carrier-proteína [[]]] y H 2 O , mientras que sus dos productos son 4 '-fosfopanteteína y [[apo- [acil-carrier-proteína]]] .
Esta enzima pertenece a la familia de las hidrolasas , específicamente las que actúan sobre los enlaces de diéster fosfórico . El nombre sistemático de esta clase de enzimas es holo- [acyl-carrier-protein] 4'-pantetheine-phosphohydrolase . Otros nombres de uso común incluyen hidrolinasa ACP , fosfodiesterasa ACP , AcpH y [acil-carrier-protein] 4'-pantetheine-phosphohydrolase . Esta enzima participa en pantotenato y biosíntesis de coa.
Estudios estructurales [ editar ]
A finales de 2007, se han resuelto dos estructuras para esta clase de enzimas, con los códigos de acceso PDB 1T5B y 1TIK .
En enzimología , una [acil-portador-proteína] S-acetiltransferasa ( EC 2.3.1.38 ) es una enzima que cataliza la reacción química
- acetil-CoA + [proteína portadora de acilo] CoA + acetil- [proteína portadora de acilo]
Por lo tanto, los dos sustratos de esta enzima son acetil-CoA y proteína portadora de acilo , mientras que sus dos productos son CoA y proteína portadora de acetil-acilo.
Esta enzima pertenece a la familia de las transferasas , específicamente aquellos grupos de transferencia de aciltransferasas que no son grupos aminoacilo. El nombre sistemático de esta clase de enzimas es acetil-CoA: [acil-carrier-protein] S-acetyltransferase . Otros nombres de uso común incluyen acetil coenzima A-acil-carrier-protein transacylase , Acetyl CoA: transacylase ACP , [acyl-carrier-protein] acetyltransferase , [ACP] acetyltransferase y ACAT . Esta enzima participa en la biosíntesis de ácidos grasos .
En enzimología , una [acil-carrier-protein] S-malonyltransferase ( EC 2.3.1.39 ) es una enzima que cataliza la reacción química
- proteína portadora de malonil-CoA + acilo ⇌ CoA + malonil- [proteína portadora de acilo]
Por lo tanto, los dos sustratos de esta enzima son malonil-CoA y proteína transportadora de acilo , mientras que sus dos productos son CoA y malonil-acil-proteína transportadora. Esta enzima pertenece a la familia de las transferasas , específicamente aquellos grupos de transferencia de aciltransferasas que no son grupos aminoacilo. Esta enzima participa en la biosíntesis de ácidos grasos .
Nomenclatura [ editar ]
El nombre sistemático de esta clase de enzimas es malonil-CoA: [acil-carrier-protein] S-malonyltransferase. Otros nombres de uso común incluyen malonil coenzima A-acil portador de proteína transacilasa,
- [proteína portadora de acilo] maloniltransferasa,
- FabD,
- malonil transacilasa,
- malonil transferasa,
- proteína transportadora de malonil-CoA-acilo transacilasa,
- malonil-CoA: transacilasa ACP,
- malonil-CoA: AcpM transacilasa,
- malonil-CoA: proteína transportadora de acilo transacilasa,
- MAT, y
- MCAT.
Estructura [ editar ]
Las estructuras cristalinas de FabD de E. coli [1] y Streptomyces coelicolor [2] son conocidas y proporcionan una gran comprensión del mecanismo catalítico de FabD. En E. coli , FABD participa principalmente en la vía de FAS. Sin embargo, en Streptomyces coelicolor , FabD participa en las vías FAS y policétido sintasa. En ambos casos, las estructuras y los sitios activos son muy similares.
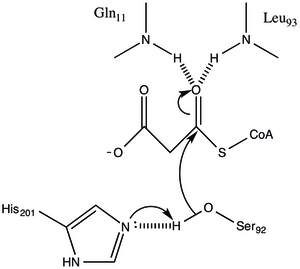
La proteína tiene una arquitectura de tipo α / β, pero el pliegue es único. el sitio activo inferido de la ubicación del Ser92 catalítico contiene un codo nucleofílico típico como se observa en las hidrolasas α / β. [1] La serina 92 está unida por hidrógeno a His 201 de manera similar a varias seridirdolasas. sin embargo, en lugar del ácido carboxílico que se encuentra típicamente en las tríadas catalíticas, el carbonilo de la cadena principal de Gln 250 sirve como un aceptor de enlace de hidrógeno en una interacción con His 201. [1] Otros dos residuos, Arg-117 y Glu-11 también se encuentran en el sitio activo, pero su función no está clara.
Función [ editar ]
La vía sintética de ácidos grasos es la ruta principal para la producción de cadenas de acilo de fosfolípidos de membrana en bacterias y plantas. [3] La secuencia de reacción se lleva a cabo por una serie de proteínas solubles individuales que están codificadas por un gen discreto, y los intermedios de la ruta se transportan entre las enzimas. [3] Malony-CoA: ACP Transacylase (FabD) es una de esas proteínas solubles individuales y cataliza la siguiente reacción:
- proteína portadora de malonil-CoA + acilo ⇌ CoA + malonil- [proteína portadora de acilo]
La transferencia de malonato a la proteína portadora de acilo (ACP) convierte los grupos acilo en formas de tioéster que son características de los intermedios de acilo en la síntesis de ácidos grasos y que son estrictamente necesarios para las reacciones de condensación catalizadas por la sintetasa de β-cetoacil-ACP. [4]
Mecanismo [ editar ]
Malonyl-CoA: ACP Transacylase utiliza un mecanismo cinético de ping-pong con un éster de malonía unido como el intermedio acilo unido a un residuo de serina que reside dentro de un pentapéptido GHSLG. [5] FabD primero se une al malonil-CoA, el resto de malonilo se transfiere luego al sitio activo Ser 92, y el CoA se libera de la enzima. A continuación, el ACP se une y el resto de malonilo se transfiere al sulfhidrilo terminal del grupo protésico de ACP. Esta reacción es fácilmente reversible. [3] [6]
Relevancia industrial [ editar ]
Entre todas las vías metabólicas conocidas en los sistemas vivos, la biosíntesis de ácidos grasos produce los productos más densos en energía. [7] Como resultado, los derivados de ácidos grasos microbianos están emergiendo como una prometedora alternativa de energía renovable a los combustibles de transporte derivados de combustibles fósiles. Recientemente, Khosla et al. [7] han ideado un procedimiento para reconstituir la sintasa de ácido graso de E. coli utilizando componentes proteicos purificados (incluido FabD) e informaron un análisis cinético detallado de este sistema reconstituido in vitro. [8] Sus hallazgos proporcionan una nueva base para evaluar el alcance y las limitaciones del uso de E. Coli como biocatalizador para la producción de combustibles diesel.
Relevancia clínica [ editar ]
FabD como objetivo para el descubrimiento de fármacos antibacterianos: un campo próximo [ editar ]
La biosíntesis de ácidos grasos se lleva a cabo mediante la ubicua sintasa de ácidos grasos . [9] Las vías de la ácido graso sintasa se dividen en dos formas moleculares distintas: Tipo I y Tipo II. [10] En el Tipo I, la sintasa de ácido graso (que se encuentra en humanos y otros mamíferos) es un polipéptido grande único compuesto por varios dominios distintos. [11] Por otro lado, cada actividad enzimática ( reacción de condensación , reacción de reducción , reacción de deshidratación ) se encuentra como una proteína discreta en los sistemas de tipo II. [12]La diferencia en la organización activa del sitio y el predominio de los sistemas FAS tipo II en bacterias hacen que las enzimas de esta vía sean objetivos atractivos para los antibacterianos. [9] [12]
FabD (Acyl-Carrier-Protein S-Malonyltransferase) es un objetivo razonable dado que hay disponible una estructura cristalina de alta resolución. [9] Sin embargo, no se han reportado inhibidores de FabD en la literatura y artículos de revisión sobre este tema. [9] La estructura simple y la acidez del malonato parecen permitir pocos enfoques para sintetizar derivados (que actúan como inhibidores potenciales) que retienen el carácter de la molécula.
Un segundo enfoque para usar FabD como objetivo farmacológico se identifica con frecuencia en la literatura: FabD puede proporcionar una etiqueta útil para localizar genes fab porque el gen FabD generalmente está adyacente a al menos otro gen fab. [13] Sin embargo (a partir de 2015), ninguna droga potencial ha intentado explotar esta característica.
En enzimología , una (desoxi) nucleósido-fosfato quinasa ( EC 2.7.4.13 ) es una enzima que cataliza la reacción química.
- ATP + fosfato desoxinucleósido ADP + desoxinucleósido difosfato
Por lo tanto, los dos sustratos de esta enzima son ATP y fosfato desoxinucleósido , mientras que sus dos productos son ADP y difosfato desoxinucleósido .
Esta enzima pertenece a la familia de las transferasas , específicamente aquellas que transfieren grupos que contienen fósforo ( fosfotransferasas ) con un grupo fosfato como aceptor. El nombre sistemático de esta clase de enzimas es ATP: desoxinucleósido-fosfato fosfotransferasa . Otros nombres de uso común incluyen desoxinucleósido monofosfato quinasa , desoxirribonucleósido monofosfoquinasa y desoxinucleósido-5'-monofosfato quinasa .



No hay comentarios:
Publicar un comentario