Acidobacteria constituye un filo creado recientemente dentro del dominio Bacteria de especies ubicuas en el suelo.1 2
Sus miembros son fisológicamente diversos siendo algunos de ellos acidófilos y fueron reconocidos por primera vez como un grupo distinto en el año 1997.3
Acidobacterium capsulatum fue el primer miembro descubierto en el año 1991. A pesar de que han sido muy poco estudiadas se sabe que contribuyen de manera muy importante a los ecosistemas, particularmente a aquellos en donde la tierra está involucrada.4 Otros miembros de este filo son Holophaga foetida, Geothrix fermentans, Acanthopleuribacter pedis y Bryobacter aggregatus.
Como se descubrieron hace poco tiempo y muchas de ellas aún no han sido cultivadas (para el año 2013), no se conoce con exactitud su metabolismo y ecología.
Recientemente, en 2007, se ha descubierto una nueva bacteria, Candidatus Chloracidobacterium thermophilum,5 la primera de este filo capaz de realizar la fototrofia basada en la bacterioclorofila.
Capsulatum Acidobacteria ( A. capsulatum ) sigue siendo relativamente nueva especie desde su clasificación oficial en 1991, pero se ha avanzado hacia la aclaración de las propiedades y la importancia ecológica de la especie. A. capsulatum son gramnegativos, acidófilo, heterótrofos, anaerobios facultativos y las bacterias en forma de varilla ( 1 ).
A. capsulatum son conocidos por poseer flagelos que permiten la motilidad dentro del entorno ( 1 ). Los flagelos son ellos mismos peritricoso o que se encuentran por toda la superficie de la célula. Otra característica de este organismo es la presencia de altas cantidades deexopolisacáridos revestimiento las células de los aislados de suelo ( 2 ). Función de exopolisacáridos se divide en cuatro categorías: Broads de protección, de adhesión, una respuesta de adaptación o de las interacciones célula-célula ( 3 ). Esto contribuye a la competitividad de A.capsulatum , por ejemplo, que tiene aumento de la adhesión puede permitir que la bacteria para adquirir nutrientes más fácilmente a partir del medio ambiente.
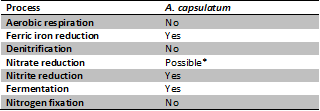
Los miembros de este filo son presumiblemente ubicuo en ambientes de suelos y A. capsulatum podría desempeñar un papel importante en los flujos de carbono , nitrógeno y hierro a través de las comunidades microbianas, debido a sus procesos metabólicos ( Fig 1 ) y la distribución en los suelos.
Ecología
A. capsulatum , aunque primero aislado de drenaje ácido ( 1 ), se presume que sea ampliamente distribuido en los suelos como lo demuestra la detección de 16s rRNA secuencias de su filo a través de una amplia gama de muestras ambientales ( 4 ). Las muestras de suelo de los bosques de Australia y Arizona indicaron que A. capsulatum estaba presente en las comunidades microbianas. Además A.capsulatum también se extiende su presencia a la rizosfera suelos , la interfaz entre la raíz de la planta y de la comunidad microbiana. En este caso, los miembros de la Acidobacteria phylum incluyendo A. capsulatum se encontró que han representado la mayoría de las secuencias de ARNr 16S se encuentran allí ( 5 ). A. capsulatum luego puede jugar un papel en los ciclos geoquímicos en estos suelos.
A. capsulatum prefieren existir en condiciones donde el suelo o los microambientes dentro de ella proporcionan un rango de pH de 3,0 a 6,0 (6 ). Además, son frecuentes y, a veces dominante en ambientes ricos en hierro tales como minas abandonadas ( 2 ). Esto se puede atribuir a su modo de respiración, hierro-reducción, que puede jugar un papel significativo en el ciclismo de hierro en estos ambientes.
Metabolismo
A. capsulatum son anaerobios facultativos ( 7 ), aunque previamente se describieron ser aeróbico cuando fueron descubiertos por primera vez ( 1 ). Cuando las concentraciones de oxígeno son bajos o agotadas, A. capsulatum son capaces de utilizar la reducción de hierro férrico dissimilatory ( 6 ). Como existe hierro férrico más favorablemente en los niveles bajos de pH, la tolerancia a la acidez aparece adaptativo para estos microbios ( Fig 2 ). La frecuencia de A. capsulatum en los suelos puede reflejar el importante papel que desempeña en el ciclo del hierro y de la comunidad microbiana proporcionando hierro ferroso, producido a partir de la reducción de hierro férrico, para la absorción de nutrientes en los organismos.
A. capsulatum tiene los genes necesarios para la celulosa síntesis, permitiendo que el potencial de producción de celulosa bacteriana. Esto es único para A. capsulatum como los genes son raros en los genomas no proteobacterial. Biofilm celulosa bacteriana es la hipótesis de ser capaz de promover y facilitar la adhesión a sustratos de hierro férrico ( 8 ), que puede ser muy útil en entornos de nutrientes hambre. Además, los beneficios de las biopelículas de celulosa también incluyen la protección de la desecación o la desecación y soporte físico. A. capsulatum entonces parecería estar equipados para sobrevivir en ambientes estresantes.
A. capsulatum puede expresar un módulo de adicción , o un sistema de toxina-antitoxina ( 2 ). Las toxinas producen por este sistema son conocidos para inhibir la síntesis de ADN y relacionado, la síntesis de proteínas ( 9 ). Este sistema permite A. capsulatum para adaptarse a entornos donde los nutrientes son escasos, reduciendo su necesidad de energía.
A. capsulatum puede ser capaz de desnitrificación, como genes nitrato reductasa análogas a las que se encuentran en las cianobacterias estaban presentes dentro de A. capsulatum genoma 's ( 2 ). Ellos son, sin embargo, capaz de la reducción de nitrito.
Los diversos modos de mecanismos de respiración y de supervivencia en el A. capsulatum repertorio 's destaca su capacidad para adaptarse a una variedad de ambientes y podría explicar su abundancia en los suelos, donde las condiciones pueden cambiar rápidamente y la escasez de receptores terminales de electrones crecimiento celular límite.
Estructura Genoma
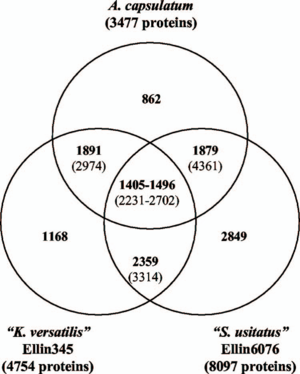
A. capsulatum tiene un tamaño relativamente pequeño genoma de aproximadamente 4,12 millones de pares de bases, mientras que otras especies Acidobacteria tales como K. versatilis y S. usitatus tiene aproximadamente 5,65 y 10 millones de pares de bases, respectivamente. El tamaño de A. capsulatum genoma 's se atribuye a que es más especializado en comparación con otros miembros del phylum, siendo capaces de degradar menos sustrato que los otros. Análisis comparativo del genoma entre las tres especies bacterianas ( Fig 2 ) mostró la mayor parte de los genes compartidos codificados para la función celular conservada. Los genes que eran diferentes representadas algunas diferencias en las vías y preferencia de sustrato. Por ejemplo, a diferencia de los otros dos, A. capsulatum no codifica celulasas de la familia 1 beta-glucosidasas, lo que sugiere la falta de degradación de la celulosa para A. capsulatum . Curiosamente, A. capsulatum tiene los genes necesarios para la síntesis de celulosa ( 2 ).
Gran parte de las proteínas codificadas por A. capsulatum genoma 's están estrechamente relacionados con las proteínas en el filoProteobacteria ( 2 ). Además, los árboles filogenéticos utilizando marcadores de 16S rRNA también muestran Acidobacteria y Proteobacteriaestá estrechamente relacionado en comparación con otros phylums ( 10 ). A. capsulatum y otra Acidobacteria tienden a co-ocurrir con los miembros de la Proteobacteria filo; es posible que la transferencia lateral de genes y co-evolución que se produzca entre los dos y esto puede explicar las similitudes en los genes.
El phylum Acidobacteria es uno de los grupos bacterianos distribuidos más abundantemente en el medio ambiente ( 32 , 34 , 84 ). Sus miembros se han detectado en numerosas encuestas gen 16S rRNA de ambientes que varían en gran medida en las características físicas y biogeoquímicos, incluyendo suelos, sedimentos, agua dulce, marina, extremo y ambientes contaminados en todo el mundo ( 3 , 32 , 34 ). Los análisis de 3000 acidobacterial secuencias de genes 16S rRNA indicaron que el phylum es muy diversa, que comprende 26 subdivisiones filogenéticos distintos ( 2 ). Secuencias Acidobacterial son particularmente abundantes en suelos y sedimentos, que comprende 10 a 50% del total de las secuencias de 16S rRNA genes bacterianos en bibliotecas de clones ( 2 , 5 , 17 , 26 , 34 , 39 , 48 , 52 , 64 , 76 , 77 ), y miembros de distintas subdivisiones se producen en estos entornos diferentes.
Los miembros del phylum Acidobacteria han sido muy difíciles de aislar y cultivar en el laboratorio, y en la actualidad existen representantes cultivadas de sólo 5 de las 26 subdivisiones ( 2 , 32 , 94 ). A través de esta pequeña colección de cepas, la mayoría son heterótrofos aerobios ( 16 , 21 , 37 , 44 , 61 , 62 , 71 ). Una fototrofo ( 6 , 81 ) y dos anaerobios obligados ( 12 , 49 ) también se han descrito. Nombres de los candidatos se han dado a varios aislados, y cinco géneros se han descrito formalmente: Holophaga y Geothrix de subdivisión 8 ( 12 , 49 ) y Acidobacterium ( 41 ), Terriglobus ( 21 ), y Edaphobacter ( 44 ) de la subdivisión 1. Los aislamientos de suelo, todos los cuales son miembros de las subdivisiones 1, 2, 3, y 4, no crecen en medios estándar. Crecen muy lentamente, lo que requiere de varios días o semanas para formar colonias visibles en medios complejos, baja en nutrientes ( 16 , 21 , 37 , 44 , 61 , 81 ). Tales dificultades cultura confunden sustancialmente nuestra capacidad para determinar el metabolismo y los rasgos fisiológicos de los miembros de este filo.
La ubicuidad y abundancia de Acidobacteria en los suelos y su capacidad para soportar ambientes contaminados y extremas sugieren que sirven funciones que son importantes en el medio ambiente y que son potencialmente muy variados. A pesar de nuestra capacidad para detectarlos en muchos ambientes por métodos moleculares, sabemos muy poco sobre su fisiología individual y menos aún sobre sus posibles funciones en, o contribuciones a metabólicas, el medio ambiente. El objetivo de este proyecto fue identificar rasgos acidobacterial potenciales que sugieren funciones ecológicas de los miembros de este filo en el suelo de manera que éstos puedan ser probados de manera más directa en entornos de suelo. Para sortear las dificultades de cultivo, tomamos un enfoque de genómica para identificar estos rasgos. Los genomas completos de tres cepas acidobacterial se describen aquí con una comparación de las características del genoma se centraron en acidobacterial rasgos que pueden contribuir a la supervivencia y el crecimiento en el suelo, incluyendo sus transportadores y sus habilidades para usar carbono, nitrógeno ciclo, scavenge hierro, y producir compuestos antimicrobianos .
MATERIALES Y METODOS
Cepas Acidobacterial.
Dos de las cepas acidobacterial secuenciados representan la subdivisión 1:capsulatum Acidobacterium , aislado de los sedimentos en el drenaje ácido de la mina de pirita Yanahara en Japón ( 41 ), y la tensión Ellin345, aislado del suelo en raigrás hierba / pasto de trébol en Australia ( 16 , 37 ) . Estas cepas de la subdivisión 1 tienen una 16S rRNA similitud de secuencia de 91%. La tercera cepa, Ellin6076, representa la subdivisión 3 y también se aisló de suelo australiano ryegrass / trébol de pasto ( 16 ,37 ). Tiene una 16S rRNA similitud de secuencia de 84% a A. capsulatum . Las dos cepas de suelos australianos han sido informalmente dados los candidatos binomios latinos " Candidatus Koribacter versatilis "Ellin345 y" Candidatus Solibacter usitatus "Ellin6076 ( www.jgi.doe.gov ). Aquí se les conoce como Ellin345 y Ellin6076, respectivamente.
Cultura y ADN genómico.
Las células de la Acidobacteria secuenciado son pequeñas varillas (Ellin345 y Ellin6076, aproximadamente 0,5 por 1,0 m; A. capsulatum , 0,5 por 2,0 micras). Los dos aislamientos de suelo están recubiertas en abundante exopolisacáridos (EPS) limo. A. capsulatum DSM 11244 T ( A. capsulatum ) se adquirió de la Deutsche Sammlung von Mikroorganismen und Zellkulturen, y su ADN genómico fue extraído con el kit de ADN del suelo UltraClean (MoBio). Ellin345 y Ellin6076 se cultivaron en medio VL55 ( 16 , 37 ). Sus ADN genómicos se extrajeron con un procedimiento de molino de homogeneización detergente-talón ( 45 ).
Secuenciación.
La secuencia completa del genoma de A. capsulatum se determinó por el método de escopeta de todo el genoma ( 83 , 87 ). Bibliotecas de clones con tamaños de inserción de 1.8 a 2.8 kpb (pequeño) y 6,5 a 11 kpb (medio) se utilizaron para la fase de secuenciación shotgun azar. Cierre de brechas físicas y de secuenciación se realizó mediante una combinación de paseo con cebador, la generación y secuenciación de bibliotecas transposón etiquetado de clones de gran inserto, y PCR multiplex. Secuencia de montaje se realizó con Celera ensamblador ( 60 ). Repite fueron identificados con RepeatFinder ( 88 ), y las secuencias y el montaje de las repeticiones se confirmaron mediante el uso de clones medio de inserción que se extendió por la repetición. Cobertura para el A. capsulatum genoma era 49.530 lecturas, con un promedio de cobertura de aproximadamente 10 veces por cada base.
El método de la escopeta al azar se utilizó para secuenciar los genomas de Ellin345 y Ellin6076. Grande (40 kb) -, medio (8 kb) -, y pequeños (de 3 kb) -Insertar bibliotecas de secuenciación al azar fueron secuenciados para estos proyectos genoma. Después de la etapa de escopeta, lecturas fueron ensamblados con paralelo Phrap (Software de alto rendimiento, LLC). Misassemblies posibles se corrigieron con Dupfinisher (C. Han, datos no publicados) o el análisis bomba transposón para salvar secuencias entre clones. Las brechas entre contigs fueron cerradas por la edición, camina imprimación costumbre o la amplificación por PCR. La secuencia del genoma completo de Ellin345 contiene 60.626 lee, mientras que la de Ellin6076 contiene 106.291 lee, con ambos genomas logrando un promedio de cobertura de secuencia 10 veces por base con una tasa de error de menos de 1 en 100.000.
Anotación de secuencias y análisis de A. capsulatum .
Identificación de genes que codifican proteínas putativas y anotación del genoma se realizaron como se ha descrito previamente ( 24 , 82 ). Un conjunto inicial de marcos de lectura abierta (ORF) prevé que codifican proteínas (secuencia de codificación) se identificó con GLIMMER. ORFs que constan de menos de 30 codones y aquellos que contienen solapamientos fueron eliminados. Marco de lectura y mutaciones puntuales fueron corregidos o designados "auténtico." Asignación funcional, la identificación de dominios que abarcan la membrana, la determinación de paralogous familias de genes, y la identificación de regiones de la composición de nucleótidos inusual se realizaron como se ha descrito previamente. Análisis Phylogenomic ( 22 , 23 se utilizó) para ayudar con las predicciones funcionales. Los alineamientos de secuencias y los árboles filogenéticos se refinaron como se ha descrito previamente ( 74 ). El A. capsulatum proyecto se ha registrado en el GenBank con el ID de proyecto 28085, y una presentación GenBank está en preparación.
Análisis de secuencias y anotación para Ellin345 y Ellin6076.
La identificación de los genes que codifican proteínas putativas y anotación automática inicial de los genomas fueron realizadas por el Departamento de Energía (DOE) Joint Genome Institute (http://compbio.ornl.gov/public/section/genome.html ). Análisis comparativo de los genomas Ellin345 y Ellin6076 se realizó en parte con el sistema integrado de genomas microbianos ( 55 ) ( http://img.jgi.doe.gov).
La reconstrucción filogenética.
Para cada uno de 31 marcadores filogenéticos ( DnaG, FRR, infC, Nusa, pgk, pyrG, RPLA, rplB, RPLC, rpld, rplE, RPLF, rplK, RPLL, rplM, rplN, rplp, RPL, rplT, RPMA, rpoB, RPSB , RPSC, rpsE, RPSI, rpsJ, RPSK, RPSM, RPS, smpB , y TSF ), un sistema automatizado se aplicó que identificó el marcador en los genomas de consulta, alineado las secuencias, y se recorta la alineación de acuerdo con una máscara predefinida. Para cada marcador, las secuencias de proteínas de semillas fueron alineados y de forma manual editado y un modelo de Markov oculto fue hecho. Una máscara de secuencia se une manualmente a cada alineación para indicar las columnas ambiguas para ser quitados. Los modelos ocultos de Markov se utilizan entonces con el paquete HMMER para llevar a cabo los pasos 1 y 2 descritos anteriormente.
α-glucuronidasa comparaciones de secuencias.
Las secuencias fueron alineadas con el músculo ( 20 ).Las secuencias alineadas se agruparon con el algoritmo Secator ( 90 ), que se basa en BIONJ ( 27 ) para construir un árbol de la alineación de secuencias múltiples y posteriormente colapsa las ramas de subárboles después de la identificación de los nodos que unen diferentes subárboles ( 90 ). El árbol vecino a participar se hizo de la matriz de distancia resultante mediante el uso de parámetros de sustitución BLOSUM62.
RESULTADOS Y DISCUSIÓN
Características Generales genoma.
Los tres genomas difieren mucho en tamaño (tabla (Tabla 1).1 ). El A.capsulatum genoma era el más pequeño, aproximadamente a 4,1 Mb. El genoma Ellin345 era aproximadamente 1,5 Mb más grande que el A. capsulatum genoma y contenía aproximadamente 50% más ORF. El genoma Ellin6076 era mucho más grande que los otros dos genomas (2,4 veces mayor que la de A. capsulatum ), con un mayor número de genes en grupos paralogous. Los plásmidos no fueron identificados en cualquiera de los genomas. Prophages integrados se encontraron en los genomas de A. capsulatum (dos regiones completas) y Ellin345 (una región completa), pero no en el de Ellin6076 (que contenía integrasas y transposases solamente aisladas). Los tres genomas se caracterizan por un bajo número de copias de genes rRNA, una copia en la subdivisión 1 genomas y dos copias en el genoma Ellin6076. Esta característica es consistente con su lento crecimiento observado y se ha postulado que es un marcador característico de crecimiento lento y una K estilo de vida -seleccionado ( 42 , 80 ). Los genomas de los tres aislados contenían complementos completos de genes flagelares y la quimiotaxis, pero flagelos sólo se han observado en cultivos de A. capsulatum y Ellin345.
Filogenia.
A través de 16S rRNA análisis de secuencias de genes, Acidobacteria se colocó como un phylum distinto y bien apoyado mayor ( 32 , 46 , 94 ), pero las relaciones más profundas a otros phyla no estaban claros. Un estudio reciente coloca dos de los aislamientos acidobacterial dentro de la claseDeltaproteobacteria ( 11 ). Sin embargo, la coherencia de la clase Deltaproteobacteria puede ser perturbada por la introducción de nuevos taxones ( 53 ). El análisis filogenético con un conjunto de datos concatenados de 31 proteínas obtenidos a partir de nuestros datos de la secuencia del genoma confirmó la monofilia de la Acidobacteria indicado por análisis de genes 16S rRNA y los colocó adyacente a la proteobacteria en 87% de los árboles generados a partir de conjuntos de datos bootstrapped (Fig. (Fig.1).1 ). Filogenias Single-proteína reconstruidos a partir de todas las proteínas codificadas por el A. capsulatum genoma apoyar la relación acidobacterial-proteobacterial; más A. capsulatum proteínas están más estrechamente relacionados con los equivalentes proteobacterial que a los de cualquier otra phylum (datos no mostrados). Otra evidencia genómico sugiere una ascendencia compartida y / o transferencia lateral de genes entre el Acidobacteria y proteobacteria ( 57 , 66 ). Un lactonasa quórum para saciar acidobacterial detectado en un metagenoma suelo se encontró que era más estrechamente relacionados con los equivalentes de rizobios ( 67 ). En la mayoría de los suelos, los dos más abundante phyla son Acidobacteria y Proteobacteria ( 34 ) y en entornos ácidos de minas, Acidobacteria y alphaproteobacteria acidófilas tales como Acidosphaera spp. y Acidicaldus organovorus ( 35 ) se han encontrado para ocurrir juntos. La co-ocurrencia y abundancia relativa de estos dos filos apoyan la posibilidad de la transferencia lateral de genes entre sus miembros.
Similitudes genoma Generales.
Utilizando el análisis bidireccional mejor hit de todas las secuencias de proteínas, se compararon las composiciones globales de estos tres genomas (Fig. (Fig.2).2 ). Alrededor del 25% de la proteína secuencia de codificación de A. capsulatum o Ellin345 era única para que el genoma, y aproximadamente 34% del genoma Ellin6076 era única. Como era de esperar, los genes comunes a las tres cepas mayoría codifican funciones de mantenimiento, mientras que los genes únicos fueron dominados por proteínas o proteínas de función desconocida hipotéticos. Para los genes que se encuentran para ser comunes a los tres genomas y en cada comparación por pares, véase el cuadro S1 en el material complementario.Además de la caracterización de los genes de la cepa específica de la expresión génica y la estructura de la proteína analiza proporcionaría información muy necesaria sobre las capacidades metabólicas y la fisiología de estos Acidobacteria.
Uso de hidratos de carbono.
La capacidad de utilizar una variedad de sustratos de carbono se utiliza a menudo como una característica de diagnóstico de especies individuales y también sugiere actividades posibles en el suelo. La presencia de genes que indican el uso de sustrato de carbono se presenta en la Tabla Table2.2 . En algunos casos, esta capacidad también ha sido verificado por la cultura en la que el sustrato (tabla (Tabla 2)2 ) (41 , 71 ). Todos los tres genomas codifican la capacidad de degradar una variedad de azúcares, aminoácidos, alcoholes, y compuestos intermedios metabólicos. También codifican la capacidad de utilizar sustratos complejos tales como xilano, hemicelulosa, pectina, almidón, y quitina. Formas de carbono poliméricos han sido exitosas sustratos para el cultivo de Acidobacteria de los suelos, posiblemente debido a que permiten la Acidobacteria a outcompete otras especies bacterianas que no pueden utilizar estos sustratos complejos a bajas concentraciones ( 16 , 37 , 73 , 75 ).
Los genomas contienen un gran número de genes que codifican hidrolasa-glucósido, colocándolos en el 5% de los> 500 genomas bacterianos encuestados en la base de datos de la enzima en hidratos de carbono activo ( www.cazy.org ). El genoma de Ellin6076 contenía más glucósido hidrolasas ( n = 117) que el genoma humano y casi tres veces más que la de Saccharomyces cerevisiae ( 14 ). Los genes implicados en el transporte y el metabolismo de hidratos de carbono constituyen una de las cuatro familias de genes más expandidas en Ellin6076 y contribuyeron al tamaño relativamente grande de su genoma.
Todos los tres genomas contenidas celulasas candidatos de la familia glicósido hidrolasa 9. Ellin345 y Ellin6076 también contenían β-glucosidasas de la familia 1, lo que sugiere que son capaces de degradar sustratos de celulosa. Aunque ninguna de las celulasas contenía dominios típicos de unión de celulosa, estudios de la cultura corroboran este hallazgo (tabla (Tabla 2)2 ) ( 71 ). El genoma Ellin6076 también era rico en enzimas capaces de degradar otros polisacáridos de la pared celular de la planta, con las vías enzimáticas completas (liasas de polisacáridos y esterasas pectina) para la degradación de las pectinas y hemicelulosas. A.capsulatum también contenía genes que codifican enzimas que degradan la pared de las células vegetales, con un grupo particularmente grande que codifica la degradación de la pectina. Ellin345 parecía capaz de la degradación de la hemicelulosa de la cadena lateral. Los tres genomas acidobacterial codifican un candidato α-glucuronidasa de GH67 familia (la degradación de la pared celular vegetal hemicelulosa) que parecía compartir la más alta similitud de secuencia con homólogos de hongos (Fig. (figura 3).3 ). Esta evidencia de un evento de transferencia horizontal ancestral no se ha informado a los miembros de cualquier otra familia hidrolasa glucósido. Rasgos que confieren Genoma mecanismos basados en carbohidratos de almacenamiento de carbono (glucógeno) y solutos compatibles (trehalosa) se encontraron en los tres genomas. Estos sugieren un papel potencial de los hidratos de carbono en las vías nutricionales, así como en la resistencia a la desecación.
Vecino a participar en árbol de genes α-glucuronidasa de hongos y bacterias, que muestra la estrecha relación de los genes acidobacterial y fúngicas.
Las propiedades de polímero degradante predichos revelan Acidobacteria como descomponedores en el suelo que potencialmente participar en el ciclo de plantas, hongos, y la materia orgánica de insectos derivados. La abundancia numérica de Acidobacteria suelo sugiere que esta contribución puede ser cuantitativamente significativo. Un análisis reciente de la metagenómica profunda de agua de mar Mediterráneo que utiliza 16S rRNA-dirigido enfoques PCR y hurgar en la basura metagenómica ( 56 ) encontró Acidobacteria para estar entre los grupos de bacterias dominantes. Los genes metabólicos eran en su mayoría relacionados con el transporte y la degradación de biopolímeros, lo que sugiere que Acidobacteria también pueden contribuir a la descomposición en el medio marino.
Los genomas acidobacterial sugieren además la flexibilidad y la novedad en su capacidad para metabolizar de carbono. Cada genoma contiene genes predijo para codificar el monóxido de carbono deshidrogenasa, como se ha señalado anteriormente para Ellin345 y Ellin6076 ( 40 ), pero no se informó anteriormente por A.capsulatum . Dada la ausencia de ribulosa-1,5-bisfosfato carboxilasa / oxigenasa u otras enzimas predichos de participar en la fijación de carbono autótrofos, parece probable que la Acidobacteria complementar su (principalmente heterótrofos) presupuesto de energía con CO en lugar de derivar cantidades significativas de carbono en De esta manera, como se sugiere recientemente para lithoheterotrophs suplementarios comoSilicibacter pomeroyi ( 59 ) (referido como "mixotrophs" en otra literatura [ 40 ]).
La oxidación de CO (ya sea experimentalmente determinado o predicho a partir de la secuenciación del genoma) se encuentra en otros grupos principales de los organismos del suelo, tales como la actinobacteria, pseudomonas, y rizobios ( 40 ). Muchos de estos organismos son también capaces de degradar los polímeros de plantas complejas; es tentador especular que esta propiedad y la oxidación de CO tanto surgieron como estrategias de barrido para optimizar la vida en un ambiente de baja emisión de carbono. Se ha estimado que las bacterias CO-oxidante eliminar aproximadamente el 20% de la entrada total de CO global a la atmósfera (40 ). El consumo de CO por Acidobacteria queda confirmado experimentalmente, pero parece plausible que Acidobacteria hacer una contribución significativa cuantitativamente a dos grandes partes del ciclo del carbono: la degradación de polímeros complejos y la oxidación de CO.
El metabolismo del nitrógeno.
Genomic evidencia sugiere que el papel de Acidobacteria en el ciclo del nitrógeno en suelos y sedimentos es la reducción de nitrato, nitrito, y óxido nítrico posiblemente. Secuencias de genes de la reductasa de nitrato de asimilación que se encuentran en la Acidobacteria son más similares a los descritos para las cianobacterias ( 68 ). El A. capsulatum y genomas Ellin6076 contenida genes candidatos para la nitrato reductasa. Estos genes tenían alguna similitud de secuencia a la asimilación ( NARB) y respiratoria ( narG ) genes de la reductasa de nitrato encontrado en las cianobacterias ( NARB ) yThermus thermophilus ( narG ) ( 8 ). Un homólogo de estos genes no se identificó en Ellin345. Todos los tres genomas contienen Nira , que codifica nitrito reductasa, así como NTCB y glnB , que codifican proteínas de nitrógeno regulatorio que afectan la transcripción de NARB y Nira . Ellin6076 tenía un candidato a otro regulador de nitrógeno, NTCA . Solamente los genomas de las dos cepas de suelo contenían norB y potencialmente un NORC homólogo, lo que sugiere la capacidad reductasa óxido nítrico. No se encontraron pruebas de la capacidad de fijar el N 2 ( nifH ), oxidar amonio ( amoA ) o desnitrificar óxido nitroso ( nosZ ) en los tres genomas. Genes candidatos para el transporte de nitrato ( nrtABCD ) se encontraron en ambas cepas de suelo.
Los transportistas.
La comparación de las principales familias Transporter en los tres genomas proporcionado información sobre las capacidades metabólicas potenciales de las cepas acidobacterial y mostró que las tres especies contenían una muy amplia variedad de transportadores individuales. En general, las cepas de suelo contenían una variedad más amplia de genes que codifican transportadores que lo hizo A.capsulatum (Tabla (Tabla 3).3 ). Los genomas acidobacterial carecían de homólogos sistema de fosfotransferasa transportadora de azúcar, un rasgo compartido con 22% de las bacterias y uno que se correlaciona con el metabolismo de hidratos de carbono estrictamente aeróbica ( 1 ). En cambio, los tres genomas contenían el principal sistema de menor especificidad superfamilia facilitadora para el transporte de azúcar, así como de alta afinidad transportadores ABC propusieron ser ventajoso en ambientes bajos en nutrientes ( 63 ) (Tabla (Tabla 33).
Una gran proporción de cada genoma codificado transportadores (alrededor de 6% de la secuencia de codificación total en cada una de las tres genomas). Los canales iónicos, transportadores ABC, y diferentes tipos de cargadores secundarios (uniporters, symporters, antiporters) se resumen en la Tabla Tabla 3.3 . Los tres genomas acidobacterial contienen un número similar de proteínas de los canales iónicos y ATPasas de tipo P para el transporte de cationes. Ellin6076 contenía el doble de los transportadores de tipo ABC como Ellin345 y cuatro veces más que A. capsulatum . Esta diferencia se debió principalmente a un gran número de exportadores macrólidos ( n = 117) en Ellin6076.
Todos los tres genomas contenían una variedad de porteros secundarias que transportan una amplia variedad de sustratos (por ejemplo, sustratos de carbono, aminoácidos, medicamentos, metales, nitrato / nitrito, ácidos grasos, iones, proteínas) en un proceso mediado por portador. Ellin6076 contenía el doble de los portadores de tipo superfamilia importante facilitador como Ellin345 y A. capsulatum , una diferencia que fue en gran parte debido a una gran cantidad de aniones: symporters cación.
La absorción del hierro y el metabolismo.
Hay una creciente evidencia de que Acidobacteria juegan un papel importante en las reacciones redox de hierro. Ellos se han detectado en y a veces se encuentra a dominar 16S rRNA bibliotecas de secuencias de genes de entornos mineros ricos en hierro (por ejemplo, véanse las referencias 4 , 43 , 69 , y 93 ).
La capacidad para eliminar hierro es crítico para la supervivencia en los suelos. Los tres genomas contenían genes que deberían permitir a estos aislados para tomar el hierro del medio ambiente, incluidos los genesfeoAB , que codifican una alta afinidad ferroso (Fe 2+ ) transportador de hierro ( 38 , 86 ). El hierro es probable que existan ion ferroso sobre todo como sólo bajo condiciones reductoras causadas por anaerobios y / o condiciones de bajo pH, y las especies bacterianas para que esta vía ha sido caracterizados son todos anaerobios facultativos. Las tres cepas acidobacterial secuenciados fueron aislados bajo condiciones aeróbicas, y su capacidad para crecer en condiciones anaeróbicas aún no ha sido probado. Una cepa acidobacterial diferente se ha demostrado para oxidar el hierro ferroso a pH> 3 ( 31 ), y A. capsulatum y cepas relacionadas podrían catalizar la reducción de hierro férrico dissimilatory bajo condiciones limitadas de oxígeno-( 14 ), lo que sugiere que la Acidobacteria puede ser capaz de oxidación de ión ferroso, así como anaeróbico o crecimiento microaerophilic.
Ellin345 y A. capsulatum , pero no Ellin6076, contenía los genes identificados como que codifica hierro permeasa de proteínas de la familia FTR1 y FTR1, así como secuencias similares a la de fet3p , que codifica una oxidasa multicobre participan en la absorción de hierro mediada por FTR1 en la levadura ( 78 ). Los experimentos con Pseudomonas aeruginosa indican que el multicobre oxidasa MCO es un ferroxidasa que funciona en Fe 2+ captación ( 34 ). Los dos A. capsulatum secuencias (ORFA01590 y ORFA00877) tienen 49 y 48% de identidades de aminoácidos a la P. MCO aeruginosa gen ( 33 ), mientras que las secuencias Ellin345 y Ellin6076 tenían identidades de ácido amino inferior (de 22 a 39%) para P. aeruginosa mco .Todas las secuencias acidobacterial tienen estructuras de dominio similar a la de la P. aeruginosa producto del gen, lo que sugiere que ellos también pueden estar involucrados en Fe 2 + captación. Los tres genomas contienen otros genes transportadores de hierro, entre ellos varios genes que codifican Mn 2+ y Fe 2 +transportistas de la familia NRAMP.
Mecanismos de reducción de hierro férrico dissimilatory han sido investigados en Shewanella oneidensis y en Geobacter especies. Las búsquedas de citocromo c proteínas demostrado estar involucrados en la reducción del hierro férrico en S. oneidensis (OmcA, OmcB, Mtra, MtrB, Cyma) identificados Mtrahomólogos en los tres Acidobacteria, uno OmcA candidatos en Ellin345 y cinco genes en Ellin6076 que podrían ser candidatos para cualquiera OmcA o OmcB. Geobacter sulfurreducens proteínas (OmcB, OmcD, OMCE, Fera [= OmcB], PPCA, Fro-1) también se han buscado, pero no se detectaron en los genomas acidobacterial. Estos resultados sugieren que la Acidobacteria no utilice una vía para la reducción de hierro férrico similar a la de cualquiera de Shewanella o Geobacter .
Las bacterias que pueden scavenge hierro mediante sideróforos excretados tienen una ventaja potencial en suelos donde el hierro está estrechamente ligado a los coloides del suelo y la concentración disponible es el crecimiento limitante. BLAST búsquedas de la A. capsulatum , genomas Ellin345 y Ellin6076 mediante el uso de las secuencias de las proteínas necesarias para la síntesis de pyoverdine, micobactina, enterobactina, bacillobactin, aerobactina, vibrioferrin, alcaligin y otros sideróforos como consultas reveló secuencias no muy similares, lo que lleva a la conclusión inicial de que ninguno de los tres genomas contenida ningún gen obvias para la biosíntesis de sideróforos. Sin embargo, hubo candidatos en cada genoma de algunos de los genes que componen la ruta biosintética enterobactina, a saber, entA, entB, Entd, Ente , y entf , así como los exportadores enterobactina genes Ents , y los tres genomas contenía genes que receptores de sideróforos candidato codificar.
La identificación y el análisis de estos genes proporcionan varias pistas que estos tres Acidobacteria pueden utilizar sideróforos producidos por otros microorganismos. (I) Todos los tres genomas contenían múltiples copias de la Tonb, exbB , y EXBD genes, que son necesarios para el transporte de sideróforos ( 28 , 50 , 51 , 65 , 70 ). (Ii) Todos los tres genomas contenían muchos genes que codifican receptores de TonB dependiente, y los receptores de sideróforos conocidos son receptores TonB dependiente (revisado en las referencias 13 , 28 , 50 , y 65 ). (III) con secuencias del receptor de sideróforo conocida como consultas, la parte superior BLAST realiza (con un E -valor de corte de 1e-05) fueron a uno o más de los genes del receptor dependiente de TonB en los tres genomas. Además de los genes del receptor de sideróforo TonB dependiente candidatos, también encontramos candidatos para pvuE , que codifica el componente de ATPasa de un transportador de vibrioferrin, y fepB , que codifica el componente de unión soluto-periplásmico de un transportador de enterobactina. Sin embargo, no se encontraron genes que codifican los otros componentes de estos transportadores.
PKS y NRPSs.
Policétido sintasa (PKS) y el péptido no ribosomal sintasa (NRPS) enzimas son bien conocidos por sus papeles en la síntesis de sideróforos, así como otros productos naturales tales como antibióticos, antifúngicos, antivirales, agentes antitumorales y agentes Antinematodos (revisado en las referencias 9 , 15 , 30 , 47 y 58 ). NRPSs también hacen biotensioactivos ( 79 ). El A. capsulatum genoma contiene tres genes que codifican agrupados NRPSs, varios genes que codifican PKS, y un gen de NRPS-PKS híbrida. Los genes NRPS contenían dominios presentes en Ente y entf. Estos son subunidades de la sintasa complejo multienzimático enterobactina (identificado por SEARCHPKS [ 92 ] y CD-Search [ 54 ]).Varios genes de PKS se encontraron en las dos cepas de suelo, aunque no se localizaron en una región como en el A. capsulatum genoma.
La síntesis de celulosa.
El A. capsulatum genoma contenía un operón con todos los genes identificados como necesarios para la síntesis de celulosa y es uno de los pocos genomas no proteobacterial reportados para contener este operón. Los dos genomas de las cepas del suelo contenían ortólogos claras de todos menos uno de los genes en este operón. Un ortólogo del gen final de la síntesis de celulosa ( CELab no fue identificado). Ellin345 y Ellin6076 ambos contenían una glicosiltransferasa gen de la familia 2 con un poco de similitud de secuencia con una celulosa sintasa UDP-formando de Nitrosomonas eutropha C71 (identidades de aminoácidos 29 y 32%, respectivamente). Sus CELab genes pueden ser demasiado novedoso para definir por similitud con genes de otros organismos. Microfibrillas de celulosa no se han observado al microscopio en cultivos de las cepas secuenciadas, pero los filamentos se han observado al microscopio asociado con Acidobacteria en suelos (CR Kuske, datos no publicados) y las dos cepas del suelo formar "grumos" visibles o "flóculos" en cultivo líquido ( 72 ).
Celulosa bacteriana ha sido implicado en varias funciones relacionadas con la supervivencia en el suelo, incluyendo la producción de biopelículas, el aumento de la supervivencia bajo condiciones de estrés mediante la retención de hidratación bajo condiciones secas, y la promoción de la aireación y capacidad de retención de agua, que contribuyen a la formación de agregados del suelo ( 85 , 89 ) . Johnson et al. ( 36 ) sugirieron que la síntesis de celulosa por Acidobacteria podría promover una biopelícula que mejora su capacidad de adherirse a sustratos ricos en hierro férrico y producir biofilm formadoras, férricos "bioshrouds" de hierro de reducción para proteger residuos mineros sulfurosos. Acidobacteria también puede sintetizar una red de celulosa que atrapa aire y proporciona una red flexible para el agua y la percolación de nutrientes a través del suelo. Future caracterización experimental de la producción de celulosa podría incluir la prueba de la capacidad de formar microfibrillas, que mejorar la formación de la red putativo.
El estrés y la respuesta del hambre.
Los genomas de los tres Acidobacteria contenían genes que codifican módulos adicción putativos, que consisten en un par de toxinas y antitoxinas y originalmente descritos como los mecanismos de mantenimiento de plásmidos. Los módulos de adicción puede operar rápidamente para inhibir la síntesis de ADN y proteínas en respuesta al estrés o el hambre, como se ha demostrado previamente por el gen de perfiles de expresión de Escherichia coli transiciones de crecimiento ( 10 ). Tal respuesta permitiría al Acidobacteria hacer el mejor uso de los escasos recursos en un entorno con períodos de carbono y la limitación energética recurrente.
Tolerancia ácido.
El phylum Acidobacteria fue nombrado después de la primera cepa cultivada, A.capsulatum , que se aisló de los sedimentos en un drenaje de mina ácido. El nombre phylum implica tolerancia al ácido o preferencia, y las tres cepas acidobacterial secuenciados son moderadamente acidófilas.A. capsulatum crece mejor a un pH de 3,0 a 6,0, Ellin345 en el 4,0 a 6,5, y Ellin6076 en el 3,5 a 6,5, y ninguna de las cepas de crecer a un pH por debajo de 3 o por encima de 6,5. Los tres genomas secuenciados contenían genes candidatos en el sistema AR3 que pueden sugerir un mecanismo de tolerancia a condiciones ácidas. Sin embargo, no se encontraron genes implicados en la resistencia a los ácidos sistema inducible AR1, AR2, o AR4. No se encontró evidencia de adaptación a las condiciones ácidas de la comparación de las frecuencias de aminoácidos individuales.
La preferencia por un pH ácido se ha encontrado para ser un rasgo de otra subdivisión 1 Acidobacteria ( 72 ), pero probablemente no es característico de todo el phylum. Por ejemplo, las secuencias acidobacterial en la subdivisión 4 se han encontrado para ser abundante en los suelos de pH neutro o alcalino (en 12 suelos diferentes), pero su presencia fue variable en suelos con pH por debajo de 5,4, y no fueron detectados en suelos con pH de menos de 4,0 ( 3 ). Además, múltiples miembros de la phylum Acidobacteria se han encontrado para ser abundante en suelos alcalinos ( 18 , 19 ).
Rasgos del genoma novela.
Los genomas acidobacterial incluyen nuevas características que son abundantes y que establecen los miembros de este filo, aparte de las de otros phyla bacteriana. En la mayoría de los casos, las funciones de estos genes no son conocidos o completamente descrito y se necesita más investigación sobre la estructura y función de las proteínas para vincularlos a la fisiología y funciones relevantes para el medio ambiente.
Una familia de permeasas huérfanos duplicados está mayormente restringido a la Acidobacteria, con una expansión en particular en Ellin6076. Estos permeasas son proteínas de transporte que contienen dos dominios permeasa transportador de eflujo ABC (PF02687) y carecen de la típica subunidad de unión al ATP. Los permeasas huérfanos se encuentran, en cada caso, como miembros de un par de genes que contiene también un miembro de un regulador transcripcional PadR subfamilia única al Acidobacteria. La configuración inusual de este par de genes sugiere un papel en el transporte (ya sea pasiva o accionado por un mecanismo que no requiere un casete de unión a ATP) y / o de señalización.
El genoma Ellin6076 contenía al menos 45 proteínas de alto peso molecular de función desconocida que se prevé que sea proteínas extracelulares o de la superficie celular. Genes abundantes en esta misma clase ( n = 74) se han encontrado en hutchinsonii Cytophaga , donde se sugirió que pueden ser parte de la maquinaria celulolítica unida a las células ( 91 ). Ellin6076 también puede haber especializado capacidades de proteínas de metas.
El genoma Ellin6076 también contenía el mayor número de PEP-CTERM proteínas ( n = 61) sin embargo, se informa en cualquier genoma microbiano. PEP-CTERM parece ser una señal de exportación de proteínas transpeptidasa mediada asociado con la biosíntesis de EPS ( 29 ). El gen de la PEP-CTERM clasificación enzima predicho EpsH normalmente se encuentra dentro EPS loci, y hay tres copias en el genoma de Ellin6076. Las proteínas PEP-CTERM mismos se postulan para agregar un componente proteico de material extracelular ( 29 ). En 63 especies bacterianas a través de 47 géneros, un genoma individual codifica ya sea la biosíntesis de celulosa (29 especies) o el sistema de PEP-CTERM (34 especies), pero no ambos. El Acidobacteria, con la biosíntesis de celulosa en A. capsulatum pero EpsH / PEP-CTERM en Ellin6076, ahora se unen al alphaproteobacteria, BETAPROTEOBACTERIA y gammaproteobacteria como subdivisiones con al menos un ejemplo de cada uno. La distribución inconexa de estos dos sistemas es compatible con la hipótesis de que tienen papeles distintos químicamente pero funcionalmente similares.
Resumen y direcciones futuras.
Los tres genomas acidobacterial son sólo lejanamente relacionados con otros organismos en los dominios Bacteria . Por esta razón, la anotación de genes acidobacterial basado en la similitud con las especies de otros phyla bacteriana era difícil debido a las similitudes menudo bajo porcentaje. Los estudios fisiológicos sobre el Acidobacteria y miembros de otros phyla diversa son muy necesarios para permitir la anotación más precisa de genes por función. Sin estudios funcionales, la riqueza de que estén disponibles los datos genómicos es de menor uso.
Este estudio genómico comparativo de los miembros del phylum Acidobacteria ha demostrado la posición monofilia y filogenético del filo y ha identificado las características genómicas distintivas que pueden predecir sus capacidades metabólicas y estilo de vida en los suelos. Ellos comparten rasgos con laProteobacteria , sino también con el cianobacterias y hongos. Las especies de bacterias que entran dentro del phylum Acidobacteria son muy diversas, y la Acidobacteria son abundantes en los suelos que difieren en gran medida de las características físicas y químicas ( 7 , 18 , 19 ). Por tanto, es difícil generalizar los rasgos metabólicos a través de la phylum basado en sólo estos tres genomas.
Las tres especies secuenciadas son heterótrofos aerobios tipificados por un número de copias rRNA baja y un crecimiento lento en la cultura. Sólo han sido cultivables en medios bajos en nutrientes. La presencia de limo extracelular abundante en los cultivos y la potencial capacidad de sintetizar celulosa extracelular sugieren una capacidad para soportar ciclos de secado y rehidratación. Estas cepas potencialmente tienen la capacidad de utilizar una amplia variedad de sustratos de carbono que incluyen sustratos complejos, y que se basan en baja especificidad y transportadores de azúcar de alta afinidad. Estos rasgos culturales y genómicos son compatibles con una amplia capacidad metabólica y un estilo de vida adecuado para frenar el crecimiento en entornos en nutrientes limitados. Tal estilo de vida se caracteriza por el uso de sustratos a bajas concentraciones, enzimas catalíticas con afinidades altas y bajas de sustrato V max características, y las tasas de rotación celular lenta ( 25 ). En este estilo de vida, las células individuales pueden permanecer metabólicamente activo o sobrevivir en el suelo durante largos períodos de tiempo, con células bacterianas individuales que dividen sólo unas pocas veces por año en el suelo ( 31 ). En base a la combinación de pruebas fisiológicas y genómica, postulamos que las células de estas cepas son de larga duración, se divide lentamente, exhiben tasas metabólicas lentas en condiciones de baja de nutrientes, y están bien equipadas para tolerar fluctuaciones en la hidratación. Validación experimental de estas predicciones revelará cómo las actividades acidobacterial afecta la calidad del suelo a través de la descomposición de materia orgánica, el ciclo de nutrientes, y la formación de los agregados del suelo.
Los resultados del genoma no han identificado un único factor importante que permitiría más simple o más rápida cultivo de los miembros de este filo. De hecho, nuestra dificultad en el cultivo de estas bacterias puede deberse más a nuestra falta de paciencia y la incapacidad de Acidobacteria para competir en la cultura con otras especies distintas a la falta de condiciones nutricionales adecuadas. Una variedad más amplia de representantes cultivadas puede ser difícil de lograr, pero, junto con los análisis continuo comparativa expresión genómica y genética, contribuiría significativamente a nuestra comprensión de este filo diversa y abundante con el medio ambiente.

No hay comentarios:
Publicar un comentario