Genomas secuenciados
Glossina es un género de dípteros hematófagas africanas conocidas vulgarmente como moscas tse-tse; se conocen 23 especies y diversas subespecies. Son vectores biológicos que transmiten el agente patógeno Trypanosoma brucei, un protozoo que causa la nagana en los animales y la enfermedad del sueño o tripanosomiasis africana en el hombre.
Las moscas tse-tse actuales existen desde al menos 34 millones de años. Se han recuperado e identificado fósilesdel yacimiento fosilífero de Florissant en Colorado datados con esa antigüedad.1
Características
Las moscas tse-tse son vagamente similares a otras moscas comunes, como la mosca doméstica, pero pueden ser distinguidas por cuatro características de su anatomía, dos de las cuales son fáciles de observar:
- La mosca tse-tse pliega sus alas completamente cuando está en reposo de tal modo que una descansa directamente sobre la otra sobre el abdomen.
- La mosca tse-tse tiene una larga probóscide, que se extiende directamente hacia delante y está conectada por un bulbo en la parte inferior de su cabeza.

Haemophilus influenzae, anteriormente llamado bacilo de Pfeiffer o Bacillus influenzae, son cocobacilos Gram-negativo no móviles descritos en 1892 por Richard Pfeiffer durante una pandemia de gripe. Es generalmenteaerobio pero puede crecer como anaerobio facultativo. H. influenzae fue considerado erróneamente como la causa de la gripe común hasta 1933, cuando la etiología viral de la gripe llegó a ser aparente. Sin embargo, H. influenzaees responsable de un amplio rango de enfermedades como meningitis, epiglotitis, neumonía, sepsis y otras de menor gravedad.1
Debido a su pequeño genoma, H. influenzae fue el primer organismo de vida libre cuyo genoma completo fue secuenciado, por Craig Venter. Su genoma consiste de 1.830.140 pares de bases y contiene 1.740 genes.
Serotipos
En 1930 se definieron dos categorías principales de H. influenzae: cepas con cápsula y sin ella. La patogénesis de las infecciones de H. influenzae no se comprende totalmente, aunque la presencia del tipo B encapsulado (HiB) es el principal factor de virulencia. Su cápsula le permite resistir la fagocitosis y la lisis en los huéspedes no inmunizados. Las cepas no encapsuladas son menos invasivas, aunque son capaces de inducir una respuesta inflamatoria que causa trastornos. Como ejemplos de infección por cepas capsuladas se puede mencionar a la meningitis, neumonía y epiglotitis. La vacunación con la vacuna Hib conjugada es efectiva en la prevención de la infección y varias vacunas se usan rutinariamente.
Enfermedades
La mayoría de las cepas de H. influenzae son patógenos oportunistas, esto es, viven en su huésped sin causar enfermedades, pero pueden causar problemas cuando otros factores (tal como una enfermedad viral que reduce la respuesta inmune) crean una oportunidad infecciosa. Se conocen seis tipos de H. influenzae capsuladas: a, b, c, d, e y f,3 así como cepas no capsuladas, responsables de enfermedades emergentes.4
Las enfermedades causadas naturalmente por H. influenzae parecen afectar solo a los seres humanos. En los niños, H. influenzae tipo B (HIB) causa bacteriemia ymeningitis bacteriana aguda. Ocasionalmente causa celulitis, osteomielitis, epiglotitis e infecciones asociadas. Debido al uso rutinario de la vacuna HIB conjugada en EE.UU. desde 1990, la incidencia de la enfermedad HIB invasiva se ha reducido a 1,3 por 100 000 niños. Sin embargo, HIB continúa siendo la causa principal de las infecciones del tracto respiratorio inferior en niños de los países en vías de desarrollo que no realizan vacunaciones. Las cepas de H. influenzae sin cápsula (no del tipo B) causan infecciones del oído (otitis media) y oculares (conjuntivitis) y sinusitis en niños y se asocian con la neumonía. La meningitis, especialmente en infantes, niños mayores de 7 años y en los ancianos, es la manifestación clínica más seria de las invasiones tisulares causadas por Haemophilus influenzae.5 Ciertas cepas de tipo no-b aparecen con mutaciones que causan enfermedades invasivas en individuos vacunados en contra del tipo b (las cepas capsuladas).4
Diagnóstico
El diagnóstico clínico del H.influenzae típicamente es realizado por cultivos o por la técnica de aglutinación en látex. El diagnóstico es considerado como confirmativo cuando el organismo es aislado en un sitio estéril del cuerpo. Cabe mencionar que el H. Influenzae cultivado a partir de el esputo o desde la cavidad nasofaríngea no es válido debido a que generalmente esas zonas están colonizadas por el agente. Otros sitios como el LCR y la sangre sí son válidos y confirmativos.
Cultivo
Los cultivos bacterianos de H. influenzae se realizan en placas de agar, de preferencia agar chocolate, con adición de X (Hematina) y V (NAD), a 37 ° C en un incubador con CO2-enriquecido.6 El crecimiento de agar sangre es sólo un fenómeno satélite alrededor de otras bacterias. Las colonias de H. influenzae aparecen como colonias convexas, lisas, pálidas, grises o transparentes. La observación con tinción de Gram y microscópicos de un espécimen de H. influenzae mostrará cocobacilos Gram-negativos, sin acuerdo específico. El organismo cultivo puede caracterizarse aún más mediante pruebas de catalasa y oxidasa, las cuales deben ser positivas. En las pruebas serológicas es necesario distinguir el polisacárido capsular y diferenciar entre la cepa b de H. influenzae y las cepas no encapsuladas. Aunque muy específicos, los cultivos bacterianos de H. influenzae carecen de la sensibilidad. El uso de antibióticos antes de la toma de la muestra reduce en gran medida la tasa de aislamiento al matar las bacterias antes de que la identificación sea posible.7 Más allá de esto, H. influenzae es una bacteria muy sensible al protocolo de cultivo, y cualquier modificación de este puede reducir las tasas de aislamiento. La H. influenzae crece en la zona hemolítica de Staphylococcus aureus en placas de agar sangre pues la hemólisis de las células de S. aureus libera nutrientes vitales para su crecimiento.
Aglutinación de partículas de látex
La prueba de aglutinación de partículas de látex LAT por sus siglas en inglés (latex particle agglutination test) es un método más sensible para la detección de la H. infuenzae.8 Debido a que el método se basa en antígenos en lugar de bacterias viables en un cultivo, los resultados no son afectados por el uso previo de antibióticos. También tiene el beneficio adicional de ser mucho más rápido que los métodos de cultivo. Sin embargo, no se puede detectar la sensibilidad a antibióticos con LAT, por lo que es necesario un cultivo en paralelo.
PCR
La reacción en cadena de polimerasa (PCR) ha demostrado ser más sensible que cualquiera de las pruebas de LAT o el cultivo.9 Sin embargo, PCR no es aún un método común en la práctica clínica. La Inmunoelectroforesis, por contra, ha demostrado ser un método de investigación de diagnóstico eficaz.
Tratamiento
Una nueva forma de combatir al H. influenzae, al neumococo, y otros patógenos respiratorios y urinarios, es usar una cefalosporina de tercera generación denominada cefditoren pivoxilo, una prodroga éster, especialmente recomendada en casos de resistencia a antibióticos habituales.10 Actualmente esta molécula está avalada por la Semergen y otras sociedades médicas como la mejor opción ante la amoxicilina con clavulánico.[cita requerida]
La resistencia a antibióticos ha incrementado entre las cepas de H. influenzae, mayormente en términos de resistencia a la ampicilina mediada por β-lactamasa, lo que representa una seria preocupación clínica a nivel mundial. Por lo general, aquellas cepas resistentes a la ampicilina son también resistentes al cloranfenicol.5
Interacciones con Streptococcus pneumoniae
H. influenzae y S. pneumoniae se pueden encontrar en el sistema respiratorio superior de los seres humanos. Un estudio de competición en un laboratorio reveló que, en una placa de Petri, S. pneumoniae siempre superaba a H. influenzae atacándolo con peróxido de hidrógeno. Este erosiona las moléculas superficiales que H. influenzae necesita para sobrevivir.
Cuando ambas bacterias se colocan juntas en la cavidad nasal, en el plazo de dos semanas sólo Hemophilus influenzae sobrevive. Cuando ambas se colocan por separado en la cavidad nasal, ambas sobreviven. Al examinar el tejido fino respiratorio superior de los ratones expuestos a ambas especies de bacterias, se encontró un número extraordinariamente grande de células inmunes neutrófilas. En los ratones expuestos a solamente una de las bacterias, estas células no estaban presentes.
Las pruebas de laboratorio demostraron que los neutrófilos expuestos a H. influenzae muertos atacaban más agresivamente a S. pneumoniae que los neutrófilos no expuestos. La exposición a H. influenzae muertos no tenía ningún efecto en H. influenzae vivos.
Dos escenarios pueden ser responsables de esta respuesta:
- Cuando S. pneumoniae ataca a H. influenzae, esto actúa como señal para que el sistema inmune ataque a S. pneumoniae.
- La combinación de las dos especies acciona una respuesta del sistema inmune que no es disparada por cualquiera de las especies individualmente.
Se desconoce porqué H. influenzae no es afectado por la respuesta inmune.

El estrechamiento de las causas bacterianas, ¿cuáles son los patógenos bacterianos más comunes asociados con este escenario infecciosa
La meningitis bacteriana es una infección grave ya veces fatal en los niños y adultos [1]
Infección por meningitis puede aparecer en un individuo por la aparición repentina de fiebre, dolor de cabeza, rigidez en el cuello e incluir otros síntomas como aumento de la sensibilidad a la luz y alteración del estado mental [2] [3]. Los factores de riesgo incluyen ajustes comunes donde grandes grupos de personas se congregan juntos como estudiantes de primer año de universidad que viven en residencias [2] [3].
La meningitis bacteriana aguda generalmente ocurre cuando las bacterias entran al torrente sanguíneo y migran al cerebro y la médula espinal. Pero también puede ocurrir cuando las bacterias invaden directamente las meninges, como resultado de una infección de oído o sinusitis, o una fractura de cráneo, o rara vez, después de algunas cirugías[4] [5]
Una serie de cepas de bacterias puede causar meningitis bacterianas agudas [1]. Los más comunes incluyen:
| Los patógenos candidatos | Descripción |
|---|---|
| Streptococcus pneumoniae(neumococo) | Alargado cocos grampositivos dispuestos en pares (diplococos) y cadenas cortas y virulencia se determina por la capacidad de colonizar orofaringe, extendido a normalmente tejidos estériles, estimular las respuestas inflamatorias locales y evadir matanza phagyocytic [4] Esta bacteria es la causa más común de infecciones bacterianas la meningitis en los bebés, niños pequeños y adultos en los Estados Unidos. [5] Es más comúnmente causa infecciones de neumonía o de oído o sinusitis. [5] Los países desarrollados de Europa, así como Estados Unidos comenzaron la implementación de un programa de vacunación con una neumocócica 7-valente vacuna conjugada (7PCV) contra neumococos en 2000-2001 que lleva a que el número de casos de enfermedad invasiva pneuomoccocal para disminuir gradualmente [1]. |
| Neisseria meningitidis(meningococo) | Diplococos Gram-negativa con exigentes requisitos de crecimiento y crece mejor a 35 grados a 37 grados Celsius [4]. Esta bacteria es otra causa principal de la meningitis bacteriana y Huamán son los únicos huéspedes naturales [5] [4]. La meningitis meningocócica ocurre comúnmente cuando las bacterias de una infección respiratoria superior entran en el torrente sanguíneo [4] [5]. Esta infección es muy contagiosa. Afecta principalmente a adolescentes y adultos jóvenes, y puede ocasionar epidemias locales en dormitorios universitarios, internados y bases militares [2] [5] [3]. Existe una vacuna para ayudar a reducir la aparición de esta infección [5] [1]. |
| Haemophilus influenzae tipo b (haemophilus) | , Varillas pequeñas, pleomórfico gram-negativas o cocobacilos que son anaerobios facultativos fermentativos [4]. Aunque algunas especies de Haemophilus comúnmente son colonizados en los seres humanos, Hib son miembros comunes de la flora normal [4]. Haemophilus influenzae tipo b (Hib) bacteria solía ser la principal causa de meningitis bacteriana en los niños. [1] [5] Sin embargo, la introducción de la vacuna Hib en 1987 condujo a la cantidad de casos de meningitis debido a Hib para disminuir drásticamente [ 1] [5]. |
Lo que se toman muestras para pruebas de laboratorio y lo importante es el Laboratorio de Microbiología en el diagnóstico de esta enfermedad?
Fluido cerebroespinal
El examen de laboratorio del LCR suele ser el primer paso para confirmar la presencia de meningitis bacteriana y tenga en cuenta que el examen citológico debe preceder a la centrifugación y calentamiento de la CSF [6] [7].
El líquido cefalorraquídeo es un líquido claro, incoloro que llena el espacio subaracnoideo ventrículos y rodea el cerebro y la médula espinal [8]
Identificación presuntiva de "N.meningitis", "S. neumonía ", y" H. influenzae "se puede hacer sobre la base de un examen citológico de la CSF, morfología de la colonia específica en sangre y / o agar chocolate, propiedades de tinción en una tinción de Gram, o mediante la detección de antígenos específicos en el CSF mediante un ensayo de aglutinación de látex o el uso de una prueba diagnóstica rápida (RDT), tales como la reacción en cadena de la polimerasa (PCR) [6]
Una vez que la CSF llega en el laboratorio, el volumen de LCR disponibles para el análisis debe tener en cuenta: si no se deben centrifugar <1 a="" agar="" chocolate="" de="" deben="" directamente="" el="" en="" gram="" inversa="" la="" lcr="" lugar="" ml="" n="" para="" placa="" sangre="" se="" sembraron="" si="" sobre="" su="" tambi="" tinci="" una="" utiliza="" y=""> 1 ml de LCR está disponible, hay que centrifuga a una fuerza suficiente para sedimentar las bacterias (normalmente a 1000 xg durante 10-15 minutos para suficiente sedimentación) [6] .
Transporte y Almacenamiento
CSF es hipotónica que significa neutrófilos pueden lisar y cuenta pueden disminuir en un 32% después de 1 hora y en un 50% después de 2 horas en las muestras de LCR después a temperatura ambiente [9]. Los patógenos candidatos son organismos exigentes que pueden no sobrevivir a largos tiempos de tránsito o las variaciones en la temperatura y refrigeración puede impedir la recuperación de estos organismos; por lo tanto, las muestras de LCR deben almacenarse a temperatura ambiente o en una incubadora a 37 grados centígrados y si no se pueden procesar inmediatamente y el procesamiento de una muestra de LCR es uno de los pocos procedimientos de microbiología clínica que se deben hacer de inmediato [9]
Procesamiento
Una vez que la CSF llega en el laboratorio de microbiología, el volumen de LCR disponibles para el análisis hay que señalar, como si <1 agar="" cambio="" centrifugada="" chapado="" chocolate="" csf="" de="" debe="" directamente="" disponible="" el="" en="" est="" gram="" i="" lcr="" ml="" n="" nbsp="" no="" para="" placa="" sangre="" ser="" sobre="" tinci="" una="" y=""> 1 ml de CSF está disponible (si el volumen de muestra es suficiente para la centrifugación), debe ser centrifugó a una fuerza suficiente para sedimentar las bacterias, típicamente centrifugación a 1000 xg durante 10- 15 minutos es suficiente para sedimentar las bacterias [6]
Después la muestra se ha centrifugado, el sobrenadante se debe extrae con una pipeta Pasteur y se reservó si se planea la detección de antígeno por aglutinación de látex [6] El sedimento se debe mezclar vigorosamente (por ejemplo, en un tubo cerrado utilizando una máquina de vórtice). Una vez que ha sido bien mezclada, una o dos gotas de sedimento deben ser utilizados para preparar la tinción de Gram y una gota deben utilizarse para consecutivas de los medios de cultivo primario [6].
Punción lumbar
Una punción lumbar se realiza para permitir que este líquido a muestrear, facilitando el diagnóstico de diversas condiciones, incluyendo meningitis bacterianas [8]. Punción lumbar 
La punción lumbar se realiza generalmente con el paciente en la posición de decúbito lateral [8]. Para evitar la rotación de la columna vertebral, alinear los hombros y la pelvis del paciente en un plano perpendicular a la cama [8]. Una línea que une el borde superior de las crestas ilíacas posteriores se cruza con la apófisis espinosa L4 o el espacio intermedio L4-L5 [8]. Inserte la aguja de punción lumbar en la línea media del L3-L4, L4-L5 (más comúnmente) o L5-S1 interespacio vertebral [8]. Estos espacios intermedios están por debajo del extremo de la médula espinal, que termina en el nivel de L1 [8]. Ángulo de la aguja hacia el ombligo y el avance del paciente lentamente [8]. La aguja penetrará el ligamento amarillo, duramadre, aracnoides y para entrar en el espacio subaracnoideo, donde se encuentra el líquido cefalorraquídeo [8].
Las complicaciones de la punción lumbar
complicaciones asociadas con la punción lumbar son variables, que van desde alteraciones leves en molestias como dolor de cabeza a hernia cerebral potencialmente mortal, que pueden ocurrir en el paciente con hipertensión intracraneal [7] [8].
complicaciones asociadas con la punción lumbar son variables, que van desde alteraciones leves en molestias como dolor de cabeza a hernia cerebral potencialmente mortal, que pueden ocurrir en el paciente con hipertensión intracraneal [7] [8].
Dolor de cabeza y dolor de espalda
dolor de cabeza y dolor de espalda son los eventos adversos más frecuentes asociados con la LP como dolor de cabeza puede ocurrir hasta en un 60% y el dolor de espalda puede ocurrir hasta en un 40% de los pacientes que se someten al procedimiento y pueden ser graves y debilitantes [8] Este se debe a la pérdida de LCR a través del sitio de la punción dural [8]
dolor de cabeza y dolor de espalda son los eventos adversos más frecuentes asociados con la LP como dolor de cabeza puede ocurrir hasta en un 60% y el dolor de espalda puede ocurrir hasta en un 40% de los pacientes que se someten al procedimiento y pueden ser graves y debilitantes [8] Este se debe a la pérdida de LCR a través del sitio de la punción dural [8]
Hernia cerebral
Después de la punción lumbar, normalmente hay una leve, transitoria disminución de la presión del LCR lumbar como resultado de la eliminación de los CSF y continuó la fuga de LCR de la abertura hecha en la membrana aracnoides que se comunica rápidamente en todo el espacio subaracnoideo [7] [ 8]. En pacientes con intracraneales, lesiones que ocupan espacio, hay un gradiente de presión relativa con desplazamiento hacia abajo del cerebro y del tronco cerebral que puede ser aumentado por punción lumbar precipitando así hernia cerebral [7] [8]. Los estudios han concluido que el riesgo real de complicaciones graves por la punción lumbar en presencia de edema de papila es inferior a 1,2% [7].
Después de la punción lumbar, normalmente hay una leve, transitoria disminución de la presión del LCR lumbar como resultado de la eliminación de los CSF y continuó la fuga de LCR de la abertura hecha en la membrana aracnoides que se comunica rápidamente en todo el espacio subaracnoideo [7] [ 8]. En pacientes con intracraneales, lesiones que ocupan espacio, hay un gradiente de presión relativa con desplazamiento hacia abajo del cerebro y del tronco cerebral que puede ser aumentado por punción lumbar precipitando así hernia cerebral [7] [8]. Los estudios han concluido que el riesgo real de complicaciones graves por la punción lumbar en presencia de edema de papila es inferior a 1,2% [7].
Sangre
Se extrae sangre de la punción venosa de modo especímenes cultivo de sangre pueden ser utilizados para medios de cultivo primario y subcultivo aísla para el ensayo de [6]
El cultivo de sangre debe ser inoculado en una botella (a menos de 1 minuto) para evitar que la sangre se coagule en el synringe [6]
Examine el frasco de hemocultivo para turbidez en 14-17 horas y luego todos los días durante un máximo de 7 días. Cualquier turbidez o lisis de los eritrocitos puede ser indicativo de crecimiento y subculturas sobre soporte cultivo primario se deben hacer de inmediato ya que todos los patógenos candidatos son organismos frágiles, subculturas deben realizarse el día 4 al día 7 independientemente de turbidez, como la ausencia de turbidez no se correlaciona con teh ausencia de crecimiento de las bacterias [6]
Transporte y Almacenamiento
Si el transporte a un laboratorio de microbiología no es posible el mismo día, coloque el frasco de hemocultivo en una incubadora a 35-37 grados centígrados, con 5% de CO 2(o en un tarro de la vela) hasta que el transporte es posible [6]
Si el transporte es posible, se incuba la botella de hemocultivo a 35-37 grados centígrados, con 5% de CO 2 [6]
Procesamiento
Antes de la subcultura, girar la botella de cultivo de sangre varias veces para mezclar el contenido, luego desinfectar el tapón de goma del frasco de cultivo de sangre con un algodón empapado en alcohol 70%, sigue este aspirando 1 ml con una jeringa y aguja estériles de la botella de hemocultivo y transferir 0,5 ml a un BAP y 0,5 ml a un CAP [6].Finalmente racha de las placas de aislamiento, se incuban a 35-37 ° C con ~ 5% de CO2 (o, en una vela-jar), y examinar todos los días durante un máximo de 72 horas [6].
Los aislamientos siempre debe ser inspeccionado por la pureza del crecimiento mirando morfología de las colonias antes de realizar cualquier prueba [6]. Si se observa cualquier tipo de contaminación, las culturas deben ser re-estrías para asegurar la pureza antes de la prueba [6].
Nota: No utilice la povidona yodada en el tapón de goma, ya que se puede llevar en el medio por la aguja que pasa, lo que inhibe el crecimiento de bacterias. Alternativamente, si la botella de cultivo de sangre tiene un tapón de rosca, abra la botella y extraer el líquido utilizando una técnica estéril (es decir, el flamear la boca de la botella después de la apertura y el cierre de la tapa) [6].
Importancia del Laboratorio de Microbiología
Debido a que la meningitis y es síntomas son causados por la inflamación resultante de las meningies, la etiología de la meningitis es enorme [10]. [11]. Además de las causas bacterianas meningitis puede ser causada por infecciones virales como el enterovirus, virus de las paperas y arbovirus; hongos tales como Candida, Cryptococcus sp. y Coccidiodes immitis, así como por causas no infecciosas tales como productos químicos, materiales relased de dermoides o epidermoides quistes, lupus sistémico eritematoso, y meningitis de Mollaret. Sin embargo, la meningitis bacteriana puede ser fatal en un corto período de tiempo desde el inicio de la infección y por lo tanto, realizaron diagnóstico adecuado a través del laboratorio de microbiología es crucial [12] [7] [13]
Explicar las pruebas que se realizarán en las muestras con el fin de detectar (todos) los patógenos bacterianos que pueden causar esta enfermedad.
General cefalorraquídeo Resultados de Fluidos
El diagnóstico de la meningitis bacteriana se basa en el examen del LCR se realiza después de la punción lumbar [7]. Anormalidades CSF típicos asociados con meningitis bacteriana incluyen lo siguiente [6] [7]
Para adultos
- Turbidez [6]
- La presión de apertura es de alrededor de la gama de 200 a 500 mm H 2 O, y la aparición de LCR puede ser turbia, dependiendo de la presencia de concentraciones significativas de glóbulos blancos (WBC), glóbulos rojos (RBC), bacterias, y / o proteína [7] [6].
- Pleocitosis (de leucocitos polimorfonucleares) WBC recuento elveated, por lo general en el rango de 1000-5000 células / mm 3 o 500 células / ul o superior aunque el intervalo puede extenderse a <100 a=""> 10, 000 células / mm 3 [7] [ 8] [6].
- La meningitis bacteriana por lo general conduce a un predominio neutophil en el LCR, típicamente entre 80% y 95%, donde 10% de los pacientes con meningitis bacteriana aguda presentan un predominio de linfocitos (definida como> 50% de linfocitos o monocitos) en el LCR [7]. Esto se debe al deterioro de la integridad de los vasos de la barrera hematoencefálica que conduce a un aumento de la migración de los neutrófilos [9].
- La concentración de glucosa CSF es <40 50-60="" a="" aproximadamente="" bacterias="" barrera="" class="reference" conduce="" consumo="" de="" dl="" durante="" el="" en="" glucosa="" hematoencef="" id="cite_ref-gluk_13-1" infecci="" infiltran="" integridad="" la="" las="" lica="" los="" mg="" n="" nbsp="" no="" pacientes="" que="" s="" se="" span="" style="font-size: 10.5px; line-height: 1em; position: relative; top: -0.5em; unicode-bidi: -webkit-isolate; vertical-align: baseline;" sufre="" transporte="" trav="" y="" ya="">[13]
Presunta CSF pruebas de diagnóstico para determinar la etiología de la meningitis bacteriana
En combinación con el examen del LCR anteriormente, un diagnóstico presuntivo de la meningitis bacteriana causada por N. meningitidis "," S. pneumoniae ", o" H. influenzae se puede hacer después de realizar una tinción de Gram del sedimento CSF o por la detección de antígenos específicos en el líquido cefalorraquídeo mediante una prueba de aglutinación de látex o el uso de las PDR. Resultados positivos para cualquiera de estas pruebas pueden proporcionar rápidamente evidencia de infección, incluso si las culturas no crecen [6]
Tinción de Gram
La tinción de Gram es un método empírico para diferenciar especies bacterianas en dos grandes grupos basados en las propiedades químicas y físicas de sus paredes celulares [4] [6]. Bacterias Gram-positivas retienen la mancha principal mientras que las bacterias gram negativas tienen el color de la contratinción [6] [4].
Tinción de Gram examen de LCR permite una identificación rápida y precisa de la bacteria causante de 60 a 90% de los pacientes con meningitis bacteriana adquirida en la comunidad y tiene una especificidad de> 97% (esta posibilidad de visualizar la bacteria en la tinción de Gram se correlaciona con la concentración de CSF de bacterias en 10 310 5 unidades formadoras de colonias (UFC) / ml produce una tinción de Gram positiva que resulta en 60% de los pacientes y las concentraciones en el LCR de 10 5 principales resultados de microscopía ot positivos en el 97% de los casos. [7]
La probabilidad de tener un resultado positivo tinción de Gram también depende de los patógenos que causan la meningitis bacterianas específicas: el 90% de los casos causados por S. pneumoniae, 86% de los casos causada por H. influenzae, 75% de los casos causada por N. meningitidis tienen resultados mancha Gram positivas [7]
El LCR debe ser centrifugada correctamente con el fin de obtener el sedimento para el procedimiento, y la preparación de frotis adecuada utilizando el sedimento CSF debe producir una monocapa de organismos suficientemente densas para la visualización fácil, pero lo suficientemente delgadas para revelar características morfológicas [6]. Además de las cepas de referencia conocidas para S. pneumoniae, H. influenzae y N. meningitidis, otras cepas de referencia deben utilizarse para cocos gram positivos (Staphylococcusaureus) y bacilos gramnegativos ("Escherichia coli") [6]. Un organismo gram-positiva aparecerá de color violeta oscuro o púrpura, mientras que un organismo gram-negativo aparecerá rojo o rosa de la contratinción [6] [4].
Los falsos resultados de la tinción Gram positiva CSF pueden resultar de una mala interpretación de observador, contaminación de los reactivos o el uso de una aguja de ocluida por punción lumbar (en el que un fragmento de piel extirpada está contaminado con bacterias) [7]. El rendimiento de la tinción de Gram del LCR también puede ser aproximadamente un 20% menor en los pacientes que han recibido tratamiento antibiótico previo [7].
La recomendación para todos los pacientes que están siendo evaluados para sospecha de meningitis se someten a un examen tinción de Gram del LCR (categoría A-III: Buena evidencia para apoyar una recomendación para su uso, siempre se debe ofrecer y la evidencia de las opiniones de autoridades respetadas, basadas en la experiencia clínica, descriptivo estudios o informes de comités de expertos) [7].
Instrucciones para efectuar una tinción de Gram Infografía [14]
[14]
Látex Aglutinación
Aglutinación de látex es fácil de realizar, prueba de diagnóstico rápido (<15 78-100="" aglutinaci="" ant="" anticuerpo="" buena="" cl="" comunes:="" con="" culas="" de="" del="" detecci="" en="" ependiendo="" geno="" genos="" ha="" i="" l="" la="" los="" men="" mezclarse="" min="" mostrado="" muestra="" n="" nbsp="" ngeo="" ngeos="" nica="" para="" part="" pat="" permite="" produzca="" que="" recubiertas="" se="" sensibilidad="" tex="" una="" utiliza="">H.
influenzae tipo b, 67 a 100% para S. penumonaie y 50-93% para N. meningitidis, sin embargo, un resultado de la prueba de antígeno bacteriano negativo no descarta la infección causada por un patógeno meníngeo específica [7].
Sin embargo, el uso rutinario de aglutinación de látex para el diagnóstico etiológico de la meningitis bacteriana se ha llamado recientemente en cuestión como prueba de antígeno bacteriano como problemas han surgió con resultados falsos positivos [7]. Por lo tanto, la aglutinación de látex puede ser más útil para el paciente que ha sido tratado previamente con terapia antimicrobiana y cuya tinción de Gram y cultivo de LCR resultados son negativos (categoría B-III: pruebas moderadas para apoyar una recomendación para su uso; se debe ofrecer y la evidencia general de las opiniones autoridades de respetadas, basadas en la experiencia clínica, estudios descriptivos o informes de comités de expertos) [7].
Varios kits comerciales están disponibles para las pruebas de aglutinación de látex y para los mejores resultados, el sobrenadante de la muestra de LCR centrifugados deben analizarse lo antes posible; si la prueba inmediata no es posible, la muestra de LCR puede ser refrigerado (entre 2 a 8 grados Celsius) durante varias horas, o se congelan a -20 Celsius durante períodos más largos [6]
Prueba de diagnóstico rápido (PDR)
PDR se han desarrollado para la prueba directa de muestras de LCR sin calor antes o centrifugación [6]
En los países en desarrollo, los enfoques más utilizados para la detección y caracterización de patógenos de meningitis bacteriana incluyen el cultivo, la tinción de Gram, y la aglutinación de látex [16]. Aunque la cultura se considera el estándar de oro para la confirmación de casos en las clínicas, la tasa de positivos es relativamente baja debido a almacenamiento y transporte condiciones subóptimas, la práctica cultural y / o tratamiento con antibióticos administrados antes de recoger la muestra [16] .. Mientras que la tinción de Gram es importante, de bajo costo, y debe llevarse a cabo siempre que sea posible, se limita a dar una pista sobre el género y la especie del agente etiológico [16] .. La lectura de los resultados de aglutinación de látex es subjetiva y puede ser difícil de interpretar, sobre todo cuando de una muestra carga bacteriana es baja, por lo tanto, tampoco es práctico hacer el control de calidad en la aglutinación de látex [16]. Cultura debe mantenerse como el patrón oro como bacterias cultivadas son fuentes de datos para la susceptibilidad a los antibióticos, subtipos completa, la expresión de los antígenos que han de incluirse en futuras vacunas, y la fisiopatología de las cepas [16]. Los especimenes que no producen ninguna cultura todavía pueden ser analizados por métodos moleculares (véase más adelante) que se pueden aplicar sobre el ADN extraído de muestras clínicas (por lo general, la sangre y LCR). [16]
Reacción en cadena de polimerasa
PCR ha sido utilizada para amplificar el ADN de pacientes con meningitis causada por los patógenos meníngeos comunes (N. meningitidis, S. pneumoniae, H. influenzae tipo b)[7] Varios estudios han demostrado que las muestras de LCR obtenidas de pacientes con meningoccal enfermedad tuvo una sensibilidad del 91% y 94% respectivamente y la especificidad de 96%, aunque se obtuvieron resultados falsos positivos [7]. Aunque las técnicas de PCR parecen ser prometedores para el diagnóstico etiológico de la meningitis bacteriana, el perfeccionamiento de las técnicas disponibles conducen a su uso en pacientes con meningitis bacteriana para quien el resultado tinción de Gram del LCR es negativo (categoría B-II: pruebas moderadas para apoyar una recomendación para su uso; se debe ofrecer en general y la evidencia de más de 1 ensayo clínico bien diseñado sin aleatorización; a partir de cohortes o de casos y controles estudios analíticos (preferentemente de> 1 centro), a partir de múltiples series de tiempo, o de los resultados dramáticos de experimentos no controlados) [ 7].
PCR ha sido utilizada para amplificar el ADN de pacientes con meningitis causada por los patógenos meníngeos comunes (N. meningitidis, S. pneumoniae, H. influenzae tipo b)[7] Varios estudios han demostrado que las muestras de LCR obtenidas de pacientes con meningoccal enfermedad tuvo una sensibilidad del 91% y 94% respectivamente y la especificidad de 96%, aunque se obtuvieron resultados falsos positivos [7]. Aunque las técnicas de PCR parecen ser prometedores para el diagnóstico etiológico de la meningitis bacteriana, el perfeccionamiento de las técnicas disponibles conducen a su uso en pacientes con meningitis bacteriana para quien el resultado tinción de Gram del LCR es negativo (categoría B-II: pruebas moderadas para apoyar una recomendación para su uso; se debe ofrecer en general y la evidencia de más de 1 ensayo clínico bien diseñado sin aleatorización; a partir de cohortes o de casos y controles estudios analíticos (preferentemente de> 1 centro), a partir de múltiples series de tiempo, o de los resultados dramáticos de experimentos no controlados) [ 7].
Un estudio realizado en 2006 también destacó el uso de PCR y Fluorescente In-Situ La hibridación (FISH) para acompañar a la tinción de Gram para el diagnóstico rápido de la meningitis [1]. Donde el pescado es el método de elección para muestras en las que múltiples bacterias son detectados por tinción de Gram [1] .. En estos casos, FISH permite la identificación del patógeno dentro de 2 horas y se incluyen ventajas de la técnica de FISH muestran el precio bajo y la pequeñas demandas relativas a los equipos técnicos en comparación con la PCR que permite su uso en países en desarrollo con pequeños laboratorios [1]. Sin embargo, debido a su sensibilidad diagnóstica relativamente bajo, el método FISH no se puede recomendar para la elaboración de las muestras con una carga bacteriana baja que no muestran ninguna o muy pocas bacterias en la tinción de Gram [1] casos .Estos pueden rápidamente ser resueltos por PCR en tiempo real con una alta sensibilidad y especificidad mediante el algoritmo del estudio de bajo número de bacterias en la tinción de Gram y cominbation de eubacterial PCR y PCR específico en casos de micropscopy negativo [1]. En conjunto, la PCR y FISH tienen el potencial de acelerar considerablemente el diagnóstico de meningitis bacteriana [1].
Notas Generales de LCR Pruebas de diagnóstico realizadas a patógenos candidatos
| Los patógenos candidatos | Notas generales sobre pruebas etiológicos Above | Cuadro |
|---|---|---|
| Streptococcus pneumoniae |
| |
| Neisseria meningitidis |
| |
| Haemophilus influenzaetipo b | Tanto la sangre y LCR se recogen en los pacientes con el diagnóstico de la meningitis [4] Como hay aproximadamente 10 7bacterias por ml de CSF con meningitis no se trata, de 1 a 2 ml de líquido es generalmente adecuado para micropscopy, la cultura, y las pruebas de detección de antígeno [4]
|
Cultura Primaria de CSF
Los resultados de los cultivos de LCR son positivos en el 70% -85% de los pacientes que no han recibido tratamiento antibiótico previo y se considera el estándar de oro para la confirmación positiva en el laboratorio microbiana pero las culturas pueden tardar hasta 48 horas para la identificación organismo, y se utilizan como el seguimiento en [7] [16] [6][4]
Si se contaminan cultivos primarios, medios selectivos pueden mejorar el aislamiento de estas bacterias de una flora mixta de bacterias u hongos a través de formulaciones de antibióticos [6]
Cultura específico para la identificación y caracterización de patógenos candidatos
| Los patógenos candidatos | Selección de Primaria Medios de Cultivo |
|---|---|
| S. pneumoniae | El medio más óptimo para el crecimiento de S. pneumoniae sería placa de agar sangre (BAP), que es una placa de agar tripticasa de soja que contiene 5% de sangre de oveja; la sangre humana es la sustitución inaceptable como la sangre contiene anticuerpos que pueda inhibir el crecimiento bacteriano [6]. Placa de agar chocolate también es aceptable que es un medio de agar sangre modificado donde se añade la hemoglobina a los medios basales climatizadas convirtiéndolo marrón y es compatible con las bacterias de crecimiento que no crecen en agar sangre [4]. |
| N.meningitis | N.meningitis crece bien tanto en BAP y CAP, donde BAP sólo se debe utilizar, ya que no contiene el hemim y NAD necesario para N.meningitis [6] Una bandeja de poca altura adicional de agua a la parte inferior de la incubadora o una toalla de papel humedecido un tarro vela debe ser considerado como N.meningitiscrece bien en una atmósfera húmeda, salvo en cuenta que la fuente de humedad debe ser cambiado regularmente para evitar la contaminación con los moldes[6]. |
| H. influenzae | Una PAC hecho con el calor lisadas sangre o suplementado con hemina (factor X) y nicotinamida-adenina-dinucleótido (NAD; factor V) se debe utilizar [6]. |
| Los patógenos candidatos | Notas sobre Primaria Medios de Cultivo | Cuadro |
|---|---|---|
| Streptococcus pneumoniae |
| |
| Neisseria meningitidis |
| |
| Haemophilus influenzaetipo b |
|
Análisis confirmatorio para la identificación de las Culturas que Morfológicamente aparecer como patógenos candidatos
| Los patógenos candidatos | Prueba de Diagnóstico y Explicación | Fotos |
|---|---|---|
| S. pneumoniae | Tres pruebas se utilizan para la identificación de S. pneumoniae: la prueba de la catalasa, la prueba optocin, y la prueba de solubilidad de bilis [17].
| |
| N.meningitis | N.meningitis puede ser identificado mediante la prueba de oxidasa y carbohidratos utilización de Kovac [18].
| |
| H. influenzae | H. influenzae puede ser identificado mediante la prueba de oxidasa de Kovac y determinar la necesidad de hemina y NAD como requisitos de crecimiento [19].
|
Reacción en Cadena de Polyermase
En la PCR, el ADN diana se amplifica exponencialmente a través de la repetición de tres pasos principales: 1) desnaturalización del ADN de doble cadena en el ADN de cadena sencilla; 2) hibridación de los cebadores a las secuencias diana de una sola hebra complementarias; y 3) la extensión de los cebadores en la dirección 5 'a 3' por la ADN polimerasa estable al calor para producir moléculas de ADN de doble cadena [20] El número de copias de moléculas de ADN se duplica en cada etapa de extensión, generando millones de copias de la moléculas de ADN originales cuando se completa PCR [20] .. Debido a que el método no requiere células vivas o intactos, la PCR es una herramienta valiosa para la detección de agentes patógenos bacterianos de muestras clínicas, donde las bacterias mueren o lisar fácilmente debido a las condiciones de almacenamiento inapropiados o antes tratamiento con antibióticos [20] .. PCR es ahora ampliamente utilizado en el diagnóstico y la vigilancia de los patógenos bacterianos debido a su alta sensibilidad y especificidad y capacidades de alto rendimiento [20]. Proporciona una herramienta complementaria a los métodos basados en el fenotipo clásico como la cultura, la tinción de Gram, y la aglutinación de látex y con frecuencia mejora los resultados confirmatorios [20].
Un número de ensayos de PCR convencionales se han desarrollado para la detección y subtipificación de bacterias y patógenos virales [20] .. PCR convencional detecta productos en el punto final de la amplificación de ADN mediante la visualización de amplicones utilizando electroforesis en gel de agarosa [20] de detección a base de gel que requiere tubos que contienen amplicones de PCR pueden abrir y manipuladas, lo que aumenta en gran medida el riesgo de contaminación del espacio de laboratorio, equipos y reactivos con los materiales amplificados [20]. PCR convencional es también muy lento y menos sensible y específico que un tipo de PCR llama PCR en tiempo real, por lo que se utiliza principalmente para escribir ensayos que emplean cultivos purificados o muestras clínicas que contienen el organismo en alta densidad [20]. El uso de PCR en tiempo real se está expandiendo rápidamente porque es más fácil de realizar y, al ser un sistema cerrado, reduce los problemas de contaminación potenciales inherentes a la PCR convencional [20]. Muchas de las mismas precauciones mencionadas para ensayos de PCR convencionales también se aplican a los ensayos de PCR en tiempo real [20]
En tiempo real de reacción en cadena de polimerasa
PCR en tiempo real combina la amplificación y detección en un solo paso mediante el uso de colorantes fluorescentes [20]. Hay dos tipos de sistemas de detección: no específicos y específicos. Sistemas de detección no específicos utilizan un colorante fluorescente que se intercala en cualquier molécula de ADN de doble cadena y emite fluorescencia mejorada. Este sistema de detección es relativamente barato, pero susceptible a la falsa positividad [20]. Sistemas de detección específicos dependen de sondas de transferencia de energía de resonancia fluorescente que reconocen específicamente secuencias diana, lo que los hace los sistemas de elección para los ensayos de detección molecular [20]. Sistemas de detección específicos son más caros que los sistemas de detección no específicos y requieren diseños de sonda sofisticados. Una señal fluorescente se genera solamente si la sonda interactúa con su objetivo específico y posteriormente se hidroliza durante la amplificación [20]. \. El incremento resultante en la fluorescencia es proporcional a la cantidad de producto de PCR amplificado en la reacción. [20]
Una sonda de doble etiquetado de hidrólisis es un oligonucleótido (~ de 17-35 pb de longitud) marcado con un fluoróforo reportero (por lo general una longitud de onda corta tinte de color) en el extremo 5 'y un fluoróforo extintor (por lo general un tinte de color longitud de onda larga) en el 3 'terminar o en una timina interna o "T" residuo [20]. Cuando la sonda está intacta y excitado por una fuente de luz, la emisión de fluorescencia del colorante informador (o donante) es absorbida por el inhibidor de la fluorescencia (o aceptor) colorante como consecuencia de la estrecha proximidad de los colorantes [20]. Este proceso también se conoce como transferencia de energía por resonancia de fluorescencia (FRET) [20]. Durante la amplificación por PCR, la sonda se hibrida con una región interna de la plantilla de ADN diana entre el cebador directo y reverso. Cuando la ADN polimerasa cataliza la extensión del cebador y llega a la región donde se une la sonda, la actividad 5 'exonucleasa de las escinde ADN polimerasa de la sonda y libera el reportero del extintor [20] Esto permite a la emisión de la fluorescencia procedente del informador medio de contraste para observar porque ya no es absorbida por el inhibidor de la fluorescencia que se ha difundido de distancia El aumento de la fluorescencia es proporcional a la cantidad de producto de PCR amplificado en la reacción y se mide de forma acumulativa durante el transcurso de toda la tirada PCR [20].
PCR y RT-PCR sobre patógenos candidatos
| Patógenos Candidato | Notas sobre la PCR o ensayo de RT-PCR |
|---|---|
| S. pneumoniae | Ambos ensayos de PCR convencional y en tiempo real se han desarrollado para la detección de S. pneumoniae y genes diana han incluido la neumolisina (capas), autolisina (lytA), y la adhesión a la superficie neumocócica (PAAS) genes [20]. Se recomienda el ensayo de detección de PCR para S. pneumoniae utilizando un segmento específico del gen autolisina (lytA), ya que es altamente conservadas dentro de la especie [20]. El tiempo real de ensayo PCR primers LYTA y sondas que se han encontrado para ser extremadamente fiable para la detección de S. pneumoniae y debido a eventos de recombinación que se producen entre los neumococos y estreptococos estrechamente relacionado, es probable que haya positivos falsos raras o falsos negativos para prácticamente cualquier ensayo en tiempo real para la identificación neumocócica [20]. |
| N. meningitidis | Dos genes pueden ser dirigidos en N. meningitidis especies específicas de ensayos: ctrA y sodC [20]. . El transporte cápsula para gen de la superficie celular, ctrA, está altamente conservada entre los aislados responsables de las infecciones meningocócicas invasivas y ha sido utilizado tanto en tiempo real y la PCR convencional para detectar N. meningitidis [20].
Se trata de un gen dentro sin embargo el locus cápsula, ya que al menos 16% de meningococos realizadas carecen de ctrA, un ensayo de PCR en tiempo real para detectar todos los meningococos, independientemente del estado de la encapsulación, se desarrolló y validó recientemente [20]. Este ensayo se dirige el gen de la superóxido dismutasa Cu, Zn, sodC, que no está genéticamente ligado al locus cápsula. El ensayo detecta sodC meningococos encapsulado, pero también es útil para detectar los meningococos nongroupable que no contienen un ctrA intacta, como se recuperaron durante los estudios de carro. Por esta razón, se recomienda que sodC ser utilizado para la detección de N. meningitidis [20]
|
| H. influenzae | El HPD gen codifica la proteína D, una lipoproteína expuesta a la superficie altamente conservada que está presente en todo encapsulado y no encapsulado H. influenzae [20] La naturaleza conservada de este gen y su presencia en todas las cepas de H. influenzae caracterizado hasta la fecha make que es un objetivo gen altamente atractivo para el desarrollo de un tiempo real de ensayo de PCR específico de la especie H. influenzae [20] |
Para cada patógeno potencial, ¿cuáles son los resultados esperados de estas pruebas
Identificación presuntiva de patógenos Candidato Resultados basados en Primaria Cultura Medios y tinción de Gram Resultados
| Crecimiento en la PAC | Crecimiento en BAP | Tinción de Gram | La identificación presuntiva |
|---|---|---|---|
| Positivo | Positivo | Gram-positiva diplococos | S. pneumonaie |
| Positivo | Positivo | Diplococos gramnegativos | N.meningitis |
| Positivo | Negativo | Gram-negativa pleomórfico cocobacilos | H. influenzae |
Presunta patógenos Candidato Identificación resultados basados en pruebas confirmatorio
S. pneumonaie
| Prueba | Resultados |
|---|---|
| Prueba de la catalasa | La ausencia de burbujeo de una colonia transferido indica un resultado negativo, mientras que cualquier formación de burbujas de una colonia transferido indica una prueba positiva. Falso positivo puede ser el resultado de la transferencia de las células rojas de la sangre así que tenga cuidado al momento de retirar las colonias del BAP para esta prueba [16] |
| Prueba optoquina | El uso de un 6 mm, 5 ug de disco, una zona de inhibición de 14 mm o mayor indica sensibilidad y permite la identificación presuntiva positiva de S. pneumonaie, donde las zonas de inhibición se debe medir desde la parte superior de la superficie de la placa con la parte superior eliminado [16]
Un caso de un aislado completamente resistentes a la optoquina, el diámetro del disco de 6 mm debe ser registrada como una zona más pequeña de la inhibición (<14 bilis="" como="" de="" i="" indica="" inhibici="" la="" mm="" n="" nbsp="" ninguna="" o="" prueba="" que="" requiere="" se="" solubulity="" zona="">S. pneumonaie
|
Gráfico de los resultados de flujo
N.meningitis
| Prueba | Resultados |
|---|---|
| Prueba de oxidasa de Kovac | Las reacciones positivas se desarrollan dentro de los 10 segundos en la forma de un color púrpura donde se aplicaron teh bacterias al papel de filtro tratado, reacción retardada es poco probable con N.meningitis mientras que las reacciones negativas no producirán un cambio de color en el papel de filtro tratado [19] |
| CTA Carbohidratos Utilización | Desarrollo de la turbidez visible y un color amarillo en la parte superior del medio indica crecimiento de bacterias y la producción de ácido y se interpreta como una prueba positiva. Aunque las reacciones pueden ocurrir tan pronto como 24 horas después de la inoculación, algunas reacciones se retrasan y los resultados negativos no deben interpretarse antes de las 72 horas de incubación, un cambio de color a amarillo y sin turbidez no es generalmente una reacción positiva [19] |
Gráfico de los resultados del flujo N.meningitis
H. influenzae
| Prueba | Resultados |
|---|---|
| Prueba de oxidasa de Kovac | Las reacciones positivas se desarrollan dentro de los 10 segundos en la forma de un color púrpura donde se aplicaron las bacterias al papel de filtro tratado y retraso reacciones son poco probable H. influenzae [19] |
| Hemina y NAD disco de papel y placas quad | Discos de papel H. influenzae sólo crecerá alrededor del disco de papel que contiene tanto hemina y NAD [19] |
La detección de patógenos candidatos Resultados Basado en PCR
Tamaños banda en geles de agarosa deben coincidir con los de los controles positivos para la confirmación [26]
Referencias
- ↑ 1.00 1.01 1.02 1.03 1.04 1.05 1.06 1.07 1.08 1.09 1.10 1.11 1.12 Poppert, S., Essig, A., Stoehr, B., Steingruber, A., Wirths, B., Juretschko, S., .. . y Wellinghausen, N. (2005). El diagnóstico rápido de la meningitis bacteriana por PCR en tiempo real y de fluorescencia de hibridación in situ. Journal of Clinical Microbiology, 43 (7), 3390 a 3.397.Consultado el 22 de febrero 2015 de http://jcm.asm.org/content/43/7/3390.full.pdf+html
- ↑ 2,0 2,1 2,2 Centros para el Control y Prevención de Enfermedades (2015) Meningitis bacteriana Meningitis Obtenido 22 de febrero 2015 dehttp://www.cdc.gov/meningitis/bacterial.html
- ↑ 3,0 3,1 3,2 3,3 Thigpen, MC, Whitney, CG, Messonnier, NE, Zell, ER, Lynfield, R., Hadler, JL, ... y Schuchat, A. (2011). La meningitis bacteriana en los Estados Unidos, 1.998 a 2007. New England Journal of Medicine, 364 (21), desde 2016 hasta 2.025. Consultado el 22 de febrero 2015 dehttp://www.nejm.org/doi/pdf/10.1056/NEJMoa1005384
- ↑ PR, Rosenthal, KS, y Pfaller, MA (2013). Microbiología Médica, con ESTUDIANTE CONSULTAR acceso en línea, 7: Microbiología Médica. Ciencias de la Salud de Elsevier. Consultado el 02 de febrero 2015
- ↑ 5,0 5,1 5,2 5,3 5,4 5,5 5,6 5,7 5,8 Mayo Clinic Staff. (2015). Mayo Clinic de Enfermedades y Condiciones Meningitis Causas Obtenido 21 de febrero 2015 dehttp://www.mayoclinic.org/diseases-conditions/meningitis/basics/causes/con-20019713.
- ↑ GW, JC Feeley, PS Hayes, AL Reingold, G. Bolan, CV Broome, y CJ Phillips. Trans-aislamiento Medio: un nuevo medio para el cultivo de primaria y Transporte de Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae y. Journal of Clinical Microbiology. 1984; 20: 55-58. 25 Obtenido 23 de febrero 2015 dehttp://www.cdc.gov/meningitis/lab-manual/chpt06-culture-id.pdf
- ↑ AR, Hartman, BJ, Kaplan, SL, Kaufman, BA, Roos, KL, Scheld, WM, y Whitley, RJ (2004). Las guías de práctica para el manejo de la meningitis bacteriana. Clinical Infectious Diseases, 39 (9), 1267-84. Consultado el 22 de febrero 2015 de http://cid.oxfordjournals.org/content/39/9/1267.full.pdf+html
- ↑ 8.00 8.01 8.02 8.03 8.04 8.05 8.06 8.07 8.08 8.09 8.10 8.11 8.12 8.13 8.14 8.15 8.16 Straus, SE, Thorpe, KE, y Holroyd-Leduc, J. (2006). ¿Cómo puedo realizar una punción lumbar y analizar los resultados para el diagnóstico de la meningitis bacteriana ?. Jama, 296 (16), 2012 a 2022. Consultado el 22 de febrero 2015 dehttp://jama.jamanetwork.com/article.aspx?articleid=203808
- ↑ 9,0 9,1 9,2 9,3 Gray, LD, y Fedorko, DP (1992). El diagnóstico de laboratorio de la meningitis bacteriana. Opiniones de microbiología clínica, 5 (2), 130-145. Consultado el 24 de febrero 2015 de http://cmr.asm.org/content/5/2/130.full.pdf
- ↑ 10.0 10.1 Proulx, N., Frechette, D., Toye, B., Chan, J., y Kravcik, S. (2005). t Los retrasos en la administración de antibióticos están asociados con la mortalidad por meningitis bacterianas agudas adultos. QJM, 98 (4), 291 hasta 298. Consultado el 22 de febrero 2015 dehttp://qjmed.oxfordjournals.org/content/qjmed/98/4/291.full.pdf
- ↑ 11.0 11.1 Dubos, F., Moulin, F., Gajdos, V., De Suremain, N., Biscardi, S., Lebon, P., ... y Chalumeau, M. (2006). Procalcitonina sérica y otros marcadores biológicos para distinguir entre la meningitis bacterianas y asépticas. El diario de la pediatría, 149 (1), 72-76. Consultado el 22 de febrero 2015
- ↑ 12.0 12.1 Mandell, LA, y Ralph, E, Fundamentos de Enfermedades Infecciosas D. (1985): Illustrated Editado Edition. Scarborough, Ontario: Blackwell Science Ltd
- ↑ 13,0 13,1 13,2 Scheld, WM (1981). Correlatos fisiopatológicos en la meningitis bacteriana. Diario de la infección, 3, 5-19. Obtenido
- ↑ Medicina Hack (2015) Gram staining- Procedimiento, mecanismo, explicación. Consultado el 23 de febrero 2015 de http://www.medicinehack.com/2012/02/gram-staining-procedure-mechanism.html
- ↑ Klatt. C, E (2015) Prueba de aglutinación de látex. Consultado el 23 de febrero 2015 de http://webpath.med.yale.edu/webpath.htm#menu
- ↑ 16,00 16,01 16,02 16,03 16,04 16,05 16,06 16,07 16,08 16,09 16,10 16,11 Centro de Control y Prevención de Enfermedades. (2015) Capítulo 10: PCR para la detección y caracterización de Meningitis Bacteriana Patógenos: Neisseria meningitidis, Haemophilus influenzae y Streptococcus pneumoniae recuperados 23 de febrero 2015 dehttp://www.cdc.gov/meningitis/lab-manual/chpt10-pcr .pdf
- ↑ 17,0 17,1 17,2 17,3 17,4 17,5 Centro para el Control y la Prevención de Enfermedades. (2015) Identificación y Caracterización de S. pneumoniae 'Obtenido 24 de febrero 2015http://www.cdc.gov/meningitis/lab-manual/chpt08-id-characterization-streppneumo.pdf
- ↑ 18,0 18,1 18,2 18,3 18,4 Centro para el Control y la Prevención de Enfermedades. (2015) Identificación y Caracterización de N.meningitis Obtenido 24 de febrero 2015 dehttp://www.cdc.gov/meningitis/lab-manual/chpt07-id-characterization-nm.pdf
- ↑ 19.0 19.1 19.2 19.3 19.4 19.5 19.6 19.7 19.8 Centro para el Control y la Prevención de Enfermedades. (2015) La identificación y caracterización de H. influenzae Obtenido 24 de febrero 2015 de http://www.cdc.gov/meningitis/lab-manual/chpt08-id-characterization-streppneumo.pdf
- ↑ de Control y Prevención de Enfermedades. (2015) Capítulo 10: PCR para la detección y caracterización de Meningitis Bacteriana Patógenos: Neisseria meningitidis, Haemophilus influenzae y Streptococcus pneumoniae. Consultado el 25 de febrero 2015 de http://www.cdc.gov/meningitis/lab-manual/chpt10-pcr.pdf
- ↑ Chiba, N., Murayama, SY, Morozumi, M., Nakayama, E., Okada, T., Iwata, S., ... y Ubukata, K. (2009). La detección rápida de patógenos causantes de ocho para el diagnóstico de meningitis bacteriana por PCR en tiempo real. Diario de infección y Quimioterapia, 15 (2), 92-98. Consultado el 22 de febrero 2015 dehttp://www.molecular-beacons.org/download/chiba,jic09(15)92.pdf
- ↑ Van de Beek, D., de Gans, J., Spanjaard, L., Weisfelt, M., Reitsma, JB, y Vermeulen, M. (2004). Las características clínicas y factores pronósticos en adultos con meningitis bacteriana. New England Journal of Medicine, 351 (18), 1849/59. Consultado el 22 de febrero 2015 de http://www.nejm.org/doi/pdf/10.1056/NEJMoa040845
- ↑ Brouwer, MC, Tunkel, AR, y van de Beek, D. (2010). Epidemiología, diagnóstico y tratamiento antimicrobiano de la meningitis bacteriana aguda. Opiniones de microbiología clínica, 23 (3), 467-492. Consultado el 22 de febrero 2015 de http://cmr.asm.org/content/23/3/467.full.pdf+html
- ↑ Ray, P., Badarou-Acossi, G., Viallon, A., Boutoille, D., Arthaud, M., Trystram, D., y Riou, B. (2007). La exactitud de los resultados del líquido cefalorraquídeo para diferenciar bacteriana de la meningitis bacteriana no, en caso de frotis teñido gram-negativa. La revista estadounidense de medicina de emergencia, 25 (2), 179-184.Consultado el 22 de febrero 2015
- ↑ Welinder-Olsson, C., Dotevall, L., Hogevik, H., Jungnelius, R., Trollfors, B., Wahl, M., y Larsson, P. (2007). Comparativa de amplio alcance PCR bacteriana y cultivo de líquido cefalorraquídeo para el diagnóstico de la meningitis bacteriana adquirida en la comunidad. Microbiología Clínica y la infección, 13 (9), 879-886. Consultado el 22 de febrero 2015 de http://onlinelibrary.wiley.com/doi/10.1111/j.1469-0691.2007.01756.x/epdf
- ↑ Centro de Control de Enfermedades. (2015) Métodos de laboratorio para la detección de la meningitis bacteriana. Consultado el 26 de febrero 2015 dehttp://www.cdc.gov/meningitis/lab-manual/full-manual.pdf
Estudiante 7
Los patógenos bacterianos
El estrechamiento de las causas bacterianas, ¿cuáles son los patógenos bacterianos más comunes asociados con este escenario infecciosa?
El shock séptico, que puede resultar en ciertos casos de bacteriemia, puede estar relacionado con el síndrome de la presión arterial baja. Del mismo modo, las infecciones del sistema nervioso central como la meningitis son también el resultado de la bacteriemia y pueden estar relacionados con el síndrome de la rigidez en el cuello. Los patógenos bacterianos comúnmente asociados con cualquiera de shock séptico, infecciones del sistema nervioso central o ambos incluyen Haemophilus influenzae, Streptococcus pneumoniae y Neisseria meningitidis (4, 5, 7). Teniendo en cuenta los síndromes relacionados con el sistema nervioso, Neisseria meningitidis se asocia más comúnmente en estos escenarios y que parece ser el agente causal más probable en este caso de estudio.
Haemophilus influenzae
Haemophilus influenzae es un cocobacilos Gram negativa que es un residente común de la nasofaringe como una parte de la microbiota (8). Como un patógeno humano obliga, lo más a menudo causa infecciones en el tracto respiratorio, pero ocasionalmente causa más infecciones que amenazan la vida tales como bacteriemia y meningitis.
Streptococcus pneumoniae
Streptococcus pneumoniae es una bacteria con forma de cocos positivos y Gram-con extremos algo puntiagudo (lanceta) que por lo general está dispuesto en las cadenas o como pares (5, 6). Se puede encontrar en las vías respiratorias de los humanos sanos, pero también puede ser comúnmente asociados con la enfermedad respiratoria en los escenarios infecciosas (9). En los casos complicados de la infección, también puede causar meningitis y septicemia (9). Streptococcus pneumoniae se divide en más de 90 serotipos sobre la base de variaciones antigénicas en su cápsula de polisacárido (6).
Neisseria meningitidis
Neisseria meningitidis es una cocos Gram negativa que es la principal causa de meningitis en todo el mundo (5). Su cápsula ofrece protección contra el sistema inmune y también se utiliza para clasificar serológicamente Neisseria meningitidis en grupos basados en sus antígenos. También es un patógeno humano obliga que se puede colonizar la nasofaringe de los seres humanos sanos como una parte de la microbiota.
Muestreo Clínica
Lo que se toman muestras para pruebas de laboratorio y lo importante es el Laboratorio de Microbiología en el diagnóstico de esta enfermedad?
Recolección de sangre y el líquido cefalorraquídeo (LCR) muestras son necesarios en este caso de estudio con el fin de diagnosticar la enfermedad (1, 6). Esto es extremadamente importante no sólo para la identificación del agente causante (y decidir lo tanto, en el mejor curso de tratamiento), sino también para identificar la extensión de la infección ya que los síndromes presentados indican un sitio sistémica de la infección y no se pueden observar a simple vista . Un mínimo de tres muestras de sangre de 10 ml se debe obtener de la paciente dentro de un período de 24 horas con el fin de asegurar la detección de bacterias (5). Durante cada muestreo, 10 ml de muestra de sangre debe ser colocado en una botella que contiene generalmente de soja tríptico y otro en una botella de 10 ml que contiene tioglicolato (6). La primera permite para el crecimiento bacteriano no específica mientras que el segundo se utiliza para aislar anaerobios obligados. Las muestras de sangre son luego observaron los cambios en la turbidez, lo que indicaría el crecimiento microbiano, y pueden ser posteriormente subcultured para las pruebas que figuran en la siguiente sección. Cuidado también se debe tomar con la desinfección durante el proceso de punción venosa para evitar la contaminación del paciente, muestra o la botella con la microbiota de la piel. Para se obtienen muestras de LCR de 1 ml en un tubo estéril a través del procedimiento de punción lumbar (6). La muestra no debe ser refrigerado y debe ser procesado en el laboratorio dentro de 1 hora de recogida (2). Si el LCR no se puede procesar en una hora, entonces debe ser inoculada en un medio Trans-aislamiento para el transporte.
Una botella de medio Trans-aislamiento.
Pruebas y Resultados Laboratorio
Explicar las pruebas que se realizarán en las muestras con el fin de detectar (todos) los patógenos bacterianos que pueden causar esta enfermedad. Para cada patógeno potencial, ¿cuáles son los resultados esperados de estas pruebas.
Fenotipificación clásica
Este método generalmente no se tratarán en profundidad aquí (hay numerosas pruebas y resultados para cada bacteria), sino formas básicamente tradicionales de identificación de bacterias en la bacteriología han descansado sobre el uso de una amplia gama de sustratos y medios específicos para categorizar sus procesos bioquímicos, inferir su fenotipo y la propia bacteria real (6). Por ejemplo, la utilización de manitol como fuente de carbono se puede ensayar usando los medios de comunicación que contiene manitol y un indicador de pH. Cuando se fermenta el manitol, el pH va a cambiar y el color de los medios de comunicación también cambiará. Una sola prueba y su resultado es muy sentido por sí mismo. Una multitud de estas pruebas debe llevarse a cabo y los resultados se toman juntos con el fin de identificar una bacteria. Alternativamente, algunas pruebas bioquímicas pueden utilizado en conjunción con los otros análisis que se describen en esta sección. Cuando las pruebas se realizan manualmente, Manual de Bacteriología Sistemática o una guía similar de Bergey se utiliza para interpretar los resultados. Las pruebas también se pueden hacer a través de kits comerciales (tales como API) que tienen una serie de pruebas recopiladas juntas a lo largo con un software que interpreta los resultados.
API NH, un kit de prueba comercial para la identificación de Neisseria y Haemophilus.
Haemophilus influenzae
Gram tinción y cultivo
Recuperación de Haemophilus influenzae como se indica a través de la tinción de Gram y cultivo de microorganismo muestras de LCR es altamente indicativo de meningitis (6).En el proceso de tinción Gram, una de las soluciones (yodo de Gram) facilita básicamente retención de cristal violeta (un colorante de color púrpura) en la capa de peptidoglicano de la pared celular bacteriana. Cuando se añade etanol o un agente decolorante similares, violeta cristal que no está asociada con peptidoglicano fácilmente se lava. En las bacterias Gram positivas, la falta de una membrana externa permite cristal violeta para penetrar y asociarse con el peptidoglicano fácilmente y para que no se puede lavar. En el último paso, una mancha contador (por lo general safranina, un tinte de color rojo) se añade, la tinción de las bacterias que se decolora y supuestamente tiene una membrana externa (bacterias Gram negativas). Por lo tanto, las bacterias Gram positivas aparecerían como cocobacilos color púrpura bajo un microscopio de luz y las bacterias Gram negativas como Haemophilus influenzae aparecerían como de color rojo.
Una tinción de Gram de Haemophilus influenzae.
El cultivo de Haemophilus influenzae se puede hacer en agar chocolate enriquecido con IsoVitaleX (IsoVitaleX aumenta el crecimiento) y las colonias aparece, grisáceo-marrón plana que son aproximadamente 1-2 mm de ancho después de 24 horas de incubación (1). Haemophilus influenzae requiere hemina (factor X ) y nicotinamida adenina dinucleótido (factor V) para el crecimiento y estos se añaden para mejorar la selección de Haemophilus influenzae. El requisito para los factores X y V y la falta de actividad hemolítica en agar sangre se utiliza para diferenciar entre Haemophilus influenzae y otros bacilos Gram negativos.
Haemophilus influenzae en agar chocolate.
Los ensayos serológicos
Una amplia gama de ensayo basado en anticuerpos se puede utilizar en muestras para la detección rápida o para complementar los resultados obtenidos a partir de cultivo de las muestras. Estas incluyen la prueba de aglutinación de látex y el ensayo inmunoabsorbente ligado a enzimas (ELISA) que detecta fosfato de ribosa polyribitol (PRP) (6). PRP es un componente del polisacárido capsular en Haemophilus influenzae y es importante para la protección del sistema inmune. En la prueba de aglutinación de látex, perlas de látex recubiertas con anticuerpos específicos para PRP se mezcla con las bacterias aisladas (5). Si PRP está presente, las cuentas se agrupan causando aglutinación. Con la (indirecto) ELISA, un anticuerpo específico para PRP se reviste sobre una placa de microtitulación bien y la muestra se hace fluir sobre ella. Un anticuerpo primario durante PRP se añadió luego seguido por un anticuerpo secundario específico para el anticuerpo primario. El anticuerpo secundario, generalmente ligado a enzima, permite la detección de PRP a través de un cambio de color o fluorescencia cuando se añade el sustrato. Esto permite tanto la detección y cuantificación de PRP en la muestra.
Diagrama de un ELISA indirecto.
Streptococcus pneumoniae
Gram tinción y cultivo
Ser capaz de cultura Streptococcus pneumoniae a partir de muestras de LCR indica un fuerte indicador de meningitis (5). Streptococcus pneumoniae exhibe α-hemólisis actividad (hemólisis parcial), produciendo un color verdoso cuando se siembran en agar sangre (6). Las colonias aparecen como pequeños, redondos y mucoide con una depresión en su centro y crecen mejor bajo alrededor de 5% de CO 2 atmósfera y es inhibida por optoquina (1, 6). Basándose en la descripción anterior en la tinción Gram,Streptococcus pneumoniae se espera que aparezca como púrpura, cocos Gram positivos cuando se tiñen y se observa bajo el microscopio.
Streptococcus pneumoniae en una placa de agar sangre. La flecha apunta al claro verdoso alrededor de una colonia que indica α-hemólisis.
Las pruebas bioquímicas
La prueba de la catalasa permite la detección de la catalasa, una enzima que descompone el peróxido de hidrógeno en agua y O 2 con el fin de diferenciar entre Streptococcus pneumoniae y otros cocos Gram positivos (2). Una reacción positiva se produce cuando no está en ebullición cuando se agregan células bacterianas al peróxido de hidrógeno.Como Streptococcus pneumoniae es la catalasa negativa, no habría ningún burbujeo esperado. La inhibición por optoquina también puede explotarse para identificarStreptococcus pneumoniae mediante la adición de un disco optoquina en agar sangre (2). Streptococcus pneumoniae crecimiento bajo esta condición sería mostrar una zona de inhibición (sin crecimiento) de 14 mm o mayor alrededor del disco. Si la zona de inhibición es menos de 14mm, debe llevarse a cabo la prueba de solubilidad de bilis (2). Esta prueba también permite la diferenciación de Streptococcus pneumoniae de otros estreptococos-α hemolítico. Streptococcus pneumoniae es la bilis soluble, por tanto, una prueba positiva sería un claro cuando la bacteria se suspende en una solución de sales biliares en comparación con un control de bacterias en suspensión en solución salina .
Un ejemplo de optoquina susceptibilidad (derecha) con una zona de inhibición y optoquina resistencia (a la izquierda).
Los ensayos serológicos
Streptococcus pneumoniae también puede ser detectada mediante el uso de la reacción de Quellung (5, 6). En la reacción de Quellung, anticuerpos a partir de un antisuero se une a la cápsula bacteriana, haciendo que se vuelva opaca y más grande si la reacción es positiva y cuando se ve bajo el microscopio. Otro método por el cual el polisacárido capsular en el CSF puede ser detectada es a través de la prueba de aglutinación de látex como se mencionó anteriormente.
Un ejemplo de una reacción positiva Quellung.
Neisseria meningitidis
Gram tinción y cultivo
Como el crecimiento de Neisseria meningitidis es inhibida por trazas de metales y ácidos grasos en algunos medios de comunicación, que se aísla por lo general utilizando agar chocolate que ha sido calentado a 80 ° C para inactivar los inhibidores (5). La incubación se realiza en alrededor de 37 ° C en 5% de CO 2 atmósfera. Las colonias en agar chocolate aparecen como incoloro a gris y son opacos (2). Se espera que la tinción de Gram para producir rojo, cocos Gram negativo.
Las pruebas bioquímicas
Una prueba bioquímica importante tener en cuenta para la identificación de Neisseria meningitidis es la prueba de oxidasa (5). En esta prueba, la presencia de citocromo c (un componente de la cadena de transporte de electrones) se oxida la fenilendiamina en el reactivo, cambiando su color a púrpura o negro. Desde Neisseria meningitidis es oxidasa positiva, se espera que la prueba producirá un cambio de color si la muestra contiene Neisseria meningitidis. La utilización de carbohidratos también puede ser probado para diferenciar entre Neisseria meningitidis y Neisseria spp. (2, 5). Es esta prueba, medios que contienen glucosa, maltosa, lactosa o sacarosa como fuentes de carbono ha añadido indicador de pH rojo fenol. Bajo pH neutral, el medio aparece como rojo, pero cuando se oxidan estos hidratos de carbono, el medio se vuelve más ácida y los cambios de color a amarillo. Neisseria meningitidis se espera que utilizar la glucosa y maltosa solamente lo que se espera para producir rojo para lactosa y sacarosa y amarillo para la glucosa y maltosa.

Enfoques de Biología Molecular
Cultura, tinción de Gram y métodos serológicos siendo un estándar para el diagnóstico en microbiología clínica pero bajo ciertas circunstancias, tales como barreras logísticas o pruebas concluyentes, otros métodos moleculares se pueden emplear con el fin de identificar el agente causante (2). Estos métodos se basan principalmente en la detección de ADN bacteriano en la muestra con el fin de identificar la bacteria que causa la infección. PCR en tiempo real cuantitativa (qPCR) es básicamente una extensión de PCR (ver mecanismo de abajo) que incluye tintes fluorescentes o sondas para relacionar los cambios en la fluorescencia a los aumentos de los amplificados de PCR. Especies objetivos de genes específicos se enumeran en la tabla siguiente (2):
| Patógenos | Genes | Target |
| Neisseria meningitidis | ctrA, soda | proteína de transporte cápsula superficie celular |
| H. influenzae | hpd | superficie expuesta proteínas lipoproteína D |
| Streptococcus pneumoniae | lytA | autolisina |
Aunque no es un componente importante en las prácticas actuales de microbiología clínica, es importante tener en cuenta que la secuenciación de nueva generación no desempeñar un papel en el diagnóstico clínico de enfermedades bacterianas (3, 10). Uno de los métodos de secuenciación de próxima generación de uso común, la secuenciación por síntesis, se muestra a continuación. Datos genómicos obtenidos a través de esta forma pueden proporcionar una mayor penetración en el patógeno bacteriano mediante la identificación de especies directa y la identificación de factores de virulencia. Metodología exacta puede variar pero generalmente implica el aislamiento de ADN, amplificación, construcción de la biblioteca seguido por secuenciación. Los datos de secuenciación se filtra por la contaminación de ADN anfitrión lee y genomas son ensamblados de novo o por mapeo para hacer referencia a los genomas.
Referencias
1. Brooks, GF, Jawetz, E, Melnick, JL, Adelberg, EA. 2013. Jawetz, Melnick, y Adelberg de microbiología médica. McGraw-Hill Medical, Nueva York.
2. Castillo, D, Organización Mundial de la Salud. 2011. Métodos de laboratorio para el diagnóstico de la meningitis causada por Neisseria meningitidis, Streptococcus pneumoniae y Haemophilus influenzae: OMS Manual. Centros de Control y Prevención de Enfermedades.
3. Didelot, X, Bowden, R, Wilson, DJ, Peto, TE, Crook, DW. 2012. La transformación de la microbiología clínica con la secuenciación del genoma bacteriano. Nature Reviews Genetics. 13: desde 601 hasta 612.
4. Johnson, JT. 1996. Microbiología de la systemin Nervioso S. Baron (ed.), Microbiología Médica, 4ª ed.,. Universidad de Texas Medical Branch, Galveston.
5. Levinson, W, Acceso a los medicamentos. 2014. Revisión de Microbiología Médica e Inmunología, 13e.
6. Longo, DL, Acceso a los medicamentos. 2012. Harrison Principios de Medicina Interna. McGraw-Hill, Nueva York.
7. Pelletier, LL. 1996. Microbiología de la Circulatorio systemin S. Baron (ed.), Microbiología Médica, 4ª ed.,. Universidad de Texas Medical Branch, Galveston.
8. Ryan, KJ, Ray, CG, Acceso a los medicamentos. 2014. microbiología médica Sherris. McGraw-Hill Medical Education, Nueva York.
9. Tikhomirova, A, Kidd, SP. 2013. Haemophilus influenzae y Streptococcus pneumoniae: la convivencia en un biofilm. Patógenos y Enfermedades. 69: 114-126.
10. Wilson, MR, Naccache, SN, Samayoa, E, Biagtan, H, Bashir, H, Yu, G, Salamat, SM, Somasekar, S, Federman, S, Miller, S. 2014. diagnóstico Actionable de neuroleptospirosis próximo -Generación de secuenciación. N. Engl. J. Med. 370: 2.408 hasta 2.417.
Estudiante 6
1. Los agentes patógenos causales comunes
El paciente, Minnie, exhibe síntomas característicos de la meningitis que se caracteriza por la hinchazón inflamatoria de las membranas que rodean el cerebro y la médula espinal. La meningitis puede ser causada por hongos, virales, o bacterias patógenos. Los factores de riesgo para la meningitis bacterianas son la falta de vacunas para adultos (después de la vacunación infantil - recomendada entre las edades 11-18), edad (por debajo de 20), que viven en un entorno comunitario (aumento del riesgo de la meningitis meningocócica debido a la propagación por vía respiratoria), embarazo (niño y están en riesgo de la bacteria listeria infección), viajar a lugares con alta prevalencia de la enfermedad meningocócica (África subsahariana durante las estaciones secas), y el sistema inmune comprometido (aumento de la susceptibilidad a la meningitis) (Mayoclinic de 2013, los CDC, 2014). Minnie cumple la mitad o más de estas condiciones de los factores de riesgo.
Hay 4 bacterias que se asocian comúnmente a ser la causa de meningitis aguda, y son Streptococcus pneumoniae, Haemophilus influenzae, Listeria monocytogenes, yNeisseria meningitidis. Meningitis es causada por la migración de estas bacterias en el cerebro y la médula espinal, a través del torrente sanguíneo o invasión directa de la infección de herida abierta (Mayoclinic, 2013). Dada la descripción de cada bacteria abajo, sospecho Minnie ha sido infectado por Neisseria meningitidis como ella ha comenzado a vivir en los dormitorios de la universidad y no ha tenido ningún tipo de vacuna después de sus queridos infancia.
Streptococcus pneumoniae
Streptococcus pneumoniae, otra conocida como neumococo, es una bacteria gram-positivas que tienen una morfología cocos "en forma de lanceta" (Levinson W., 2014, Mayoclinic, 2013). Ellos son comúnmente observados estar en pares (diplococos) o formar cadenas cortas (Levinson W., 2014). Este patógeno es la causa más común de meningitis bacteriana en lactantes, niños pequeños y adultos en los Estados Unidos. Sin embargo, este organismo es más comúnmente conocido por causar infecciones de neumonía o la oreja / sinusitis. Hay vacunas disponibles contra el neumococo para prevenir y / o reducir la infección (Mayoclinic, 2013).
 Figura 1. Digitalización de fotos EM de Streptococcus pneumoniae
Figura 1. Digitalización de fotos EM de Streptococcus pneumoniaeHaemophilus influenzae
Haemophilus influenzae, otra conocida como Haemophilus, son bacterias pequeñas, gram-negativas con una morfología cocobacilo (Brooks GF, 2013, Mayoclinic, 2013). A veces se encuentra que en parejas o en cadenas cortas. Haemophilus son bacterias pleomórficas y por lo tanto son capaces de responder a las señales del medio ambiente mediante la alteración de su tamaño o forma (Brooks GF, 2013). En concreto, es el Haemophilus influenzae tipo b (Hib), que fue conocido antes como la principal causa de meningitis bacteriana en niños (Mayoclinic, 2013). Tipo b, fosfato polyribitol, es uno de los tipos de polisacáridos capsulares que se encuentran en Haemophilus, y es este tipo que causa 95% de las enfermedades invasoras (Levinson W., 2014). Afortunadamente, debido a la inmunización infantil de rutina en los Estados Unidos, la prevalencia de la meningitis causadas por este agente ha disminuido significativamente (Mayoclinic, 2013).
Listeria monocytogenes
Listeria monocytogenes, otros conocidos como Listeria, son anaerobio facultativo, no esporuladas, y las bacterias gram-positivas, que tiene una morfología en forma de varilla (Hohmann EL, 2.012, Mayoclinic, 2013). Son conocidos para crecer en una amplia gama de temperaturas, incluyendo temperaturas de refrigeración (Listera tiene una motilidad volteo en medios fluidos a temperaturas inferiores a 30 ° C), lo que les permite estar presentes en alimentos como quesos blandos, perros calientes y fiambres (Hohmann EL 2012, Mayoclinic, 2013, Ryan KJ, 2014). Aunque la mayoría de las personas sanas no sucumben a la enfermedad cuando se expone a la listeria, este patógeno es más susceptible en las mujeres embarazadas, recién nacidos, adultos mayores y personas con sistemas inmunes comprometidos. Por otra parte, Listera puede viajar a través de la barrera placentaria en las mujeres embarazadas e infectar al feto que causa el nacimiento mortinato o el nacimiento de corta duración. Las personas que están inmunocomprometidas son los más vulnerables a las infecciones de listeria (MayoClinic, 2013).
Neisseria meningitidis
Neisseria meningitidis, otra conocida como meningococo, son bacterias cocos gram-negativas (MayoClinic, 2013, Ryan KJ, 2014). Este patógeno es uno de los más prominente causa de la meningitis bacteriana y lo hace mediante la introducción de la corriente sanguínea después de infectar el tracto respiratorio superior (mayo). La transmisión de este organismo se produce por las vías respiratorias y secreciones de la garganta (CDC, 2014). El meningococo es un patógeno altamente contagiosa que con frecuencia causa epidemias locales en zonas de estar comunales tales como dormitorios (MayoClinic, 2013). 10% de las personas que tienen meningococo en la parte posterior de la nariz y la garganta, a mantener un estado de portador (CDC, 2014). parte posterior de la nariz y la garganta. Por otra parte, meningococo afecta principalmente a adolescentes y adultos jóvenes. Existe una vacuna para prevenir la infección y reducir las ocurrencias de meningococo (Mayoclinic, 2013).
2. Microbiología Laboratorio Diagnóstico
Muestras
Muestra de sangre
Los médicos van a extraer la sangre de la vena del paciente y enviarla al laboratorio. Para los niños, se deben recoger 1-3 ml de sangre, pero para un adulto, deben recogerse 5-10 ml de sangre. La sangre debe ser almacenado y transportado en una botella de cultivo de sangre que tiene anticoagulantes para evitar que la sangre se coagule mientras que el almacenamiento y el transporte. En la botella de cultivo de sangre no es caldo de cultivo de sangre (ya sea 20 ml para niños o para adultos 50ml), en el que la muestra de sangre inocula. Las muestras entonces se deben enviar de inmediato a un laboratorio de microbiología médica para su análisis. Si no es posible, se debe almacenar en 35-37 ° C con dióxido de carbono al 5% hasta ~ enviado para análisis. Las muestras de sangre nunca deben ser almacenados en el refrigerador (CDC, 2012)
En el laboratorio, el microbiólogo médico colocar la sangre en agares de cultivo para buscar el crecimiento de ciertas bacterias. La sangre también se tiñeron para observar bajo el microscopio para la morfología de cualquier bacteria que está presente en la muestra (Mayoclinic, 2013).
Muestra de líquido cefalorraquídeo
El líquido cefalorraquídeo (LCR) se obtiene a través de un procedimiento llamado punción lumbar, y se analiza en el laboratorio (Mayoclinic, 2013). Punción lumbar o punción lumbar se realiza mediante la inserción de una aguja espinal entre vértebras lumbares del paciente en el canal espinal (CDC, 2014, Mayoclinic, 2013). Después de la perforación y metiendo la mano en el espacio subaracnoideo, CSF se aspira (CDC, 2014, Robbins E., 2012). Tres tubos de 1 ml de LCR se deben recoger de la paciente para la microbiología, la química y análisis de la citología. Muestras de LCR deben ser transportados a un laboratorio de microbiología médica dentro de 1 hora. Si no es posible, muestras de LCR deben ser inoculadas en medio TI, o incuban a 35-37 ° C con dióxido de carbono ~ 5% en un área de almacenamiento aprobados (CDC, 2012).
Los pacientes con meningitis suelen tener CSF que tiene un nivel bajo de glucosa, pero con un mayor nivel de proteínas y recuento de glóbulos blancos. El análisis del LCR puede determinar la causa de la meningitis, distinguiendo si el patógeno es causada por un virus o una bacteria. Por otra parte, el análisis del LCR puede determinar qué bacteria es la causa exacta de la patología (Mayoclinic, 2013). Muestras de LCR se pueden teñir para la identificación bacteriana (Ryan KJ, 2014)
 Figura 5. procedimiento de punción lumbar.
Figura 5. procedimiento de punción lumbar.Líquido amniótico
Las muestras de líquido amniótico de las mujeres embarazadas se pueden utilizar para el análisis en la determinación del patógeno (Hohmann EL, 2.012)
Esputo
A medida que estas bacterias se propagan de vías respiratorias, esputo es un potencial ejemplar de muestra que se pueden recoger de los pacientes para la identificación bacteriana y diagnóstico. Sin embargo, la recolección de esputo puede no ser la mejor muestra para obtener como muestras de cosecha son duras y ejemplares pueden estar contaminados con la flora respiratorias que afectarán negativamente a los resultados de laboratorio (Ryan KJ, 2014).
Orina
Las muestras de orina pueden ser utilizados para las pruebas de aglutinación para detectar presencias de antígenos bacterianos (Levinson W., 2014, Vorvick JL, 2015)
Exudado nasofaríngeo
Culturas creados a partir de este ejemplar de muestra son adecuados para Meningococo encuestas portadoras (Brooks GF, 2013).
Importancia de los resultados de laboratorio
Estas infecciones bacterianas pueden ser potencialmente mortal y el diagnóstico precoz y el tratamiento es crítico (CDC, 2014). Mediante la identificación de la fuente de la infección, los médicos pueden crear de inmediato el plan de tratamiento antibiótico adecuado para curar más rápido el paciente antes de resultados fatales ocurren (CDC, 2014, Mayoclinic, 2013). Sin embargo, algunas de las causas de la meningitis pueden ser autolimitada y por lo tanto un régimen de medicamentos de antibióticos puede causar efectos adversos graves en su lugar (MayoClinic, 2013). Por otra parte, si se aplica el tratamiento medicamento equivocado, tales como antibióticos aplicados a una infección viral o el uso de un antibiótico que no afecta a ciertas bacterias, el patógeno existente causará estragos en la parte superior de los efectos secundarios adversos no deseados de los fármacos innecesarios. Por lo tanto la confirmación de la presencia y tipo de bacterias de los resultados de laboratorio son cruciales en el tratamiento de un paciente con meningitis. Aparte de la muerte, meningitis tratadas inadecuadamente pueden causar discapacidades permanentes, como pérdida de la audición y daño cerebral (CDC, 2014)
3. Las pruebas para detección de bacterias
Crecimiento en agar
Agar como un medio de crecimiento contiene los requisitos orgánicos y minerales para las células en crecimiento. Debido a los diferentes tipos de metabolismo que se encuentran en diferentes bacterias, los requerimientos de nutrientes pueden variar en el agar. Por lo tanto, el ajuste de la composición de agar para permitir ciertos tipos de bacterias para crecer es una herramienta clave en la identificación de bacterias de una muestra (Benbasat J., 2012).
Crecimiento y Actividad en agar sangre
Un agar sangre se utiliza generalmente para la cultura, aislar, y observar las bacterias de inoculaciones de muestra. A agar de sangre común se compone de una soja-caseína digerir agar con 5% vol / vol de sangre de mamífero (ya sea humano o animal) (Buxton R., 2013). Agar sangre no siempre es un medio definido (uno que contiene la fuente purificada de carbono y sales puras específicas), sino que se refiere generalmente como un medio de base enriquecido para que desfibrinada sangre ha sido añadido (Benbasat J., 2012, Buxton R., 2013).
Las bacterias que crecen en agar sangre son organismos exigentes que permiten el microbiólogo médico para observar y aislar bacterias presentes en una muestra de rayar técnicas (Benbasat J., 2012, Buxton R., 2013). Bacterias aisladas a continuación, puede poner a través de pruebas bioquímicas y genéticas posteriores a ser identificado.Además, un agar sangre permite que sea posible diferenciar entre diferentes tipos de bacterias, basados en la capacidad hemolítica del organismo. El tipo de sangre en el agar también afecta a la capacidad de las bacterias para crecer. Por ejemplo, nicotinamida adenina dinucleótido (NAD) -requiring organismos, tales como Haemophilus, crecen en el conejo y el caballo de sangre. En los Estados Unidos, se utiliza regularmente sangre de oveja. Sin embargo, la sangre humana se utiliza desalentador debido al aumento del riesgo de exposición a patógenos transmitidos por la sangre humana (Buxton R., 2013).
Una variante de agar sangre también se utiliza para la identificación de bacterias se llama agar chocolate. La diferencia clave entre esta variante y la sangre agar es que estas placas se calientan a 80 ° C para lisar las células rojas de la sangre que libera nutrientes intracelulares tales como la hemoglobina, hemina (factor X) y la coenzima NAD (Factor V). Estos nutrientes son requeridos por ciertas bacterias para crecer que ayuda a diferenciar entre ciertos organismos (Acharya T., 2013).
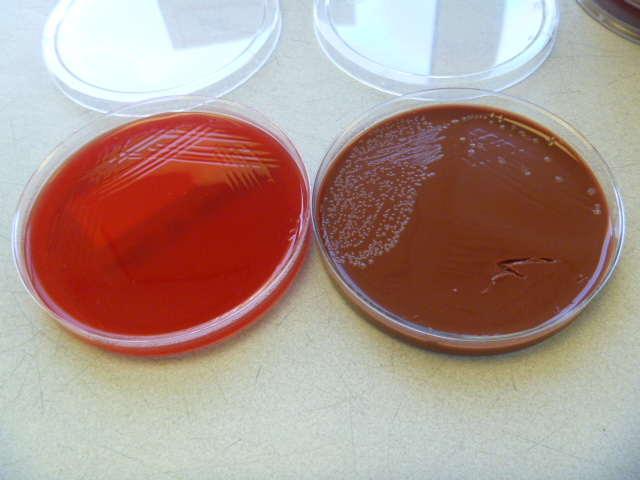 La Figura 7. Agar sangre a la izquierda. Chocolate agar sangre a la derecha.
La Figura 7. Agar sangre a la izquierda. Chocolate agar sangre a la derecha.
Interpretando el patrón hemolítico creado por ciertas bacterias en el crecimiento en el agar sangre se pueden clasificar en 3 categorías diferentes. Los patrones se leen mediante la colocación de las placas hasta una fuente de luz en la que los rayos de luz vienen a través de la parte posterior de la placa (Buxton R., 2013).
El primer patrón es la hemólisis β en la que se observa una lisis completa de las células rojas de la sangre. Productos tóxicos creados por las bacterias destruyen las células rojas de la sangre que resulta en un lugar transparente del agar en la placa alrededor de la colonia de bacterias. Hay algunas bacterias que sólo exhiben el patrón hemolítica β sólo en ciertas condiciones tales como en niveles bajos o normales de oxígeno. Además, algunas bacterias exhiben producido lentamente o débilmente patrón hemolítico (Buxton R., 2013).
En hemólisis α, la bacteria convierte la hemoglobina, de las células rojas de la sangre en el medio, a metahemoglobina. Esta conversión cambia los medios de comunicación a un color verde o marrón. Esto se refiere a veces como la lisis parcial, ya que después de una inspección microscópica, membrana de los glóbulos rojos "están todavía intactas.Si el microorganismo se deja crecer continuamente en los medios de comunicación durante largos períodos de tiempo, incluso con hemólisis α las bacterias con el tiempo borrar el agar sangre (Buxton R., 2013).
γ hemólisis se refiere a una reacción nula en el que no se observa ninguna actividad hemolítica por el organismo en crecimiento. Medio que rodea las colonias bacterianas permanecen intactos (Buxton R., 2013).
La tinción de Gram
Las muestras de sangre y LCR se tratan con una solución de tinción y se observaron bajo un microscopio para las bacterias presentes en la muestra para determinar el tipo de bacterias por distinciones notables (Hohmann EL, 2012, Mayoclinic, 2013). Hay dos tipos de manchas: manchas simple y diferencial mancha (Benbasat J., 2012).
Manchas simples se utilizan para determinar el tamaño y forma de las bacterias. Estas manchas se pueden utilizar en cualquiera de los montajes húmedos o secos como el colorante de tinción no consigue enjuaga. Tinción diferencial separa las bacterias sobre la base de su composición de la pared celular en dos grupos separados: bacterias Gram-negativas y gram-positivas. Colorantes que contienen cromóforos se utilizan en la mancha diferencial para unirse a la pared celular de las bacterias. Debido a la presencia de un lípido de membrana externa de las bacterias gram-negativas, el tinte más oscuro inicial utilizado es lavar durante una etapa de lavado con etanol en el procedimiento de tinción. Un segundo colorante más ligero se utiliza para teñir las bacterias gram-negativas claras para visualizarse y diferenciarlos de las bacterias gram-positivas (Benbasat J., 2012).
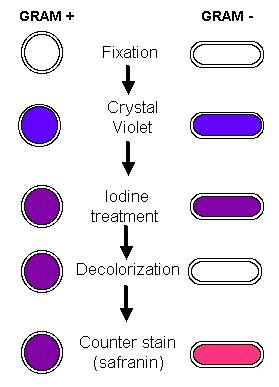 Figura 9. procedimiento de tinción de Gram
Figura 9. procedimiento de tinción de GramActividad Catalasa
Otra herramienta de diferenciación microbiólogo médico puede utilizar es la actividad de la catalasa que detecta la presencia de flavoproteínas en bacterias que permite al organismo a reducir el oxígeno a peróxido de hidrógeno o superóxido. Estos productos son altamente tóxicos para la célula y por lo tanto requieren la presencia de superóxido dismutasa y catalasa o peroxidasa, ya sea con el fin de sobrevivir en un ambiente con oxígeno. El objetivo de esta prueba es ver si las bacterias en la muestra contiene estas enzimas reductoras. El peróxido de hidrógeno se coloca en a las bacterias en la muestra en un portaobjetos para ver si se producen burbujas. El peróxido de hidrógeno se descompone enzimáticamente en agua y oxígeno (Benbasat J., 2012).
Oxidasa Actividad
Las bacterias producen citocromo oxidasa en presencia de oxígeno para oxidar el citocromo C. En esta prueba bioquímica, el citocromo c oxidado oxida un reactivo de oxidasa (tetrametil-p-fenileno o p-aminodimethylaniline oxalato) para producir un compuesto coloreado que es observable. El reactivo de oxidasa es un agente reductor cromogénico y cambiará de color (es decir, prueba positiva), cuando las bacterias con actividad oxidasa se coloca en el reactivo de ensayo / oxidasa (Benbasat J., 2012).
Látex Prueba de aglutinación
Esta prueba comprueba antisuero para ciertos anticuerpos o antígenos en una variedad de fluidos corporales, como CSF, sangre y orina. En el laboratorio, las muestras se mezclan con perlas de látex recubiertas con un anticuerpo o antígeno específico. La presencia de las sustancias sospechosas en la muestra analizada reaccionará a las perlas y hacer que se agrupen o aglutinan juntos. Resultados observables se recuperan alrededor de 15 minutos a una hora (Vorvick JL, 2015).
Quellung Reacción
Esta es una prueba bioquímica que puede identificar directamente varios en muestras clínicas, en base a la observación microscópica. Los anticuerpos específicos que se unen a cápsulas bacterianas se añaden a las muestras. Unión positiva del anticuerpo a las bacterias capsulares se agrandar el tamaño de las bacterias (iethe hincha cápsula en la presencia de antisuero homólogo). Los antisueros contra todos los serotipos de S. pneumoniae, H. influenzae tipo b, y N. meningitidis grupos A y C están disponibles (Levinson W., 2014).
4. Resultados
Resultados para neumococo
El crecimiento de neumococo en agar es inhibida por optoquina y la bilis. Por otra parte, neumococo colonias son bilis soluble (Levinson W., 2014). En agar sangre neumocococrecer durante la noche en redondo, brillante colonias que van de 0,5 a 2,0 mm (Ryan KJ, 2014). Esta bacteria exposiciones alfa actividades hemolíticas (Levinson W., 2014).
De tinción frotis de esputo podemos ver bacterias cocos "en forma de lanceta," gram-positivas que pueden ocurrir en parejas o en cadenas cortas (Levinson W., 2014).
Estas bacterias son catalasa-negativos (Levinson W., 2014).
El neumococo poseen una cápsula de polisacárido prominente. Pruebas de antisuero pueden mostrar positivo para antígenos capsulares en CSF y polisacárido C en orina (Levinson W., 2014).
Reacciones Quellung ocurren si este patógeno está presente (Levinson W., 2014).
Resultados para Haemophilus
El crecimiento de Haemophilus requiere factores X y V, por lo que sólo puede crecer en agar de chocolate como opacos-crema a gris colonias (Acharya T., 2013, Levinson W., 2014). Por otra parte, Haemophilus no puede crecer en agar sangre de oveja a menos que sea alrededor de estafilococos colonias (Brooks GF, 2013, Levinson W., 2014).Después de 24 horas de incubación, Haemophilus colonias se describen como plana, marrón grisáceo con diámetros de 1-2 (Brooks GF, 2013). Los hidratos de carbono en los medios de comunicación se fermentan mal e irregularmente por Haemophilus (Brooks GF, 2013). Haemophilus crecimiento en exhibición agar chocolate γ actividad hemolítica (Acharya T., 2013).
Procedimientos de tinción de muestras frotis revelarán pequeñas bacterias en forma de bastoncillos gram-negativas en el tamaño de 1,5 micras (Brooks GF, 2013, Levinson W., 2014). Morfología de las bacterias cambios dependiendo de la duración de la incubación y el tipo de medios de comunicación. Después de 6-8 horas de crecimiento, las pequeñas formas cocobacilares predominan, mientras que más tarde se enfrentará a una morfología en forma de barra (Brooks GF, 2013)
Culturas jóvenes que han estado creciendo durante unos 6 a 18 horas, poseen una cápsula definida (Brooks). Realización de pruebas de aglutinación de látex en muestras de LCR o suero detectarán Hib antígenos capsulares (Levinson W., 2014).
Reacciones Quellung ocurren si este patógeno está presente (Levinson W., 2014).
Hib también puede detectarse inmunológicamente utilizando kits comerciales que confirman las presencias de antígenos Hib en el LCR. Sin embargo, estos kits no son ampliamente utilizados debido a la sensibilidad más baja en la prueba (Brooks GF, 2013)
Resultados para Listeria
Listeria a partir de muestras de sangre, crecer como pequeñas colonias en agar sangre y exhibirá una débil actividad hemolítica β (Hohmann EL 2012, Levinson W., 2014, Ryan KJ, 2014). Hemolisina β del patógeno es fundamental para mantener su patogenicidad (Hohmann EL, 2012).
En frotis teñidos de muestras de LCR, son gram-positivos y tienen una morfología en forma de barra que se asemeja mucho Corynebacterium (Levinson W., 2014).
A fin de distinguir Listeria de otros patógenos, las colonias se pueden cultivar en temperaturas por debajo de 30 ° C. Listeria son capaces de crecer a temperaturas más bajas debido a la inducción de enzimas helicasa de ARN a bajas temperaturas. En medios fluidos, Listeria crece en temperaturas inferiores a 30 ° C se mostrará la motilidad volteo, un movimiento flagelar impulsada (Levinson W., 2014, Ryan KJ, 2014). Estas bacterias son catalasa-positivos que los distingue más lejos de otras especies de bacterias, como los estreptococos (Ryan KJ, 2014).
Resultados para Meningococo
El meningococo crece en agar chocolate después de 18 horas y produce colonias que son grisáceas, redondos, convexos, y húmedas, brillantes colonias lisas con un borde claramente definido (Acharya T., 2013, Brooks GF, 2013, Ryan KJ, 2014). El meningococo crece mejor en las proteínas de origen animal que contiene los medios de comunicación y en una atmósfera que contiene 5% de dióxido de carbono (Brooks GF, 2013). El dióxido de carbono no es necesaria, pero no mejorará la capacidad de esta bacteria para crecer (Ryan KJ, 2014). Sin embargo, el crecimiento se inhibe por la presencia de ácidos grasos o sales en los medios de comunicación. Las muestras de sangre son generalmente cultivadas en medios de comunicación sin sulfonato de sodio polyanethol, mientras que las muestras de LCR se cultivan en agar chocolate; tanto agar produce el crecimiento de cultivo puro en los medios de comunicación (Brooks GF, 2013). Estas bacterias exhiben actividad hemolítica γ en agar chocolate (Acharya T., 2013).Por otra parte, este patógeno fermenta la maltosa (Levinson W., 2014).
La tinción de frotis de LCR revelaría "riñón de frijol" bacterias de forma diplococos gramnegativos. Una cápsula de polisacáridos grande también se puede observar (Levinson W., 2014). Estas bacterias son inmóviles y son de aproximadamente 0,8 m de diámetro. Centrifugados frotis de LCR se suele encontrar esta bacteria dentro o alrededor de los leucocitos polimorfonucleares (Brooks GF, 2013).
 Figura 20. El meningococo gramo manchada gram-negativa dentro de una célula
Figura 20. El meningococo gramo manchada gram-negativa dentro de una célula
El meningococo es una bacteria (Levinson W., 2014) positivos oxidasa.
Realización de pruebas de aglutinación de látex en muestras de LCR o suero detectarán meningococo antígenos capsulares (Brooks GF, 2013, Levinson W., 2014).



No hay comentarios:
Publicar un comentario