La 7-aminoactinomicina D ( 7-AAD ) es un compuesto químico fluorescente con una fuerte afinidad por el ADN . Se utiliza como un marcador fluorescente para el ADN en microscopía de fluorescencia y citometría de flujo . Se intercala en el ADN de doble cadena, con una alta afinidad por las regiones ricas en GC, [2] por lo que es útil para los estudios de bandas de cromosomas .
Aplicaciones [ editar ]
Con un máximo de absorción a 546 nm, 7-AAD se excita de manera eficiente utilizando un láser de helio-neón de 543 nm ; también se puede excitar con una eficiencia algo menor utilizando un láser de argón de 488 nm o 514 nm. Su emisión tiene un cambio de Stokesmuy grande con un máximo en el rojo profundo: 647 nm. Por lo tanto, 7-AAD es compatible con la mayoría de los fluoróforos azules y verdes, e incluso con muchos fluoróforos rojos, en aplicaciones de varios colores.
7-AAD no pasa fácilmente a través de las membranas celularesintactas ; Si se va a utilizar como una mancha para obtener imágenes de fluorescencia de ADN, la membrana celular debe permeabilizarse o romperse. Este método se puede utilizar en combinación con la fijación con formaldehído de las muestras.
7-AAD también se utiliza como una tinción de viabilidad celular . Las células con membranas comprometidas se teñirán con 7-AAD, mientras que las células vivas con membranas celulares intactas permanecerán oscuras.
Actinomicina D [ editar ]
El compuesto relacionado actinomicina D no es fluorescente, pero se une al ADN de la misma manera que 7-AAD. Su absorbancia cambia cuando se une al ADN, y se puede utilizar como una mancha en la microscopía de transmisión convencional.
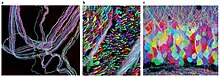
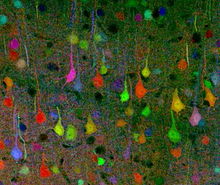
Brainbow es un proceso mediante el cual las neuronas individuales en el cerebro pueden distinguirse de las neuronas vecinas que utilizan proteínas fluorescentes. Al expresar aleatoriamente diferentes proporciones de rojo, verde y azul derivados de la proteína verde fluorescente en neuronas individuales, es posible marcar cada neurona con un color distintivo. Este proceso ha sido una importante contribución al campo de la conectómica o al estudio de las conexiones neuronales en el cerebro. El estudio de las vías neurales también es conocido como hodología por los neuroanatomistas anteriores.
La técnica fue desarrollada originalmente en la primavera de 2007 por un equipo liderado por Jeff W. Lichtman y Joshua R. Sanes, ambos profesores de biología molecular y celular en la Universidad de Harvard . Su demostración de la técnica en ratones apareció por primera vez en la edición del 1 de noviembre de 2007 de la revista Nature . [1] La técnica original recientemente ha sido adaptado para su uso con otros organismos modelo, incluyendo Drosophila melanogaster , Caenorhabditis elegans [ cita requerida ] , y Arabidopsis thaliana . [2]
Si bien las técnicas de etiquetado anteriores permitieron el mapeo de solo unas pocas neuronas, este nuevo método permite que más de 100 neuronas mapeadas de manera diferente se iluminen de manera simultánea y diferencial de esta manera. Las imágenes resultantes pueden ser bastante sorprendentes y han ganado premios en concursos de fotografía científica [ cita requerida ] .
Historia y desarrollo [ editar ]
La técnica de neuroimagen Brainbow fue desarrollada inicialmente por un equipo de investigadores en la Universidad de Harvard en 2007. [1] En ese momento estaban trabajando en la Universidad de Washington en St. Louis. Este grupo particular de científicos fue dirigido por los profesores Jeff W. Lichtman y Joshua R. Sanes, quienes se especializan en biología molecular y celular y son muy conocidos por su trabajo. El equipo construyó Brainbow mediante un proceso de dos pasos: primero, se generó una construcción genética específica que podría recombinarse en múltiples disposiciones para producir uno de tres o cuatro colores basados en las proteínas fluorescentes particulares (XFP) que se están implementando. [3] A continuación, múltiples copias del mismo transgénico.El constructo se insertó en el genoma de la especie objetivo, lo que dio como resultado la expresión aleatoria de diferentes proporciones de XFP y, posteriormente, provocó que diferentes células exhibieran una variedad de tonos de colores. [3]
Brainbow se creó originalmente como una mejora con respecto a las técnicas de neuroimagen más tradicionales , como la tinción de Golgi y la inyección de tinte, que presentaban graves limitaciones para los investigadores en su capacidad para visualizar la arquitectura intrincada de los circuitos neuronales en el cerebro . [1] Si bien las técnicas más antiguas solo podían teñir células con una gama limitada de colores, a menudo utilizan ratones transgénicos bicolores y tricolores para revelar información limitada con respecto a las estructuras neuronales, Brainbow es mucho más flexible en cuanto a su capacidad para etiquetar de forma fluorescente neuronas individuales con hasta aproximadamente 100 tonos diferentes para que los científicos puedan identificar e incluso diferenciar entreProcesos dendríticos y axonales . [3] Al revelar información tan detallada sobre la conectividad neuronal y los patrones, a veces incluso en vivo, los científicos a menudo pueden inferir información sobre las interacciones neuronales y su impacto posterior sobre el comportamiento y la función. Por lo tanto, Brainbow llenó el vacío dejado por los métodos de neuroimagen anteriores.
Con el reciente advenimiento de Brainbow en la neurociencia , los investigadores ahora pueden construir mapas específicos de circuitos neuronales e investigar mejor cómo se relacionan con varias actividades mentales y sus comportamientos conectados (es decir, Brainbow revela información sobre las interconexiones entre las neuronas y sus interacciones subsiguientes que afectan funcionalidad general del cerebro). Como una extrapolación adicional de este método, Brainbow también se puede usar para estudiar trastornos neurológicos y psicológicos mediante el análisis de las diferencias en los mapas neuronales. [3]
Métodos [ editar ]
Las técnicas de Brainbow se basan en la recombinación de Cre-Lox , en la que la proteína Cre recombinase impulsa la inversión o la escisión del ADN entre los sitios loxP. El método original de Brainbow incluye Brainbow-1 y Brainbow-2, que utilizan diferentes formas de recombinación cre / lox. Brainbow-3, una versión modificada de Brainbow-1, se desarrolló en 2013. [4] Para todos los subtipos de Brainbow, la expresión de un XFP dado es un evento estocástico o aleatorio.
Brainbow-1 utiliza construcciones de ADN con diferentes genes de proteínas fluorescentes (XFP) separados por formas mutantes y canónicas de loxP. Esto crea un conjunto de posibilidades de escisión mutuamente excluyentes, ya que la recombinación mediada por cre se produce solo entre sitios loxP idénticos. [1] Después de que se produce la recombinación, la proteína fluorescente que queda directamente después de que el promotor se expresa de manera única. Por lo tanto, una construcción con cuatro XFP separados por tres sitios loxP diferentes, tres eventos de escisión y la construcción original puede producir cuatro proteínas fluorescentes diferentes. [3]
Brainbow-2 usa la excisión y la inversión de Cre para permitir múltiples posibilidades de expresión en una construcción dada. En un segmento de ADN con dos XFP de orientación opuesta, Cre inducirá un evento de inversión aleatorio que deja a una proteína fluorescente en la orientación adecuada para la expresión. Si dos de estas secuencias invertibles están alineadas, son posibles tres eventos de inversión diferentes. Cuando también se consideran los eventos de escisión, una de las cuatro proteínas fluorescentes se expresará para una combinación dada de escisiones e inversiones de Cre.
Brainbow-3 conserva el formato loxP de Brainbow-1, pero reemplaza los genes RFP, YFP y CFP con mOrange2, EGFP y mKate2. Se eligió mO2, EGFP y mK2 debido a que sus espectros de emisión y excitación fluorescentes se superponen mínimamente, y porque comparten una homología de secuencia mínima, lo que permite el diseño de anticuerpos selectivos que pueden usarse para detectarlos en protocolos inmunohistoquímicos . Brainbow-3 también aborda el problema del llenado desigual de las neuronas con XFP mediante el uso de derivados farnesilados de los XFP, que se distribuyen de manera más uniforme hacia las membranas neuronales. [4]
Brainbow se implementa in vivo al cruzar dos cepas de organismos transgénicos : una que expresa la proteína Cre y otra que se ha transfectado con varias versiones de un constructo loxP / XFP. El uso de múltiples copias del transgén permite que los XFP se combinen de una manera que puede dar uno de aproximadamente 100 colores diferentes. [3] Por lo tanto, cada neurona está marcada con un tono diferente en función de su expresión combinatoria y estocástica dada de proteínas fluorescentes.
Con el fin de dilucidar los patrones de expresión de XFP diferenciales en una forma visible, se realizan imágenes de cortes cerebrales con microscopía confocal . Cuando se expone a un fotón con su longitud de onda de excitación particular, cada fluoróforo emite una señal que se recoge en un canal rojo, verde o azul, y la combinación de luz resultante se analiza con un software de análisis de datos. [1] La superposición de neuronas de colores diferenciales permite el desenredo visual de circuitos neuronales complicados.
Brainbow ha sido probado predominantemente en ratones hasta la fecha; sin embargo, la técnica básica descrita anteriormente también se ha modificado para su uso en estudios más recientes desde la introducción del método original introducido en 2007.
Ratones [ editar ]
El cerebro del ratón tiene 75,000,000 neuronas y es más similar a un cerebro humano que a la drosophila y otros organismos comúnmente utilizados para modelar esta técnica, como C. elegans . Los ratones fueron los primeros organismos en los que se utilizó con éxito el método de neuroimagen Brainbow. [1] Livet et al. (2007) desarrollaron dos versiones de ratones Brainbow usando Brainbow-1 y Brainbow-2, que se describen anteriormente. [1] Al usar estos métodos para crear un mapa completo y rastrear los axones de un músculo del ratón, es necesario recopilar decenas de miles de imágenes y compilarlas en pilas para crear un esquema completo. [3]Entonces es posible rastrear cada axón motor y sus contactos sinápticos para construir un conectoma completo del músculo.
Más ejemplos de neuronas examinadas utilizando la técnica Brainbow en ratones transgénicos se encuentran en el nervio motor que inerva los músculos de la oreja, tractos axónicos en el tronco cerebral y el giro del hipocampo dentado . [3]
Drosophila [ editar ]
La complejidad del cerebro de Drosophila, que consta de alrededor de 100.000 neuronas, lo convierte en un excelente candidato para la implementación de técnicas de neurofisiología y neurociencia como Brainbow. De hecho, Stefanie Hampel et al. (2011) combinaron Brainbow junto con herramientas de selección genética para identificar neuronas individuales dentro del cerebro de Drosophila y varios linajes neuronales. [5] Una de las herramientas de selección genética fue un GAL4 / UASSistema de expresión binaria que controla la expresión de UAS-Brainbow y dirige la expresión a pequeños grupos de neuronas. El uso de los métodos de 'Salida hacia fuera' aumentó la resolución celular de la estructura del reportero. La expresión de las proteínas fluorescentes, como en el Brainbow original, dependía de la recombinación de Cre correspondiente a los sitios lox emparejados. Hampel et al. (2011) también desarrollaron su propia variación de Brainbow (dBrainbow), basada en el marcado de anticuerpos de epítopos en lugar de fluorescencia endógena. [5]Dos copias de su construcción producen seis colores brillantes y separables. Esto, junto con las simplificaciones en la asignación de colores, les permitió observar las trayectorias de cada neurona en largas distancias. Específicamente, rastrearon las neuronas motoras desde el lóbulo de la antena hasta las uniones neuromusculares, lo que les permite identificar los objetivos musculares específicos de las neuronas individuales.
En última instancia, esta técnica brinda la capacidad de mapear eficazmente los circuitos neuronales en Drosophila para que los investigadores puedan descubrir más información sobre la estructura cerebral de este invertebrado y cómo se relaciona con su comportamiento posterior.
Limitaciones [ editar ]
Al igual que con cualquier técnica de neuroimagen , Brainbow tiene una serie de limitaciones que se derivan de los métodos necesarios para realizarla. Por ejemplo, el proceso de reproducción de al menos dos cepas de animales transgénicos a partir de células madre embrionarias es a la vez complejo y requiere mucho tiempo. Incluso si dos especies transgénicas se crean con éxito, no todos sus descendientes mostrarán la recombinación. Por lo tanto, esto requiere una planificación extensa antes de realizar un experimento. [3]
Además, debido a la naturaleza aleatoria en la expresión de las proteínas fluorescentes, los científicos no pueden controlar con precisión el etiquetado de los circuitos neuronales, lo que puede resultar en la mala identificación de neuronas específicas.
El uso del cerebro en las poblaciones de mamíferos también se ve obstaculizado por la increíble diversidad de neuronas del sistema nervioso central . La gran densidad de las neuronas junto con la presencia de largas extensiones de axones dificulta la visualización de regiones más grandes del SNC con alta resolución. Brainbow es más útil cuando se examina la resolución de una sola célula en el contexto de un entorno multicelular complejo. Sin embargo, debido a los límites de resolución de la microscopía óptica , la identificación concluyente de las conexiones sinápticas entre las neuronas no se logra fácilmente. Este problema se evita de alguna manera mediante el uso de marcadores sinápticos para complementar el uso de la microscopía óptica en la visualización de conexiones sinápticas.
La obtención de imágenes de calcio es una técnica científica que generalmente se lleva a cabo en investigaciones diseñadas para mostrar el estado del calcio (Ca 2+ ) de una célula , tejido o medio aislado . Las técnicas de obtención de imágenes de calcio aprovechan los llamados indicadores de calcio , moléculas fluorescentes que pueden responder a la unión de los iones Ca 2+ al cambiar sus propiedades de fluorescencia. Existen dos clases principales de indicadores de calcio : indicadores químicos e indicadores de calcio codificados genéticamente (GECI). Las imágenes de calcio pueden utilizarse para sondear ópticamente el calcio intracelular en animales vivos. [1] Esta técnica ha permitido estudios de señalización de calcio. en una amplia variedad de tipos de células y actividad neuronal en cientos de neuronas y células gliales dentro de los circuitos neuronales.
Indicadores químicos [ editar ]
Los indicadores químicos son pequeñas moléculas que pueden quelar los iones de calcio. Todas estas moléculas se basan en un homólogo de EGTA llamado BAPTA , con alta selectividad para los iones de calcio (Ca 2+ ) frente a los iones de magnesio (Mg 2+ ).
Estos tintes se usan a menudo con los grupos quelantes carboxiloenmascarados como ésteres de acetoximetilo , para hacer que la molécula sea lipófila y para permitir la entrada fácil en la célula. Una vez que esta forma del indicador esté en la célula, las esterasas celulares liberarán los grupos carboxilo y el indicador podrá unirse al calcio. La forma ácida libre de los colorantes (es decir, sin la modificación del éster de acetoximetilo) también se puede inyectar directamente en las células a través de un microelectrodo o micropipeta que elimina las dudas sobre el compartimento celular que contiene el tinte (el éster de acetoximetilo también puede entrar en el retículo endoplásmico y la mitocondria). ). Encuadernación de un CaEl ión 2+ a una molécula indicadora fluorescente conduce a un aumento en el rendimiento cuántico de la fluorescencia o al cambio de longitud de onda de excitación / emisión . Se utilizan indicadores químicos de Ca 2+ químicos individuales para las mediciones de calcio citosólico en una amplia variedad de preparaciones celulares. La primera imagen en tiempo real (tasa de video) de Ca 2+ se llevó a cabo en 1986 en células cardíacas utilizando cámaras de video intensificadas. [2] El desarrollo posterior de la técnica con microscopios confocales de barrido láser reveló señales de Ca 2+ subcelulares en forma de chispas de Ca 2+ y Ca 2+blips Las respuestas relativas de una combinación de indicadores fluorescentes químicos de Ca 2+ también se utilizaron para cuantificar transitorios de calcio en orgánulos intracelulares como las mitocondrias . [3]
Las imágenes de calcio, también conocidas como mapeo de calcio, también se utilizan para realizar investigaciones en tejido miocárdico. [4] El mapeo de calcio es una técnica ubicua utilizada en corazones enteros, aislados, como las especies de ratones, ratas y conejos.
Indicador de calcio codificados genéticamente [ editar ]
Estos indicadores son proteínas fluorescentes derivadas de la proteína fluorescente verde (GFP) o sus variantes (p. Ej., GFP permutada circularmente, YFP, CFP), fusionadas con calmodulina (CaM) y el dominio M13 de la cinasa de cadena ligera de la miosina , que es capaz de unirse a la CaM . Alternativamente, las variantes de GFP se fusionan con la proteína de unión a calcio troponina C (TnC), aplicando el mecanismo de FRET (transferencia de energía de resonancia de Förster) para la modulación de la señal.
Los indicadores codificados genéticamente no necesitan cargarse en las células, en lugar de eso, los genes que codifican estas proteínas pueden transfectarse fácilmente a líneas celulares. También es posible crear animales transgénicos que expresan el tinte en todas las células o selectivamente en ciertos subtipos celulares. Los GECI se han utilizado en los estudios de neuronas [5] , [6] células T , [7] cardiomiocitos , [8] etc.
| GECI | Año | Detectando | Informes | Precursor |
|---|---|---|---|---|
| Camaleones [9] | 1997 | Calmodulina | Par de FRET: BFP o CFP, y GFP o YFP | - |
| FIP-CB SM[10] | 1997 | Calmodulina | Par de FRET: BFP y RFP | - |
| Pericams[11] | 2000 | Calmodulina | cpGFP | - |
| GCaMP [12] | 2000 | Calmodulina | cpEGFP | - |
| TN-L15 [13] | 2004 | Músculo esquelético de pollo modificado troponina C | Par de FRET: YFP (Citrine) y CFP (Cerulean) | - |
| TN-humTnC[13] | 2004 | Troponina C cardíaca humana | Par de FRET: YFP (Citrine) y CFP (Cerulean) | - |
| TN-XL [14] | 2006 | Músculo esquelético de pollo modificado troponina C | Par de FRET: YFP permutado (Citrine) y CFP (Cerulean) | TN-L15 |
| TN-XXL [15] | 2008 | CsTnC modificado en TN-XL | Par de FRET: YFP permutado (Citrine) y CFP (Cerulean) | TN-XL |
| De Twitch[16] | 2014 | Troponina c | Par de FRET (varios de dos FPs) | - |
Uso [ editar ]
Independientemente del tipo de indicador utilizado, el procedimiento de imagen es generalmente muy similar. Las células aisladas cargadas con un indicador o que lo expresan en el caso de GECI, pueden verse con un microscopio de fluorescencia y capturarlas con una cámara Scientific CMOS (sCMOS) [17] o CCD . Losmicroscopios confocales y de dos fotones brindan una capacidad de corte mejorada para que las señales de calcio se puedan resolver en microdominios o (por ejemplo) boutons sinápticos . Las imágenes se analizan midiendo los cambios en la intensidad de la fluorescencia para una sola longitud de onda o dos longitudes de onda expresadas como una relación (indicadores ratiométricos). Las intensidades y relaciones de fluorescencia derivadas se representan frente a los valores calibrados de Ca 2+ conocidoNiveles para aprender la concentración de Ca 2+ . Los métodos de microscopía de campo de luz [18] extienden la lectura funcional de las capacidades de actividad neuronal en volúmenes 3D.




No hay comentarios:
Publicar un comentario