Emparejamiento de bases
En una doble hélice de ADN, cada tipo de nucleobase en una hebra se une con un solo tipo de nucleobase en la otra hebra. Esto se denomina apareamiento de bases complementarias . Aquí, las purinas forman enlaces de hidrógeno a pirimidinas, con enlaces de adenina solo a timina en dos enlaces de hidrógeno, y enlaces de citosina solo a guanina en tres enlaces de hidrógeno. Esta disposición de dos nucleótidos que se unen entre sí en la doble hélice se llama un par de bases de Watson-Crick. Otro tipo de emparejamiento de bases es el emparejamiento de bases de Hoogsteen, donde se forman dos enlaces de hidrógeno entre la guanina y la citosina. [48] Como los enlaces de hidrógeno no son covalentes, se pueden romper y volver a unir con relativa facilidad. Las dos cadenas de ADN en una doble hélice pueden así ser separadas como una cremallera, ya sea por una fuerza mecánica o por una alta temperatura . [49] Como resultado de esta complementariedad de pares de bases, toda la información en la secuencia de doble cadena de una hélice de ADN se duplica en cada cadena, lo que es vital en la replicación del ADN. Esta interacción reversible y específica entre pares de bases complementarias es crítica para todas las funciones del ADN en los organismos. [7]
 |
 |
Arriba, un par de bases GC con tres enlaces de hidrógeno . Abajo, un par de bases AT con dos enlaces de hidrógeno. Los enlaces de hidrógeno no covalentes entre los pares se muestran como líneas discontinuas.
Los dos tipos de pares de bases forman diferentes números de enlaces de hidrógeno, AT formando dos enlaces de hidrógeno y GC formando tres enlaces de hidrógeno (ver figuras, a la derecha). El ADN con alto contenido de GC es más estable que el ADN con bajo contenido de GC.
Como se señaló anteriormente, la mayoría de las moléculas de ADN son en realidad dos cadenas de polímero, unidas entre sí de forma helicoidal por enlaces no covalentes; esta estructura de doble cadena ( dsDNA ) se mantiene en gran medida por las interacciones de apilamiento de bases intrastrand, que son más fuertes para las pilas de G, C. Las dos cadenas pueden separarse, un proceso conocido como fusión, para formar dos moléculas de ADN de cadena simple ( ADNss ). La fusión se produce a alta temperatura, baja sal y alto pH (el bajo pH también funde el ADN, pero como el ADN es inestable debido a la depuración del ácido, rara vez se usa un bajo pH).
La estabilidad de la forma del dsDNA depende no solo del contenido de GC (% G, pares de bases C) sino también de la secuencia (ya que el apilamiento es específico de la secuencia) y también la longitud (las moléculas más largas son más estables). La estabilidad se puede medir de varias maneras; una forma común es la "temperatura de fusión", que es la temperatura a la cual el 50% de las moléculas ds se convierten en moléculas ss; La temperatura de fusión depende de la fuerza iónica y de la concentración de ADN. Como resultado, es tanto el porcentaje de pares de bases GC como la longitud total de una doble hélice de ADN lo que determina la fuerza de la asociación entre las dos cadenas de ADN. Las hélices de ADN largas con un alto contenido de GC tienen hebras de interacción más fuertes, mientras que las hélices cortas con alto contenido de AT tienen hebras de interacción más débil. [50]En biología, las partes de la doble hélice de ADN que deben separarse fácilmente, como la caja TATAAT Pribnow en algunos promotores , tienden a tener un alto contenido de AT, lo que hace que las hebras sean más fáciles de separar. [51]
En el laboratorio, la fuerza de esta interacción se puede medir mediante la búsqueda de la temperatura necesaria para romper los enlaces de hidrógeno, su temperatura de fusión (también llamado T m valor). Cuando todos los pares de bases en una doble hélice de ADN se funden, las cadenas se separan y existen en solución como dos moléculas completamente independientes. Estas moléculas de ADN de cadena simple no tienen una forma común única, pero algunas conformaciones son más estables que otras. [52]
Sentido y antisentido
Una secuencia de ADN se denomina secuencia de "sentido" si es la misma que la de una copia de ARN mensajero que se traduce en proteína. [53] La secuencia en la cadena opuesta se llama secuencia "antisentido". Las secuencias tanto sentido como antisentido pueden existir en diferentes partes de la misma cadena de ADN (es decir, ambas cadenas pueden contener secuencias tanto sentido como antisentido). Tanto en procariotas como en eucariotas, se producen secuencias de ARN antisentido, pero las funciones de estos ARN no están del todo claras. [54] Una propuesta es que los ARN antisentido participan en la regulación de la expresión génica através del emparejamiento de bases ARN-ARN. [55]
Unas pocas secuencias de ADN en procariotas y eucariotas, y más en plásmidos y virus , borran la distinción entre cadenas sentido y antisentido al tener genes superpuestos . [56] En estos casos, algunas secuencias de ADN cumplen una doble función, ya que codifican una proteína cuando se lee a lo largo de una cadena, y una segunda proteína cuando se leen en la dirección opuesta a lo largo de la otra cadena. En bacterias , esta superposición puede estar involucrada en la regulación de la transcripción de genes, [57] mientras que en los virus, los genes superpuestos aumentan la cantidad de información que puede codificarse dentro del pequeño genoma viral. [58]
Superenrollado
El ADN se puede torcer como una cuerda en un proceso llamado supercoiling de ADN . Con el ADN en su estado "relajado", una hebra generalmente rodea el eje de la doble hélice una vez cada 10,4 pares de bases, pero si el ADN se tuerce, las hebras se enrollan más o menos. [59] Si el ADN se tuerce en la dirección de la hélice, esto es un supercoiling positivo, y las bases se mantienen más juntas entre sí. Si están torcidos en la dirección opuesta, esto es un supercoiling negativo, y las bases se separan más fácilmente. En la naturaleza, la mayoría del ADN tiene un ligero supercoil negativo que es introducido por las enzimas llamadas topoisomerasas . [60]Estas enzimas también son necesarias para aliviar las tensiones de torsión introducidas en las cadenas de ADN durante procesos como la transcripción y la replicación de ADN . [61]
Estructuras de ADN alternativas
El ADN existe en muchas posibles conformaciones que incluyen lasformas A-DNA , B-DNA y Z-DNA , aunque solo se han observado directamente B-DNA y Z-DNA en organismos funcionales. [14] La conformación que adopta el ADN depende del nivel de hidratación, la secuencia del ADN, la cantidad y la dirección del superenrollamiento, las modificaciones químicas de las bases, el tipo y la concentración de iones metálicos y la presencia de poliaminasen solución. [62]
Los primeros informes publicados de los patrones de difracción de rayos X del ADN-A , y también del ADN-B, utilizaron análisis basados en las transformaciones de Patterson que proporcionaron solo una cantidad limitada de información estructural para las fibras orientadas del ADN. [63] [64] Luego, Wilkins et al. Propusieron un análisis alternativo . , en 1953, para los patrones de dispersión de difracción de rayos X de ADN-B in vivo de fibras de ADN altamente hidratadas en términos de cuadrados de funciones de Bessel . [65] En la misma revista, James Watson y Francis Crick presentaron sus modelos moleculares.Análisis de los patrones de difracción de rayos X del ADN para sugerir que la estructura era una doble hélice. [9]
Aunque la forma de B-ADN es más común en las condiciones encontradas en las células [66], no es una conformación bien definida, sino una familia de conformaciones de ADN relacionadas [67] que se producen en los altos niveles de hidratación presentes en las células. Sus correspondientes patrones de difracción y dispersión de rayos X son característicos de los paracristales moleculares con un grado significativo de trastorno. [68] [69]
En comparación con el ADN-B, la forma del ADN-A es una espiral más ancha hacia la derecha , con un surco pequeño y poco profundo y un surco mayor más estrecho y profundo. La forma A se produce en condiciones no fisiológicas en muestras de ADN parcialmente deshidratadas, mientras que en la célula puede producirse en pares híbridos de cadenas de ADN y ARN, y en complejos de enzima-ADN. [70] [71] Los segmentos de ADN donde las bases han sido modificados químicamente por metilación pueden sufrir un cambio más grande en la conformación y adoptar la forma Z . Aquí, las hebras giran alrededor del eje helicoidal en una espiral a la izquierda, lo opuesto a la forma B más común. [72]Estas estructuras inusuales pueden ser reconocidas por proteínas específicas de unión a Z-ADN y pueden estar involucradas en la regulación de la transcripción. [73]
Química alternativa del ADN
Durante muchos años, los exobiólogos han propuesto la existencia de una biosfera de sombra , una biosfera microbiana postulada de la Tierra que utiliza procesos bioquímicos y moleculares radicalmente diferentes a los de la vida actual. Una de las propuestas fue la existencia de formas de vida que utilizan arsénico en lugar de fósforo en el ADN . Se anunció un informe en 2010 sobre la posibilidad de la bacteria GFAJ-1 , [74] [74] [75], aunque se cuestionó la investigación, [75] [76] y la evidencia sugiere que la bacteria impide activamente la incorporación de arsénico en El esqueleto del ADN y otras biomoléculas. [77]
Estructuras cuadruplex
En los extremos de los cromosomas lineales hay regiones especializadas de ADN llamadas telómeros . La función principal de estas regiones es permitir que la célula replique los extremos del cromosoma utilizando la enzima telomerasa , ya que las enzimas que normalmente replican el ADN no pueden copiar los extremos extremos 3 'de los cromosomas. [78] Estas tapas cromosómicas especializadas también ayudan a proteger los extremos del ADN y evitan que los sistemas de reparación de ADN en la célula los traten como daños que deben corregirse. [79] En las células humanas , los telómeros suelen ser longitudes de ADN monocatenario que contienen varios miles de repeticiones de una secuencia TTAGGG simple. [80]
Estas secuencias ricas en guanina pueden estabilizar los extremos de los cromosomas formando estructuras de conjuntos apilados de unidades de cuatro bases, en lugar de los pares de bases habituales que se encuentran en otras moléculas de ADN. Aquí, cuatro bases de guanina forman una placa plana y estas unidades planas de cuatro bases se apilan una encima de la otra para formar una estructura estable de cuadrúplex de G. [82] Estas estructuras están estabilizadas por enlaces de hidrógeno entre los bordes de las bases y la quelación de un ion metálico en el centro de cada unidad de cuatro bases. [83] También se pueden formar otras estructuras, con el conjunto central de cuatro bases provenientes de una sola hebra doblada alrededor de las bases, o varias hebras paralelas diferentes, cada una de las cuales contribuye con una base a la estructura central.
Además de estas estructuras apiladas, los telómeros también forman estructuras de bucle grandes llamadas bucles telómeros o bucles-T. Aquí, el ADN de una sola hebra se enrolla alrededor de un círculo largo estabilizado por proteínas de unión a telómeros. [84] Al final del bucle T, el ADN del telómero monocatenario se mantiene en una región del ADN bicatenario por medio de la cadena de telómeros que interrumpe el ADN de doble hélice y el emparejamiento de bases en una de las dos cadenas. Esta estructura de triple cadena se llama un bucle de desplazamiento o D-loop . [82]
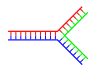 |  |
| Rama única | Múltiples ramas |
El ADN ramificado puede formar redes que contienen múltiples ramas.
ADN ramificado
En el ADN, el desgaste se produce cuando existen regiones no complementarias al final de una doble cadena de ADN que de otro modo sería complementaria. Sin embargo, el ADN ramificado puede ocurrir si se introduce una tercera cadena de ADN y contiene regiones contiguas capaces de hibridar con las regiones deshilachadas de la doble cadena preexistente. Aunque el ejemplo más simple de ADN ramificado involucra solo tres cadenas de ADN, también son posibles complejos que involucran cadenas adicionales y múltiples ramas. [85] ElADN ramificado se puede utilizar en nanotecnología para construir formas geométricas, consulte la sección sobre usos en tecnología a continuación.
Bases artificiales
Se han sintetizado varias nucleobases artificiales que se han incorporado con éxito en el análogo de ADN de ocho bases denominado ADN de Hachimoji . Apodadas S, B, P y Z, estas bases artificiales son capaces de unirse entre sí de una manera predecible (S – B y P – Z), mantienen la estructura de doble hélice del ADN y se transcriben al ARN. Su existencia implica que no hay nada especial en las cuatro nucleobases naturales que evolucionaron en la Tierra.

No hay comentarios:
Publicar un comentario