| Parte de una serie en |
| Genética |
|---|
 |
| Componentes clave |
| Historia y temas |
| Investigación |
| Medicina personalizada |
| Medicina personalizada |
La secuenciación del ADN es el proceso de determinar la secuencia de ácido nucleico , el orden de los nucleótidos en el ADN. Incluye cualquier método o tecnología que se utiliza para determinar el orden de las cuatro bases: adenina , guanina , citosina y timina . El advenimiento de los métodos rápidos de secuenciación de ADN ha acelerado enormemente la investigación y el descubrimiento biológico y médico. [1]
El conocimiento de las secuencias de ADN se ha vuelto indispensable para la investigación biológica básica, y en numerosos campos aplicados como el diagnóstico médico , la biotecnología , la biología forense , la virología y la sistemáticabiológica . La rápida velocidad de secuenciación obtenida con la moderna tecnología de secuenciación de ADN ha sido fundamental en la secuenciación de secuencias de ADN completas, o genomas , de numerosos tipos y especies de vida, incluido el genoma humanoy otras secuencias de ADN completas de muchos animales, plantas y microbios. especies.
Las primeras secuencias de ADN se obtuvieron a principios de la década de 1970 por investigadores académicos utilizando métodos laboriosos basados en cromatografía bidimensional . Tras el desarrollo de métodos de secuenciación basados en la fluorescencia con un secuenciador de ADN , [2] la secuenciación de ADN se ha vuelto más fácil y las órdenes de magnitud más rápidas.
Aplicaciones [ editar ]
La secuenciación de ADN se puede usar para determinar la secuencia de genes individuales , regiones genéticas más grandes (es decir, agrupaciones de genes u operones ), cromosomas completos o genomas completos de cualquier organismo. La secuenciación de ADN también es la forma más eficiente de secuenciar ARN o proteínas de manera indirecta (a través de sus marcos de lectura abiertos ). De hecho, la secuenciación del ADN se ha convertido en una tecnología clave en muchas áreas de la biología y otras ciencias, como la medicina, la medicina forense y la antropología .
La biología molecular [ editar ]
La secuenciación se utiliza en biología molecular para estudiar los genomas y las proteínas que codifican. La información obtenida mediante la secuenciación permite a los investigadores identificar cambios en los genes, asociaciones con enfermedades y fenotipos, e identificar posibles dianas farmacológicas.
La biología evolutiva [ editar ]
Dado que el ADN es una macromolécula informativa en términos de transmisión de una generación a otra, la secuenciación del ADN se utiliza en la biología evolutiva para estudiar cómo se relacionan los diferentes organismos y cómo evolucionaron.
Metagenómica [ editar ]
El campo de la metagenómica implica la identificación de organismos presentes en un cuerpo de agua, aguas residuales , suciedad, residuos filtrados del aire o muestras de hisopos de organismos. Saber qué organismos están presentes en un entorno particular es fundamental para la investigación en ecología , epidemiología , microbiología y otros campos. La secuenciación permite a los investigadores determinar qué tipos de microbios pueden estar presentes en un microbioma , por ejemplo.
Medicina [ editar ]
Los técnicos médicos pueden secuenciar genes (o, en teoría, genomas completos) de pacientes para determinar si existe riesgo de enfermedades genéticas. Esta es una forma de prueba genética , aunque algunas pruebas genéticas pueden no implicar la secuenciación del ADN.
Forense [ editar ]
La secuenciación de ADN se puede usar junto con los métodos de perfilado de ADN para la identificación forense [4] y las pruebas de paternidad . Las pruebas de ADN han evolucionado enormemente en las últimas décadas para, en última instancia, vincular una impresión de ADN con lo que se está investigando. Los patrones de ADN en huellas dactilares, saliva, folículos pilosos, etc. separan de manera única cada organismo vivo de otro. La prueba de ADN es una técnica que puede detectar genomas específicos en una cadena de ADN para producir un patrón único e individualizado. Cada organismo vivo jamás creado tiene un patrón de ADN único, que puede determinarse a través de pruebas de ADN. Es extremadamente raro que dos personas tengan exactamente el mismo patrón de ADN, por lo tanto, las pruebas de ADN son altamente exitosas.
Las cuatro bases canónicas [ editar ]
La estructura canónica del ADN tiene cuatro bases: timina (T), adenina (A), citosina (C) y guanina (G). La secuenciación del ADN es la determinación del orden físico de estas bases en una molécula de ADN. Sin embargo, hay muchas otras bases que pueden estar presentes en una molécula. En algunos virus (específicamente, bacteriófagos ), la citosina puede ser reemplazada por hidroxi metil o hidroxi metil glucosa citosina. [5] En el ADN de mamíferos, se pueden encontrar bases variantes con grupos metilo o fosfosulfato. [6] [7]Según la técnica de secuenciación, una modificación particular, por ejemplo, el 5mC ( 5 metil citosina) comunes en humanos, pueden o no ser detectados. [8]
Historia [ editar ]
Descubrimiento de la estructura del ADN y la función [ editar ]
El ácido desoxirribonucleico ( ADN ) fue descubierto y aislado por primera vez por Friedrich Miescher en 1869, pero se mantuvo poco estudiado durante muchas décadas porque se pensaba que las proteínas, en lugar del ADN, mantenían el plan genético a la vida. Esta situación cambió después de 1944 como resultado de algunos experimentos realizados por Oswald Avery , Colin MacLeod y Maclyn McCarty que demostraron que el ADN purificado podría transformar una cepa de bacterias en otra. Esta fue la primera vez que se mostró que el ADN era capaz de transformar las propiedades de las células.
En 1953, James Watson y Francis Crick presentaron su modelo de ADN de doble hélice , basado en estructuras de rayos X cristalizadas estudiadas por Rosalind Franklin.- Y sin darle crédito. Según el modelo, el ADN está compuesto por dos cadenas de nucleótidos enrolladas entre sí, unidas entre sí por enlaces de hidrógeno y que se ejecutan en direcciones opuestas. Cada hebra está compuesta por cuatro nucleótidos complementarios, adenina (A), citosina (C), guanina (G) y timina (T), con una A en una hebra siempre emparejada con T en la otra, y C siempre emparejada con G. Propusieron que una estructura de este tipo permitiera que cada hebra se usara para reconstruir la otra, una idea central para la transmisión de información hereditaria entre generaciones. [9]
La base para la secuenciación de proteínas se estableció primero por el trabajo de Frederick Sanger, quien en 1955 había completado la secuencia de todos los aminoácidos en la insulina., una pequeña proteína secretada por el páncreas. Esto proporcionó la primera evidencia concluyente de que las proteínas eran entidades químicas con un patrón molecular específico en lugar de una mezcla aleatoria de material suspendido en un fluido. El éxito de Sanger en la secuenciación de la insulina en los electrógrafos de rayos X muy electrificados, entre ellos Watson y Crick, que ahora intentaban comprender cómo el ADN dirigía la formación de proteínas dentro de una célula. Poco después de asistir a una serie de conferencias impartidas por Frederick Sanger en octubre de 1954, Crick comenzó a desarrollar una teoría que sostenía que la disposición de los nucleótidos en el ADN determinaba la secuencia de aminoácidos en las proteínas, lo que a su vez ayudaba a determinar la función de una proteína. Publicó esta teoría en 1958. [10]
RNA secuenciación [ editar ]
La secuenciación de ARN fue una de las formas más tempranas de secuenciación de nucleótidos. El principal hito de la secuenciación de ARN es la secuencia del primer gen completo y el genoma completo de Bacteriophage MS2 , identificado y publicado por Walter Fiers y sus colaboradores en la Universidad de Gante ( Gante , Bélgica ), en 1972 [11] y 1976. [12] Los métodos tradicionales de secuenciación de ARN requieren la creación de una molécula de ADNc que debe ser secuenciada. [13]
Métodos de secuenciación de ADN Early [ editar ]
El primer método para determinar las secuencias de ADN involucró una estrategia de extensión del cebador específica de la ubicación establecida por Ray Wu en la Universidad de Cornell en 1970. [14] La catálisis por ADN polimerasa y el marcado de nucleótidos específicos, ambos de los cuales figuran prominentemente en los esquemas de secuenciación actuales, se utilizaron para secuenciar Los extremos cohesivos del ADN del fago lambda. [15] [16] [17] Entre 1970 y 1973, Wu, R Padmanabhan y sus colegas demostraron que este método puede emplearse para determinar cualquier secuencia de ADN utilizando cebadores sintéticos específicos de la ubicación. [18] [19] [20] Frederick Sangerluego adoptó esta estrategia de extensión de cebadores para desarrollar métodos de secuenciación de ADN más rápidos en el MRC Center , Cambridge , Reino Unido y publicó un método para "secuenciación de ADN con inhibidores de terminación de cadena" en 1977. [21] Walter Gilbert y Allan Maxam en Harvard también desarrolló métodos de secuenciación, incluido uno para "secuenciación de ADN por degradación química". [22] [23] En 1973, Gilbert y Maxam informaron la secuencia de 24 pares de bases utilizando un método conocido como análisis de punto errante. [24] Los avances en la secuenciación fueron ayudados por el desarrollo concurrente de ADN recombinante Tecnología, que permite aislar muestras de ADN de fuentes distintas a los virus.
Secuenciación de genomas completos [ editar ]
El primer genoma completo de ADN que se secuenció fue el del bacteriófago 1977X174 en 1977. [25] Los científicos del Consejo de Investigación Médica descifraron la secuencia completa de ADN del virus de Epstein-Barr en 1984, encontrando que contenía 172,282 nucleótidos. La finalización de la secuencia marcó un punto de inflexión significativo en la secuenciación del ADN porque se logró sin un conocimiento previo del perfil genético del virus. [26]
Un método no radiactivo para transferir las moléculas de ADN de las mezclas de reacción de secuenciación a una matriz inmovilizadora durante la electroforesis fue desarrollado por Pohl y colaboradores a principios de los años ochenta. [27] [28] Seguido de la comercialización del secuenciador de ADN "Direct-Blotting-Electrophoresis-System GATC 1500" por GATC Biotech , que se utilizó intensivamente en el marco del programa de secuenciación del genoma de la UE, la secuencia completa de ADN del Levadura Saccharomyces cerevisiae cromosoma II. [29] El laboratorio de Leroy E. Hood en el Instituto de Tecnología de California anunció la primera máquina semiautomática de secuenciación de ADN en 1986. [30] Esto fue seguido porApplied Biosystems comercializó la primera máquina de secuenciación totalmente automatizada, la ABI 370, en 1987 y por Dupont's Genesis 2000 [31], que utilizó una novedosa técnica de etiquetado fluorescente que permite identificar los cuatro dideoxinucleótidos en un solo carril. Para 1990, los Institutos Nacionales de la Salud (NIH, por sus siglas en inglés) de los EE. UU . Habían iniciado ensayos de secuenciación a gran escala en Mycoplasma capricolum , Escherichia coli , Caenorhabditis elegans y Saccharomyces cerevisiae a un costo de US $ 0.75 por base. Mientras tanto, la secuenciación de secuencias de ADNc humano denominadas etiquetas de secuencia expresada comenzó en Craig VenterEl laboratorio, un intento de capturar la fracción de codificación del genoma humano . [32] En 1995, Venter, Hamilton Smith y sus colegas del Instituto para la Investigación Genómica (TIGR) publicaron el primer genoma completo de un organismo de vida libre, la bacteria Haemophilus influenzae . El cromosoma circular contiene 1,830,137 bases y su publicación en la revista Science [33] marcó el primer uso publicado de la secuenciación de escopeta de genoma completo, eliminando la necesidad de esfuerzos iniciales de mapeo.
Para el año 2001, los métodos de secuenciación de escopeta se habían utilizado para producir un borrador de secuencia del genoma humano. [34] [35]
Métodos de secuenciación de alto rendimiento (HTS) [ editar ]
Varios métodos nuevos para la secuenciación de ADN se desarrollaron a mediados y finales de los años 90 y se implementaron en secuenciadores de ADN comerciales.para el año 2000. Juntos, se denominaron métodos de secuenciación de "segunda generación" o "segunda generación" (NGS), para distinguirlos de los métodos anteriores mencionados anteriormente, como la secuenciación de Sanger. A diferencia de la primera generación de secuenciación, la tecnología NGS se caracteriza por ser altamente escalable, lo que permite secuenciar todo el genoma a la vez. Por lo general, esto se logra al fragmentar el genoma en fragmentos pequeños, muestrear aleatoriamente un fragmento y secuenciarlo utilizando una de una variedad de tecnologías, como las que se describen a continuación. Un genoma completo es posible porque se secuencian múltiples fragmentos a la vez (dándole el nombre de secuenciación "masivamente paralela") en un proceso automatizado.
La tecnología NGS ha permitido que los investigadores busquen información sobre la salud, que los antropólogos investiguen los orígenes humanos y está catalizando la " medicina personalizada "."movimiento. Sin embargo, también ha abierto la puerta a más margen de error. Hay muchas herramientas de software para llevar a cabo el análisis computacional de los datos NGS, cada uno con su propio algoritmo. Incluso los parámetros dentro de un paquete de software pueden cambiar el resultado de Además, las grandes cantidades de datos producidos por la secuenciación de ADN también han requerido el desarrollo de nuevos métodos y programas para el análisis de secuencias. Se han intentado varios esfuerzos para desarrollar estándares en el campo de NGS para abordar estos desafíos, la mayoría de los cuales han sido esfuerzos a pequeña escala que surgen de laboratorios individuales. Más recientemente, un esfuerzo grande, organizado y financiado por la FDA ha culminado en el estándar BioCompute .
El 26 de octubre de 1990, Roger Tsien , Pepi Ross, Margaret Fahnestock y Allan J Johnston presentaron una patente que describe la secuenciación paso a paso ("base por base") con bloqueadores 3 'removibles en arreglos de ADN (transferencias y moléculas de ADN individuales). [36] En 1996, Pål Nyrén y su estudiante Mostafa Ronaghi en el Instituto Real de Tecnología de Estocolmo publicaron su método de pirosecuenciación . [37]
El 1 de abril de 1997, Pascal Mayer y Laurent Farinelli presentaron patentes a la Organización Mundial de la Propiedad Intelectual que describen la secuenciación de colonias de ADN. [38] La preparación de la muestra de ADN y los métodos aleatorios de preparación de PCR en superficie descritos en esta patente, junto con el método de secuenciación "base por base" de Roger Tsien et al., Ahora se implementan en los secuenciadores del genoma Hi-Seq de Illumina . .
En 1998, Phil Green y Brent Ewing de la Universidad de Washington describieron su puntaje de calidad de phredpara el análisis de datos del secuenciador, [39] una técnica de análisis histórica que obtuvo una adopción generalizada y que sigue siendo la métrica más común para evaluar la precisión de una secuenciación plataforma. [40]
Lynx Therapeutics publicó y comercializó la secuenciación masiva de firmas paralelas (MPSS), en el año 2000. Este método incorporó una tecnología de secuenciación basada en perlas, mediada por adaptadores / ligaduras, paralelizada y sirvió como el primer método de secuenciación de "próxima generación" disponible comercialmente, aunque Los secuenciadores de ADN fueron vendidos a laboratorios independientes. [41]
Métodos básicos [ editar ]
Maxam-Gilbert secuenciación [ editar ]
Allan Maxam y Walter Gilbert publicaron un método de secuenciación de ADN en 1977 basado en la modificación química del ADN y la subsiguiente separación en bases específicas. [22] También conocido como secuenciación química, este método permitió el uso de muestras purificadas de ADN de doble cadena sin clonación adicional. El uso de este método de etiquetado radioactivo y su complejidad técnica desalentaron el uso extensivo después de que se hubieran hecho refinamientos en los métodos de Sanger.
La secuenciación de Maxam-Gilbert requiere un marcado radioactivo en un extremo 5 'del ADN y la purificación del fragmento de ADN que se va a secuenciar. El tratamiento químico genera entonces rupturas en una pequeña proporción de una o dos de las cuatro bases de nucleótidos en cada una de las cuatro reacciones (G, A + G, C, C + T). La concentración de los productos químicos modificadores se controla para introducir en promedio una modificación por molécula de ADN. Por lo tanto, se genera una serie de fragmentos marcados, desde el extremo radiomarcado hasta el primer sitio "cortado" en cada molécula. Los fragmentos en las cuatro reacciones se someten a electroforesis lado a lado en geles de acrilamida desnaturalizantes para la separación por tamaños. Para visualizar los fragmentos, el gel se expone a una película de rayos X para autorradiografía, produciendo una serie de bandas oscuras que corresponden cada una a un fragmento de ADN radiomarcado, del cual se puede inferir la secuencia.[22]
Métodos de terminación de cadena [ editar ]
El método de terminación de cadena desarrollado por Frederick Sanger y sus colaboradores en 1977 pronto se convirtió en el método de elección, debido a su relativa facilidad y fiabilidad. [21] [42] Cuando se inventó, el método de terminación de cadena usaba menos químicos tóxicos y cantidades de radioactividad más bajas que el método de Maxam y Gilbert. Debido a su facilidad comparativa, el método Sanger pronto se automatizó y fue el método utilizado en la primera generación de secuenciadores de ADN .
La secuenciación de Sanger es el método que prevaleció desde la década de 1980 hasta mediados de la década de 2000. Durante ese período, se hicieron grandes avances en la técnica, como el etiquetado fluorescente, la electroforesis capilar y la automatización general. Estos desarrollos permitieron una secuenciación mucho más eficiente, lo que lleva a costos más bajos. El método Sanger, en forma de producción en masa, es la tecnología que produjo el primer genoma humano en 2001, marcando el comienzo de la era de la genómica . Sin embargo, más adelante en la década, los enfoques radicalmente diferentes llegaron al mercado, lo que hizo que el costo por genoma bajara de $ 100 millones en 2001 a $ 10,000 en 2011. [43]
Métodos avanzados y de novo secuenciación [ editar ]
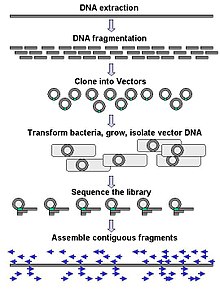
La secuenciación a gran escala a menudo apunta a la secuenciación de fragmentos de ADN muy largos, como cromosomas completos , aunque la secuenciación a gran escala también se puede usar para generar un gran número de secuencias cortas, como las que se encuentran en la visualización de fagos . Para objetivos más largos, como los cromosomas, los enfoques comunes consisten en cortar (con enzimas de restricción ) o cortar (con fuerzas mecánicas) grandes fragmentos de ADN en fragmentos de ADN más cortos. El ADN fragmentado puede luego clonarse en un vector de ADN y amplificarse en un huésped bacteriano como Escherichia coli . Los fragmentos cortos de ADN purificados a partir de colonias bacterianas individuales se secuencian individualmente y se ensamblan electrónicamenteen una secuencia larga y contigua. Los estudios han demostrado que agregar un paso de selección de tamaño para recolectar fragmentos de ADN de tamaño uniforme puede mejorar la eficacia de la secuenciación y la precisión del ensamblaje del genoma. En estos estudios, el dimensionamiento automatizado ha demostrado ser más reproducible y preciso que el dimensionamiento manual en gel. [44] [45] [46]
El término " secuenciación de novo " se refiere específicamente a los métodos utilizados para determinar la secuencia de ADN sin una secuencia conocida previamente. De novo se traduce del latín como "desde el principio". Los huecos en la secuencia ensamblada se pueden rellenar con la imprimación caminando . Las diferentes estrategias tienen diferentes ventajas y desventajas en velocidad y precisión; Los métodos de escopeta a menudo se usan para secuenciar genomas grandes, pero su ensamblaje es complejo y difícil, particularmente con repeticiones de secuencias que a menudo causan huecos en el ensamblaje del genoma.
La mayoría de los enfoques de secuenciación utilizan una etapa de clonación in vitro para amplificar moléculas de ADN individuales, porque sus métodos de detección molecular no son lo suficientemente sensibles para la secuenciación de una sola molécula. Emulsion PCR [47] aísla moléculas de ADN individuales junto con perlas recubiertas con cebador en gotitas acuosas dentro de una fase oleosa. Una reacción en cadena de la polimerasa(PCR) luego cubre cada perla con copias clonales de la molécula de ADN seguida de inmovilización para su posterior secuenciación. La PCR de emulsión se utiliza en los métodos desarrollados por Marguilis et al. (comercializado por 454 Life Sciences ), Shendure y Porreca et al. (también conocida como " secuenciación Polony ") y secuenciación SOLiD , (desarrollada por Agencourt, posteriormente Applied Biosystems , ahora Life Technologies ). [48] [49] [50] Emulsion PCR también se usa en las plataformas GemCode y Chromium desarrolladas por 10x Genomics. [51]
Secuenciación Shotgun [ editar ]
La secuenciación de Shotgun es un método de secuenciación diseñado para el análisis de secuencias de ADN de más de 1000 pares de bases, hasta e incluyendo cromosomas completos. Este método requiere que el ADN objetivo se rompa en fragmentos aleatorios. Después de la secuenciación de fragmentos individuales, las secuencias se pueden volver a ensamblar en función de sus regiones superpuestas. [52]
Puente PCR [ editar ]
Otro método para la amplificación clónica in vitro es la PCR de puente, en la que los fragmentos se amplifican sobre cebadores unidos a una superficie sólida [38] [53] [54] y forman " colonias de ADN " o "agrupaciones de ADN". Este método se utiliza en los secuenciadores Illumina Genome Analyzer . Los métodos de una sola molécula, como el desarrollado por el laboratorio de Stephen Quake (comercializado posteriormente por Helicos ) son una excepción: utilizan fluoróforos brillantes y excitación con láser para detectar eventos de adición de bases de moléculas de ADN individuales fijadas a una superficie, eliminando la necesidad de amplificación molecular.



No hay comentarios:
Publicar un comentario