El ADNccNA ( ADN circular covalentemente cerrado ) es una estructura especial de ADN que surge durante la propagación de algunos virus en el núcleo celular y puede permanecer allí permanentemente. Es un ADN de doble cadena que se origina en una forma lineal que se liga por medio de la ADN ligasa a un anillo cerrado covalentemente . En la mayoría de los casos, la transcripción del ADN viral puede ocurrir únicamente a partir de la forma circular. El ADNccNA de los virus también se conoce como ADN episomal u ocasionalmente como un minicromosoma .
La existencia de un ADNccNA durante la propagación no diferencia el grupo taxonómico de los retrovirus "reales" ( Orthoretrovirinae ) del pararetrovirus .
El ADNccNA se describió por primera vez en bacteriófagos , pero también se encontró en algunos cultivos celulares donde se detectó una infección de virus de ADN ( Polyomaviridae ). [1] [2] El ADNccNA es típico de Caulimoviridae y Hepadnaviridae , incluido el virus de la hepatitis B (VHB). El ADNccNA en HBV se forma por conversión de ADN circular relajado asociado a la cápside (rcDNA). [3] Después de las infecciones de hepatitis B, el cccDNA puede permanecer después del tratamiento clínico en las células del hígado y rara vez puede reactivarse. La cantidad relativa de cccDNA presente es un indicador para el tratamiento del VHB.
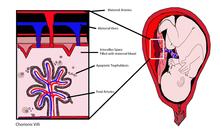
El ADN fetal libre de células ( ADNcf ) es un ADN fetal que circula libremente en la sangre materna. La sangre materna se muestrea por venopunción . El análisis de cffDNA es un método de diagnóstico prenatal no invasivo que se solicita con frecuencia a mujeres embarazadas en edad materna avanzada . Dos horas después del parto, el cffDNA ya no es detectable en la sangre materna.
Fondo [ editar ]
cffDNA se origina a partir de trofoblastos placentarios . [1] [2] El ADN fetal se fragmenta cuando las micropartículas placentarias se desprenden de la circulación sanguínea materna . [3]
Los fragmentos de ADNcff tienen aproximadamente 200 pares de bases (pb) de longitud. Son significativamente más pequeños que los fragmentos de ADN maternos. [4] La diferencia de tamaño permite distinguir el ADNcff de los fragmentos de ADN maternos. [5] [6]
Aproximadamente del 11 al 13,4 por ciento del ADN libre de células en la sangre materna es de origen fetal. La cantidad varía ampliamente de una mujer embarazada a otra. [7] cffDNA está presente después de cinco a siete semanas de gestación. La cantidad de cffDNA aumenta a medida que avanza el embarazo. [8] La cantidad de cffDNA en la sangre materna disminuye rápidamente después del parto. Dos horas después del parto, el cffDNA ya no es detectable en la sangre materna. [9]
El análisis de cffDNA puede proporcionar un diagnóstico más temprano de afecciones fetales que las técnicas actuales. Como el cffDNA se encuentra en la sangre materna, el muestreo no conlleva un riesgo asociado de aborto espontáneo . [10] [11] [12] [13] [14] El análisis del cffDNA tiene los mismos problemas éticos y prácticos que otras técnicas como la amniocentesis y el muestreo de vellosidades coriónicas . [15]
Algunas desventajas del muestreo de cffDNA incluyen una baja concentración de cffDNA en la sangre materna; variación en la cantidad de cffDNA entre individuos; una alta concentración de ADN materno libre de células en comparación con el ADNcff en la sangre materna. [dieciséis]
La nueva evidencia muestra que la tasa de fracaso de la prueba de cffDNA es mayor, la fracción fetal (proporción de ADN fetal en comparación con el materno en la muestra de sangre materna) es menor y el VPP para las trisomías 18, 13 y SCA disminuyen en los embarazos de FIV en comparación con los concebidos espontáneamente. [17]
Métodos de laboratorio [ editar ]
Se han desarrollado varios métodos de laboratorio para la detección del ADN fetal sin células para detectar defectos genéticos. Los principales son (1) Secuenciación masiva de escopeta paralela (MPSS), (2) Secuenciación masiva dirigida dirigida (t-MPS) y (3) Enfoque basado en polimorfismo de nucleótido único (SNP). [18] [19] [20]
Se toma una muestra de sangre periférica materna por venesección a aproximadamente diez semanas de gestación. [21]
Separación de cffDNA [ editar ]
El plasma sanguíneo se separa de la muestra de sangre materna mediante una centrífuga de laboratorio . Luego se aisla y se purifica el ADNcff. [22] Un protocolo estandarizado para hacer esto fue escrito a través de una evaluación de la literatura científica . El mayor rendimiento en la extracción de ADNcff se obtuvo con el "Kit de Virus QIAamp DSP". [23]
La adición de formaldehído a las muestras de sangre materna aumenta el rendimiento de cffDNA. El formaldehído estabiliza las células intactas y, por lo tanto, inhibe la liberación adicional de ADN materno. Con la adición de formaldehído, el porcentaje de cffDNA recuperado de una muestra de sangre materna varía entre 0.32 por ciento y 40 por ciento, con una media de 7.7 por ciento. [24] Sin la adición de formaldehído, el porcentaje medio de ADNcc recuperado se ha medido en 20.2 por ciento. Sin embargo, otras cifras varían entre el 5 y el 96 por ciento. [25] [26]
La recuperación del ADNcff puede estar relacionada con la longitud de los fragmentos de ADN. Otra forma de aumentar el ADN fetal se basa en la longitud física de los fragmentos de ADN. Los fragmentos más pequeños pueden representar hasta el setenta por ciento del ADN total libre de células en la muestra de sangre materna.
Análisis de cffDNA [ editar ]
En la PCR en tiempo real, se utilizan sondas fluorescentes para controlar la acumulación de amplicones . La señal fluorescente del reportero es proporcional al número de amplicones generados. El protocolo de PCR en tiempo real más apropiado está diseñado de acuerdo con la mutación o genotipo particular que se detectará. Las mutaciones puntuales se analizan con PCR cualitativa en tiempo real con el uso de sondas específicas de alelos. Las inserciones y deleciones se analizan mediante mediciones de dosis mediante PCR cuantitativa en tiempo real.
El ADNcff puede detectarse encontrando secuencias de ADN heredadas paternalmente mediante la reacción en cadena de la polimerasa (PCR). [27] [28]
PCR cuantitativa en tiempo real [ editar ]
El gen de la región Y determinante del sexo (SRY) y la repetición corta en tándem del cromosoma Y "DYS14" en el cffDNA de 511 embarazos se analizaron mediante PCR cuantitativa en tiempo real (RT-qPCR). En 401 de 403 embarazos en los que se extrajo sangre materna a las siete semanas de gestación o más, se encontraron ambos segmentos de ADN. [29]
Nested PCR [ editar ]
El uso de la reacción en cadena de la polimerasa anidada (PCR anidada) se evaluó para determinar el sexo mediante la detección de una señal específica del cromosoma Y en el ADNcc del plasma materno. PCR anidada detectó 53 de 55 fetos masculinos. El cffDNA del plasma de 3 de 25 mujeres con fetos femeninos contenía la señal específica del cromosoma Y. La sensibilidad de la PCR anidada en este experimento fue del 96 por ciento. La especificidad fue del 88 por ciento. [30]
PCR digital [ editar ]
Los dispositivos microfluídicos permiten la cuantificación de segmentos de cffDNA en plasma materno con una precisión superior a la de la PCR en tiempo real. Las mutaciones puntuales , la pérdida de heterocigosidad y la aneuploidía se pueden detectar en un solo paso de PCR. [31] [32] [33] La PCR digital puede diferenciar entre el plasma sanguíneo materno y el ADN fetal de forma múltiple . [31]
Secuenciación Shotgun [ editar ]
La secuenciación de escopeta de alto rendimiento con herramientas como Solexa o Illumina, produce aproximadamente 5 millones de etiquetas de secuencia por muestra de suero materno. Los embarazos aneuploides, como la trisomía, se identificaron al realizar la prueba en la decimocuarta semana de gestación. La totalidad del mapeo del genoma fetal mediante análisis de haplotipos parentales se completó utilizando la secuenciación del ADNcff del suero materno. [13] Las hembras embarazadas se estudiaron utilizando una secuenciación de ADN en plasma materno paralela masivamente paralela y la trisomía se diagnosticó con una puntuación z mayor que 3. [34] La secuenciación dio una sensibilidad del 100 por ciento, una especificidad del 97.9 por ciento, un valor predictivo positivode 96.6 por ciento y un valor predictivo negativo de 100 por ciento.
La espectrometría de masas [ editar ]
Desorción / ionización con láser asistida por matriz : la espectrometría de masas de tiempo de vuelo (MALDI-TOF MS) combinada con la extensión de una sola base después de la PCR permite la detección de cffDNA con una especificidad de base única y una única sensibilidad de molécula de ADN. [35] El ADN se amplifica por PCR. Luego, la amplificación lineal con la reacción de extensión de la base (con un tercer cebador) se diseña para acoplarse a la región corriente arriba del sitio de mutación . Se agregan una o dos bases al cebador de extensión para producir dos productos de extensión a partir de ADN de tipo salvaje y ADN mutante. La especificidad de base única proporciona ventajas sobre las técnicas basadas en hibridación que utilizan TaqManSondas de hidrólisis. Al evaluar la técnica, no se encontraron falsos positivos ni negativos al buscar cffDNA para determinar el sexo fetal en dieciséis muestras de plasma materno. [35] El sexo de noventa de noventa y un fetos masculinos se detectó correctamente utilizando MALDI-TOF mas espectrometría. La técnica tuvo una precisión, sensibilidad y especificidad de más del 99 por ciento. [36]
Las modificaciones epigenéticas [ editar ]
Se pueden aprovechar las diferencias en la activación de genes entre el ADN materno y fetal. Se pueden usar modificaciones epigenéticas ( modificaciones hereditarias que cambian la función del gen sin cambiar la secuencia de ADN) para detectar el ADNcff. [37] [38] El promotor hipermetilado de RASSF1 A es un marcador fetal universal utilizado para confirmar la presencia de ADNcff. [39] Se describió una técnica donde el cffDNA se extrajo de plasma materno y luego se digirió con enzimas de restricción insensibles y sensibles a la metilación . Luego, se realizó un análisis de PCR en tiempo real de RASSF1A, SRY y DYS14. [39] El procedimiento detectó 79 de 90 (88 por ciento) muestras de sangre materna donde estaba presente RASSF1A hipermetilado.
ARNm [ editar ]
Los transcritos de ARNm de genes expresados en la placenta son detectables en el plasma materno. [40] En este procedimiento, el plasma se centrifuga para que aparezca una capa acuosa. Esta capa se transfiere y de ella se extrae el ARN . RT-PCR se utiliza para detectar una expresión seleccionada de ARN. Por ejemplo, el lactógeno placentario humano (hPL) y el ARNm de beta-hCG son estables en el plasma materno y pueden detectarse. (Ng et al. 2002). Esto puede ayudar a confirmar la presencia de cffDNA en el plasma materno. [dieciséis]
Aplicaciones [ editar ]
Prenatal del sexo discernimiento [ editar ]
El análisis de cffDNA de una muestra de plasma materno permite el discernimiento sexual prenatal . Las aplicaciones del discernimiento sexual prenatal incluyen:
- Pruebas de enfermedad : si el sexo del feto es masculino o femenino permite determinar el riesgo de un trastorno genético recesivo ligado a X en particular en un embarazo particular, especialmente cuando la madre es un portador genético del trastorno. [41]
- Preparación , para cualquier aspecto dependiente del sexo de la crianza.
- Selección de sexo , que después del diagnóstico genético previo a la implantación se puede realizar seleccionando solo los embriones del sexo preferido o, después de los métodos de postimplante, realizandoun aborto selectivo según el resultado de la prueba y la preferencia personal.
En comparación con la ecografía obstétrica que no es confiable para la determinación del sexo en el primer trimestre y la amniocentesis que conlleva un pequeño riesgo de aborto espontáneo , la toma de muestras del plasma materno para el análisis del ADNcff no tiene ningún riesgo. [42] Los principales objetivos en el análisis de cffDNA son el gen responsable de la proteína de la región Y (SRY) que determina el sexo en el cromosoma Y y la secuencia DYS14. [43] [44]
Hiperplasia suprarrenal congénita [ editar ]
En la hiperplasia suprarrenal congénita , la corteza suprarrenal carece de la síntesis apropiada de corticosteroides, lo que lleva a un exceso de andrógenos suprarrenales y afecta a los fetos femeninos. [45] Hay una masculinización externa de los genitales en los fetos femeninos. [46] A las madres de fetos en riesgo se les administra dexametasona a las 6 semanas de gestación para suprimir la liberación de andrógenos en la glándula pituitaria . [47]
Si el análisis de cffDNA obtenido de una muestra de plasma materno carece de marcadores genéticos que se encuentran solo en el cromosoma Y, es sugestivo de un feto femenino. Sin embargo, también podría indicar una falla del análisis en sí (un resultado falso negativo). Paternal polimorfismos genéticos y marcadores sexo independientes pueden ser utilizados para detectar cffDNA. Un alto grado de heterocigosidad de estos marcadores debe estar presente para esta aplicación. [48]
Las pruebas de paternidad [ editar ]
La prueba de paternidad de ADN prenatal está disponible comercialmente. La prueba se puede realizar a las nueve semanas de gestación. [ cita requerida ]
Trastornos de genes individuales [ editar ]
Los trastornos de un solo gen autosómicos dominantes y recesivos que se han diagnosticado prenatalmente mediante el análisis de ADN heredado por vía paterna incluyen fibrosis quística , talasemia beta , anemia de células falciformes , atrofia muscular espinal y distrofia miotónica . [27] [43] El diagnóstico prenatal de trastornos de un solo gen que se deben a una mutación autosómica recesiva, una mutación autosómica dominante hereditaria de la madre o mutaciones de secuencias grandes que incluyen duplicación, expansión o inserción de secuencias de ADN es más difícil. [49]
En cffDNA, los fragmentos de 200 a 300 pb de longitud involucrados en trastornos de un solo gen son más difíciles de detectar.
Por ejemplo, la condición autosómica dominante, la acondroplasia es causada por la mutación puntual del gen FGFR3. [50] En dos embarazos con un feto con acondroplasia se encontró una mutación G1138A hereditaria paterna de cffDNA de una muestra de plasma materno en una y una mutación de novo G1138A de la otra. [50]
En estudios de la genética de la corea de Huntington utilizando qRT-PCR de cffDNA de muestras de plasma maternas, se han detectado repeticiones de CAG a niveles normales (17, 20 y 24). [51]
cffDNA también se puede utilizar para diagnosticar trastornos de un solo gen . [15] Los desarrollos en procesos de laboratorio que utilizan ADNcc pueden permitir el diagnóstico prenatal de aneuploidías como la trisomía 21(síndrome de Down) en el feto. [52] [32]
Enfermedad hemolítica del feto y el recién nacido [ editar ]
La incompatibilidad de los antígenos RhD maternos y fetales es la causa principal de la enfermedad hemolítica del recién nacido . [53] Aproximadamente el 15 por ciento de las mujeres caucásicas , el 3 a 5 por ciento de las mujeres negras de África y menos del 3 por ciento de las mujeres asiáticas son RhD negativo. [54]
El diagnóstico prenatal preciso es importante porque la enfermedad puede ser fatal para el recién nacido y porque el tratamiento que incluye inmunoglobulina intramuscular (Anti-D) o inmunoglobulina intravenosa puede administrarse a madres en riesgo. [55]
PCR para detectar RHD (gen), gen, exones 5 y 7 del ADNcn obtenido del plasma materno entre las 9 y las 13 semanas de gestación, proporciona un alto grado de especificidad, sensibilidad y precisión diagnóstica (> 90 por ciento) en comparación con la determinación de RhD en suero de sangre del recién nacido. . [53] Se obtuvieron resultados similares dirigidos a los exones 7 y 10. [56] La PCR digital en gotitas en la determinación de RhD fetal fue comparable a una técnica de PCR en tiempo real de rutina. [57]
La determinación de rutina del estado de RhD fetal a partir de cffDNA en suero materno permite el manejo temprano de embarazos en riesgo mientras disminuye el uso innecesario de Anti-D en más del 25 por ciento. [58]
Aneuploidía [ editar ]
- Cromosomas sexuales
Análisis de cffDNA suero materno por secuenciación de alto rendimiento puede detectar cromosomas sexuales fetales comunes aneuploidías tales como el síndrome de Turner , el síndrome de Klinefelter y el triple X síndromepero el procedimiento de valor predictivo positivo es bajo. [59]
- Trisomia 21
La trisomía fetal del cromosoma 21 es la causa del síndrome de Down. Esta trisomía se puede detectar mediante el análisis del ADNcff de la sangre materna mediante la secuenciación masiva de escopetas paralelas (MPSS). [60] Otra técnica es el análisis digital de regiones seleccionadas (DANSR). [60] Sin embargo, tales pruebas muestran grados inconsistentes de sensibilidad y especificidad y, por lo tanto, pueden utilizarse mejor para confirmar una prueba de detección materna positiva, como los marcadores de ultrasonido de la afección. [60] [61]
- Trisomia 13 y 18
Es posible el análisis del ADNcff del plasma materno con MPSS en busca de trisomía 13 o 18 [62]
Los factores que limitan la sensibilidad y la especificidad incluyen los niveles de cffDNA en el plasma materno; Los cromosomas maternos pueden tener mosaicismo . [63]
Un número de moléculas de ácidos nucleicos fetales derivados de cromosomas aneuploides puede ser detectada incluyendo SERPINEB2 mRNA, revestido B, SERPINB5 hypomethylated del cromosoma 18, placenta específicas de 4 (PLAC4), hypermethylated holocarboxilasa sintetasa (HLCS) y ARNm c21orf105 del cromosoma 12. [64 ]Con la trisomía completa, los alelos de ARNm en el plasma materno no tienen la proporción normal de 1: 1, pero en realidad son de 2: 1. Las relaciones alélicas determinadas por marcadores epigenéticos también se pueden usar para detectar las trisomías completas. La secuenciación masiva en paralelo y la PCR digital para la detección de aneuploidía fetal se pueden usar sin restricción para las moléculas de ácido nucleico específicas del feto. (MPSS) se estima que tiene una sensibilidad de entre 96 y 100%, y una especificidadentre el 94 y el 100% para detectar el síndrome de Down. Se puede realizar a las 10 semanas de edad gestacional . [65] Un estudio en los Estados Unidos estimó una tasa de falsos positivos de 0.3% y un valor predictivo positivo de 80% cuando se usa el cffDNA para detectar el síndrome de Down. [66]
Preeclampsia [ editar ]
La preeclampsia es una condición compleja del embarazo que involucra hipertensión y proteinuria, generalmente después de las 20 semanas de gestación. [67] Se asocia con una mala invasión citotrofoblástica del miometrio . El inicio de la condición entre las 20 y 34 semanas de gestación, se considera "temprano". [68] Las muestras de plasma materno en embarazos complicados por preeclampsia tienen niveles significativamente más altos de cffDNA que aquellos en embarazos normales. [69] [70] [71] Esto se aplica a la preeclampsia de inicio temprano. [68]
Perspectivas de futuro [ editar ]
La secuenciación de nueva generación se puede usar para producir una secuencia del genoma completo a partir de ADNcff. Esto plantea cuestiones éticas. [72] Sin embargo, la utilidad del procedimiento puede aumentar a medida que se descubren asociaciones claras entre variantes genéticas específicas y estados de enfermedad.
No hay comentarios:
Publicar un comentario